相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 询价记录
- 文献和实验
- 技术资料
- 提供商:
表观生物
- 服务名称:
acRIP-seq

N4-acetylcytidine (ac4C),N4位乙酰胞嘧啶,是真核原核生物中保守的化学修饰,早期研究认为ac4C主要存在tRNA和18S rRNA上[1]。而近期研究显示,mRNA上也存在大量的ac4C,该修饰在促进蛋白翻译,影响RNA稳定性和可变剪接,调控基因表达发挥重要作用,有望成为表观转录组学的新兴发展方向[2,3]。
表观生物积极创新,在国际率先推出ac4C免疫共沉淀测序服务(acRIP-seq),全面解析ac4C在转录组的分布及变化,帮助科研人员深入理解RNA乙酰化在生命和疾病中的作用,促进疾病的精准诊断与治疗研究。
技术原理
RNA乙酰化免疫共沉淀 (acetylated RNA Immunoprecipitation,acRIP)基于抗体特异性结合乙酰化修饰的碱基的原理,以RNA免疫共沉淀富集甲基化修饰片段为基础,然后通过高通量测序,在全转录组范围内研究发生乙酰化的RNA区域,高效获得结果。
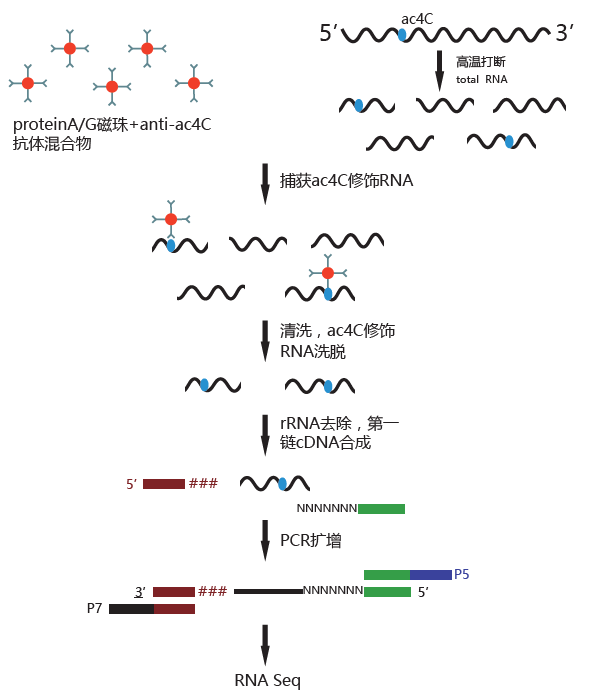
图1. acRIP-seq实验流程图
分析内容
基本分析
1. 测序原始reads 去接头,质量控制(QC)
2. 参考基因组比对(Mapping)
3. 富集区域鉴定(PeakCallling)
4. 富集区域注释(PeakAnno)
5. lncRNA修饰分析、mRNA修饰分析
6. 富集区域metagene图、pie图
7. Motif分析(CXX repeat motif)
8. Peak基因GO 和KEGG 分析
9. 差异Peak分析(差异乙酰化分析)
10. 差异Peak基因GO 和KEGG 分析
11. 单位点修饰预测分析(Single Site,CXXCXXCXXCXX 的预测)
12. 关联分析(与RNA-seq 关联分析)
13. 关联分析四象限图
14. 关联分析热图(仅限有生物学重复)
高级分析
1. RNA-seq 表达差异与ac4C修饰差异关联分析
2. mRNA 可变剪切与ac4C修饰差异关联分析
3. 翻译组Ribo-seq 翻译差异与ac4C修饰差异关联分析
实测数据
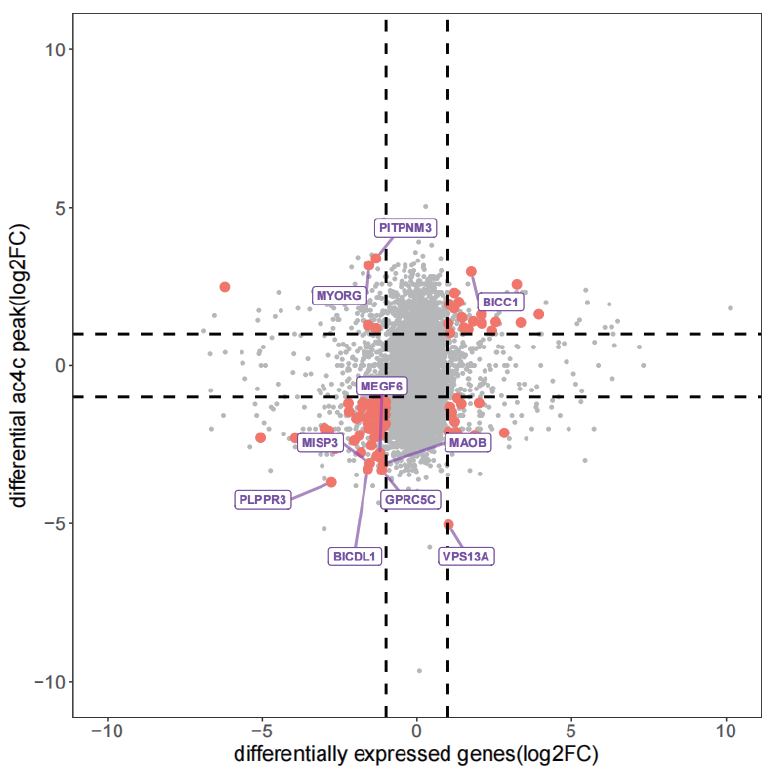 图2.多组学关联分析四象限图(RNA-seq 与acRIP-seq)
图2.多组学关联分析四象限图(RNA-seq 与acRIP-seq)
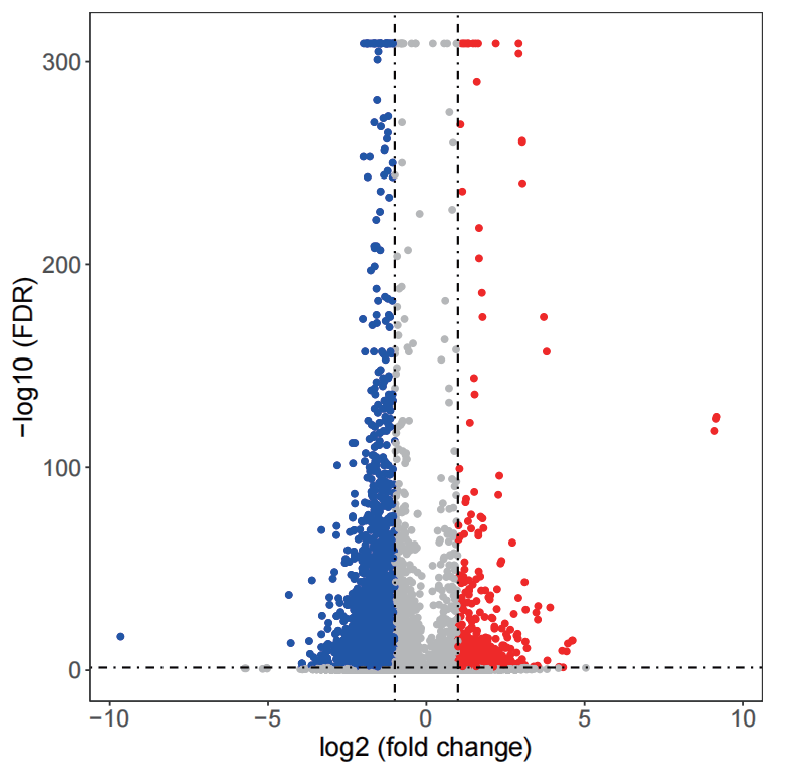
图3. 差异Peaks火山图
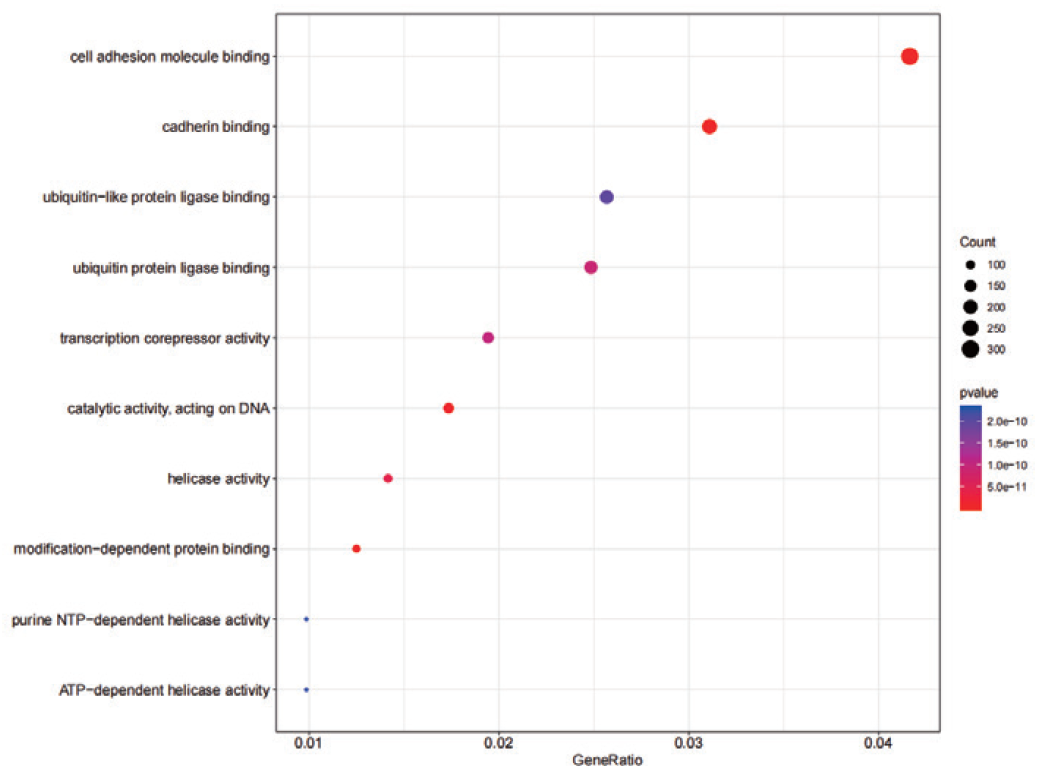
图4. 差异GO分析气泡图
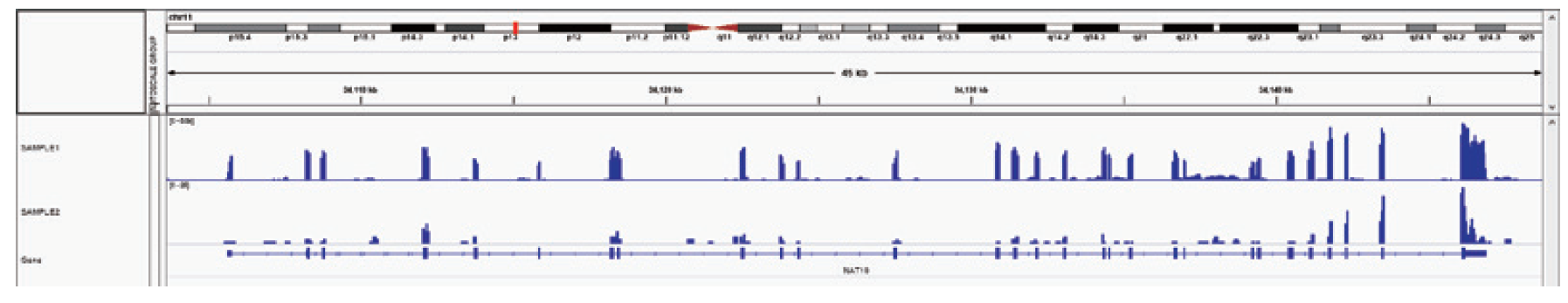
图5. IGV峰图
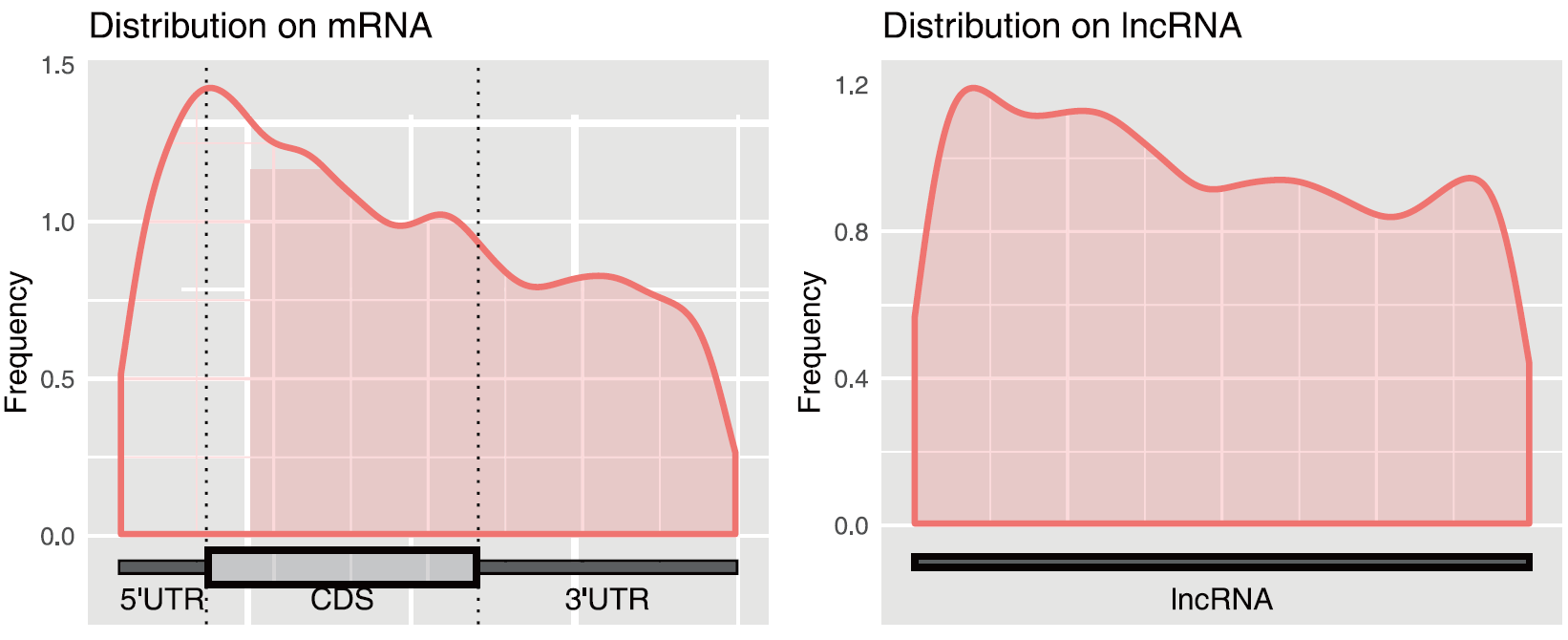
图6. ac4C修饰metagene图
送样要求
样本类型:
1. 总RNA质量>50ug
2. 细胞数量>1.5x107
3. 组织质量>50mg
样本物种:仅限人、大小鼠,其他物种需评估
客户文章
Cancer Res: NAT10 通过增强ac4C相关的DNA修复导致膀胱癌症顺铂化疗耐药[1]
为了阐明NAT10介导化疗耐药的调控机制,研究者通过acRIP- seq发现mRNA 上存在大量的ac4C 修饰,包括终止密码子附近亦存在大量的ac4C修饰位点。接着研究者敲低NAT10 后做了RNA-seq,联合acRIP-seq 的结果发现,和以往报道的5’端ac4C 修饰的促翻译作用不同,NAT10 能够直接调控ac4C 修饰在终止密码子附近的mRNA 的稳定性。随后通过IP实验,研究者鉴定了与NAT10 相互结合的蛋白HNRNPQ 及THRAP3,并揭示了NAT10 通过结合HNRNPQ及THRAP3防止下游靶基因mRNA降解。临床数据分析表明NAT10/HNRNPQ/THRAP3联合表达水平可显著预示膀胱癌患者的不良预后,具有重要的临床意义。最后,结合一系列的细胞实验、动物实验及类器官实验,证实Remodelin能够安全有效地增强顺铂对膀胱癌细胞的杀伤作用。
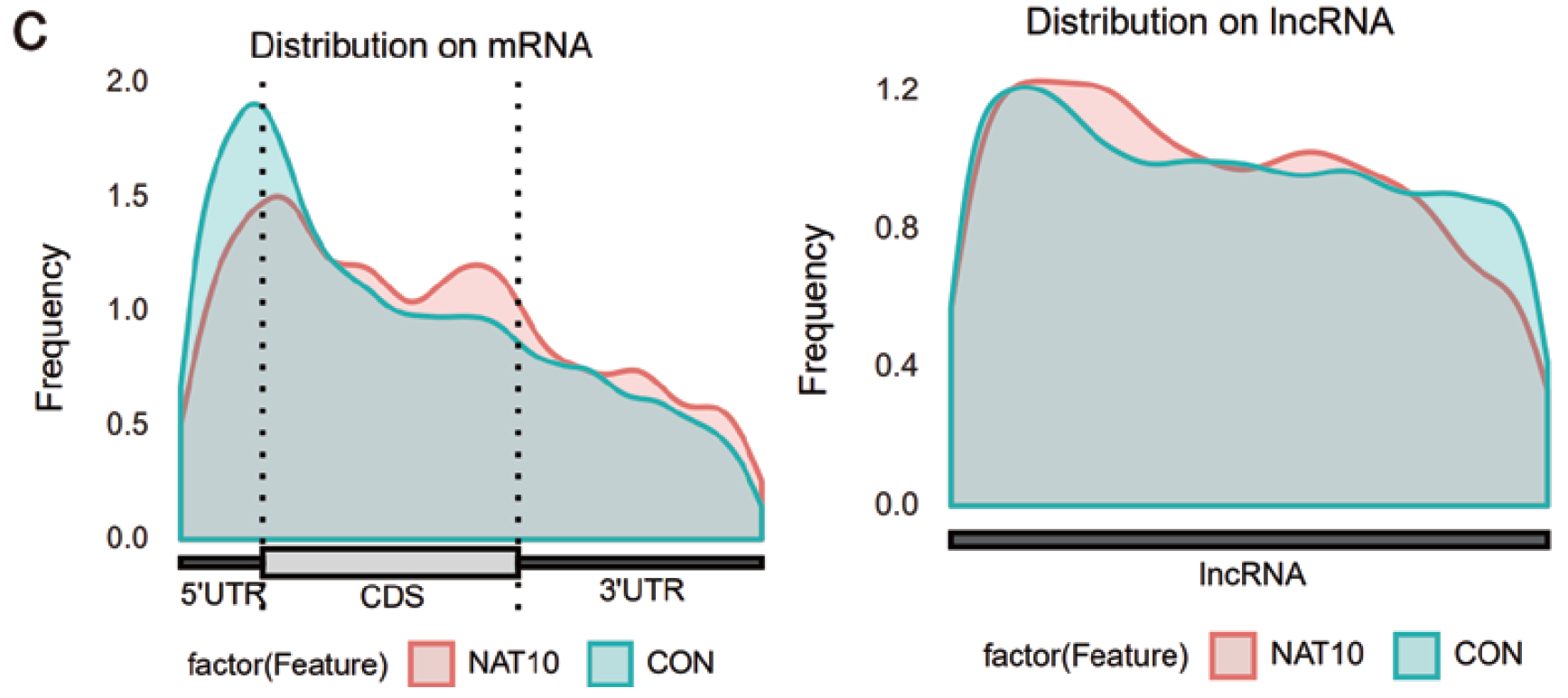 图8. NAT10过表达和对照组细胞的ac4C修饰metagene图
图8. NAT10过表达和对照组细胞的ac4C修饰metagene图
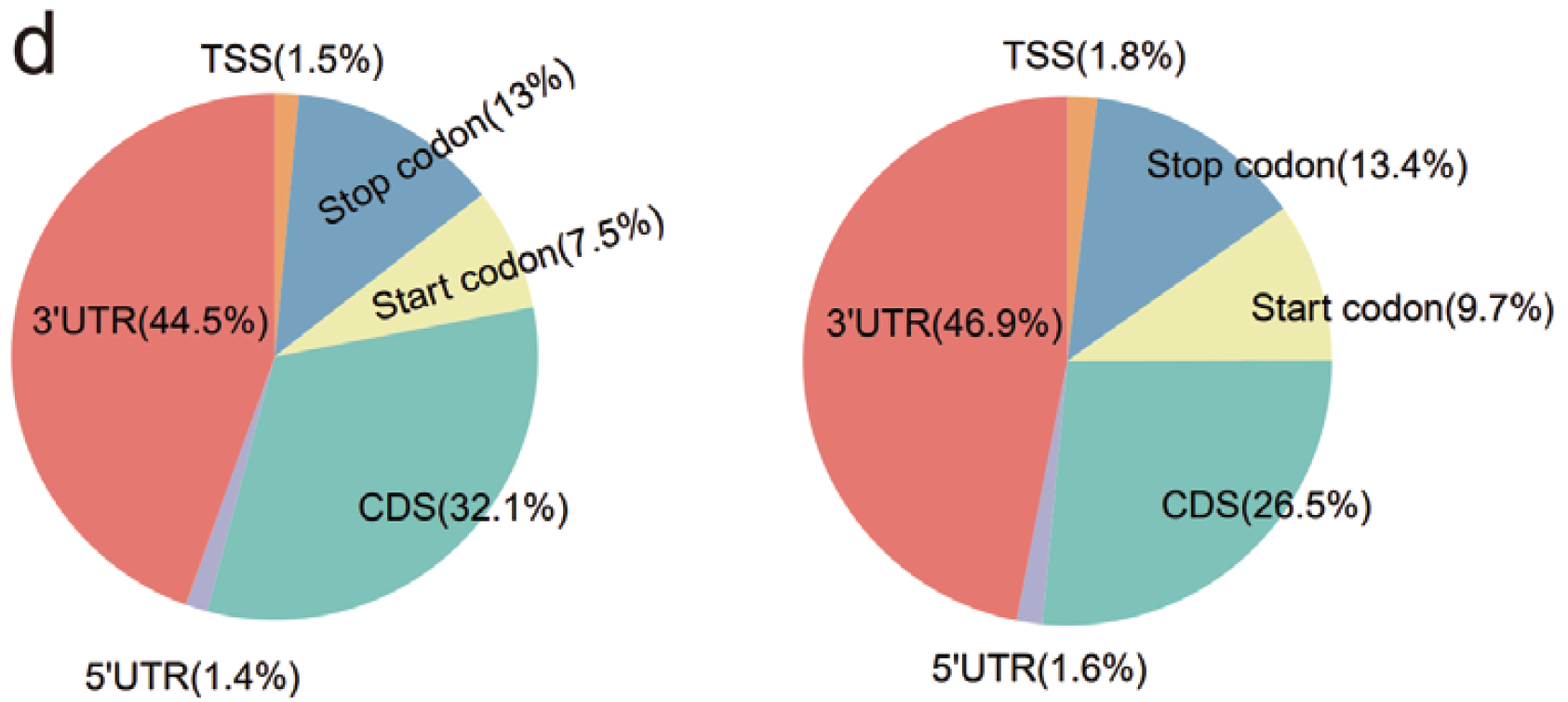
图9. ac4C peaks基因区间分布情况
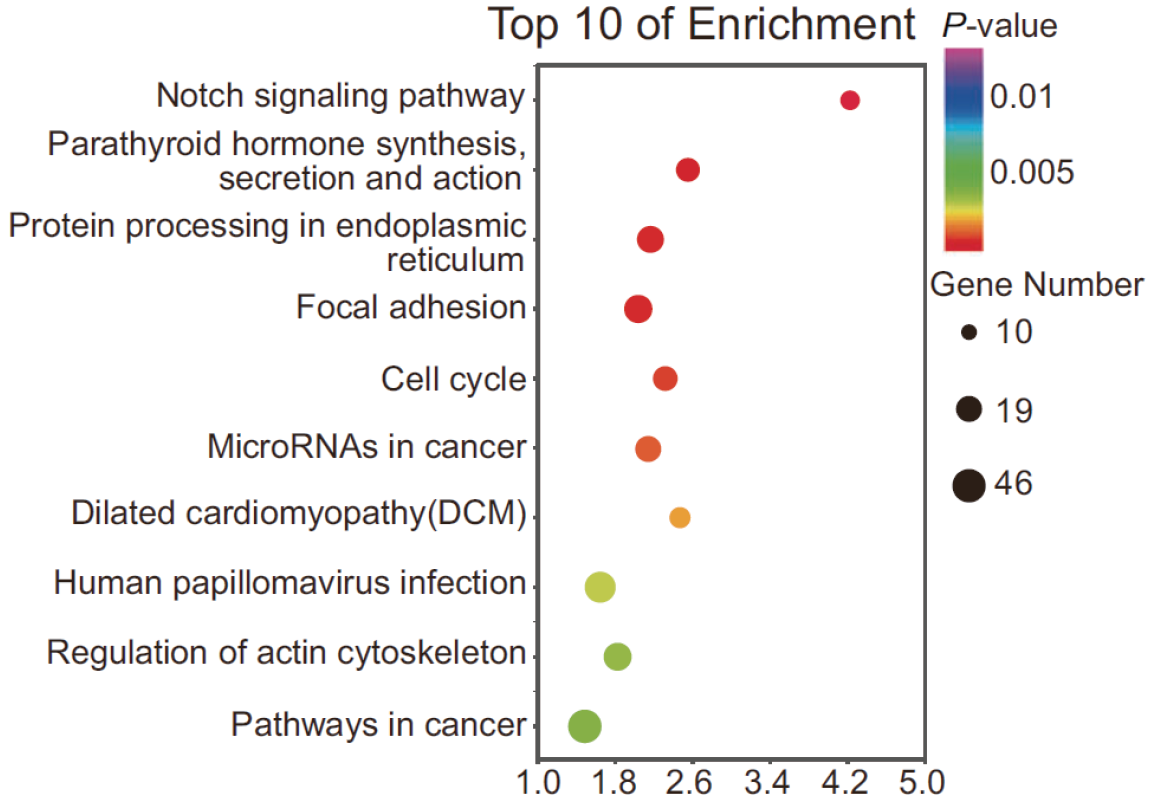
图10. KEGG富集分析显示,乙酰化mRNA编码的基因与NOTCH信号通路相关
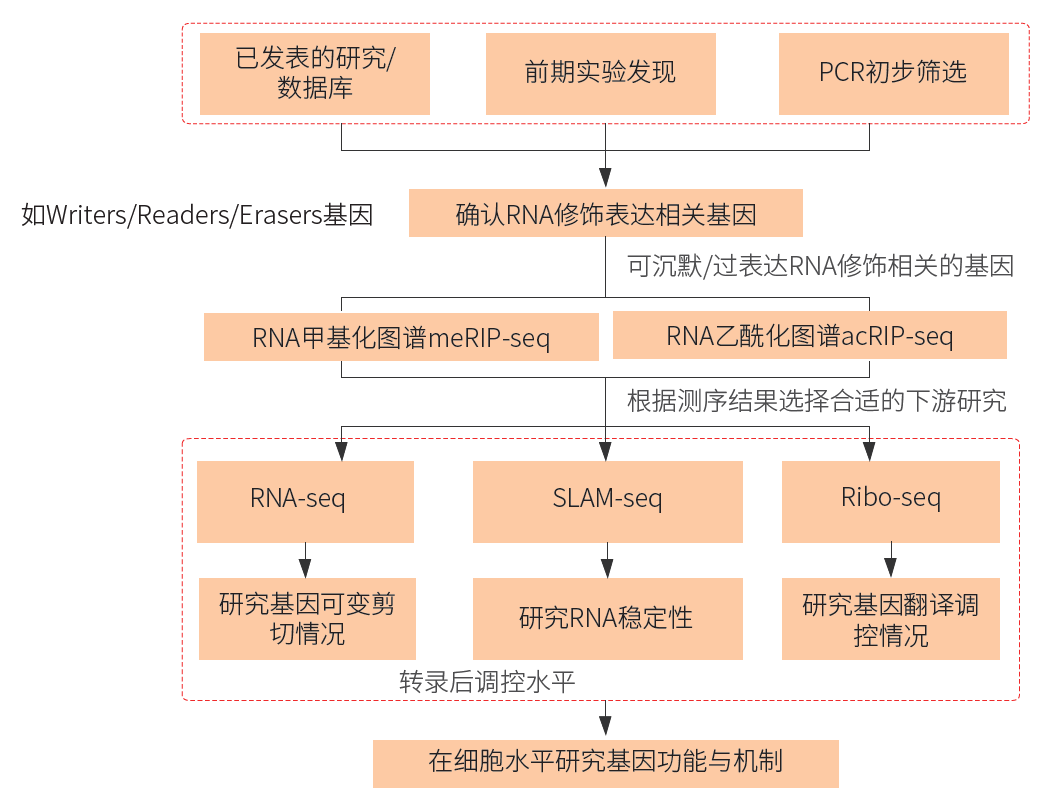
RNA修饰研究路径图
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
- 作者
- 内容
- 询问日期
 文献和实验
文献和实验兰州大学第一医院李汛团队首次揭示胃癌演进中mRNA乙酰化修饰新机制
4C 等。 其中, 2018 年 Arango D 等人首次报道了 mRNA 上存在 ac4C,也就是乙酰化修饰方式,功能上它可促进 mRNA 翻译上调 mRNA 稳定性。但目前在癌症领域尚未见 mRNA 乙酰化修饰的相关报道。 2021 年 5 月 3 日,兰州大学第一医院李汛教授课题组在 Signal Transduction and Targeted Therapy 杂志上在线发表 NAT10 promotes gastric cancer metastasis via N4
-Whitney检验),T C转化率仍然低于预期的测序错误率(图4c),在没有代谢标记的情况下,用碘乙酰胺处理总RNA并不影响定量基因表达分析 。 图4 SLAm-seq对s4U-标记的转录本进行定量 SLAm-seq可评估增强子的活性 在丰度相对稳定的,具有可比性的转录本中(~100 cpm),T>C reads数在特定转录本之间存在差异(图5a)。ESC特异性转录因子Sox2以及pri-miR-290-295,T>C reads数水平高,而管家基因GDPH T>C reads水平很低。这可能
,建议细胞活力大于 90% 以上。经过 PFA 交联过的细胞同样也适用于 CUT&Tag ,但我们不建议使用 PFA 交联的细胞进行实验,因为经过交联的细胞有可能会引起表位覆盖,从而降低数据质量。 3.针对于 Tn5 切割开放染色质区域的特性,CUT&Tag 是否更加适合于开放染色质区域的研究,异染色质上的位点是否适用? CUT&Tag 是 ChIP-seq 的替代技术,主要用于分析全基因组范围内的 DNA-蛋白质相互作用。容易将 ATAC-seq 技术与CUT
 技术资料
技术资料资料下载:
客户文章 李汛 教授兰州大学 acrip-seq.pdf 附 (下载 2 次)
客户文章【acRIP-seq】IF 37.6 Eur Heart J:NAT10 promotes vascular remodelling via mRNA ac4C acetylation.pdf 附 (下载 0 次)
请 [登录] 后再下载!