相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 询价记录
- 文献和实验
- 技术资料
- 服务名称:
微量meRIP-seq测序技术服务
- 提供商:
表观生物
- 规格:
10G*2(IP+input)
项目简介:
RNA 甲基化指发生在RNA 分子上不同位置的甲基化修饰现象,常见的RNA转录后修饰方式有6-甲基腺嘌呤(N6-methyladenosine,m6A) 和5-甲基胞嘧啶(C5-methylcytidine,m5C)等。
研究发现,m6A修饰在调控基因表达、剪接、RNA 编辑、RNA 稳定性、控制mRNA寿命和降解、介导环状RNA翻译等方面扮演重要角色。因此meRIP-seq助力解决细胞分化,生物发育、疾病发生发展,热休克反应等生物学问题。
表观生物率先开发微量meRIP-seq技术,通过技术优化,大幅降低meRIP的样本要求量,500ng-20ug 总RNA即可满足实验要求。利用创新的去rRNA技术,可以同时检测mRNA, lncRNA及环状RNA的甲基化修饰谱,帮助研究人员对RNA转录后甲基化修饰图谱进行全面研究,是表观转录组学研究的关键技术。
技术优势
1. 样本要求低,500 ng~20 μg总RNA即可
2. 分析内容再次升级,囊括高分文章热门图
3. 实验技术与生信分析技术过硬,motif 分析结果保证有显著RRACH 特征,如无可申请全额退款或者免费重测!
技术应用
1. 绘制全转录组的m6A修饰图谱
2. 研究肿瘤等疾病的m6A修饰异常表达
3. 研究各种生理/ 病理过程决定细胞命运的调控机制
送样要求
样本类型:
1. 总RNA,≥ 5 μg/ 样本,RIN 值≥ 6
2. 细胞,数量为5x106~107
3. 组织,≥ 10 mg/ 样本
样本物种:
仅限人、大小鼠物种,其他物种需评估
送样要求
基本分析
1.测序原始reads去接头,质量控制(QC)
2.参考基因组比对(Mapping)
3.富集区域鉴定(PeakCalling)
4.富集区域注释(PeakAnno)
5.lncRNA修饰分析、mRNA修饰分析
6.富集区域metagene plot图、pie plot图、venny图
7.Motif分析
8.Peak基因GO和KEGG分析
9.差异Peak分析(即差异RNA修饰mRNA、lncRNA)
10.差异Peak基因GO和KEGG分析
高级分析
1.单位点修饰预测分析(SingleSite)
2.差异RNA修饰热图(Heatmap)
3.累计分布曲线分析(Cumulative Distribution Fraction Analysis)
4.关联分析(与RNA-seq 关联分析或多组学关联分析)
5.关联分析四象限图
6.关联分析热图(Heatmap)
7.IGV峰图(附5个基因)
表观生物实测数据
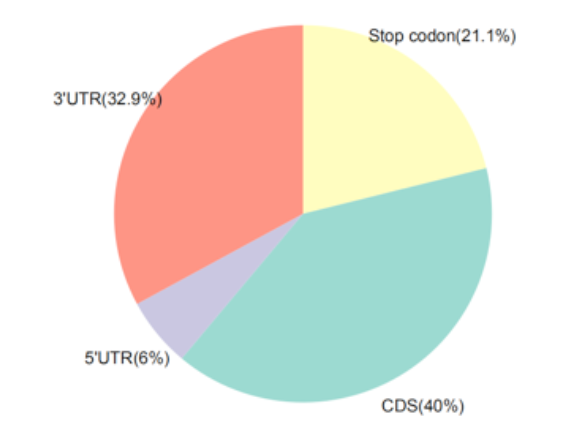
图1. Peak在RNA结构上的分布pie图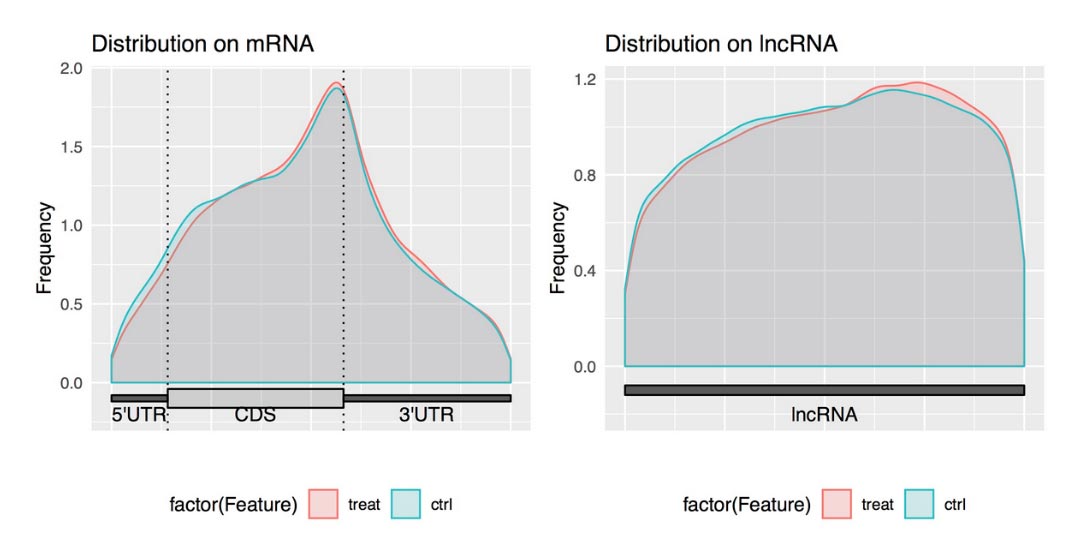
图2. Peak在RNA结构上的分布metageneplot图
图3. motif分析结果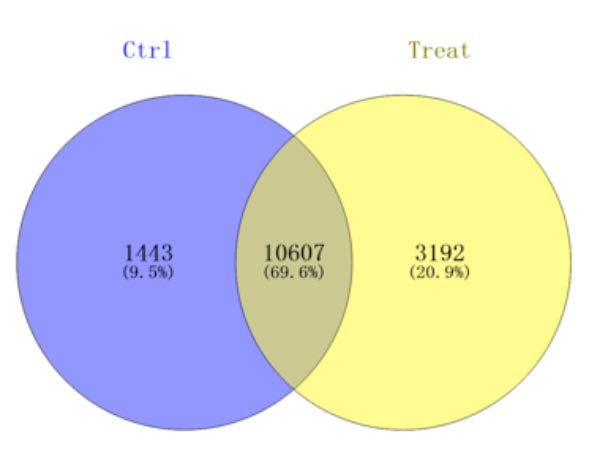
图4. RNA修饰位点交集韦恩图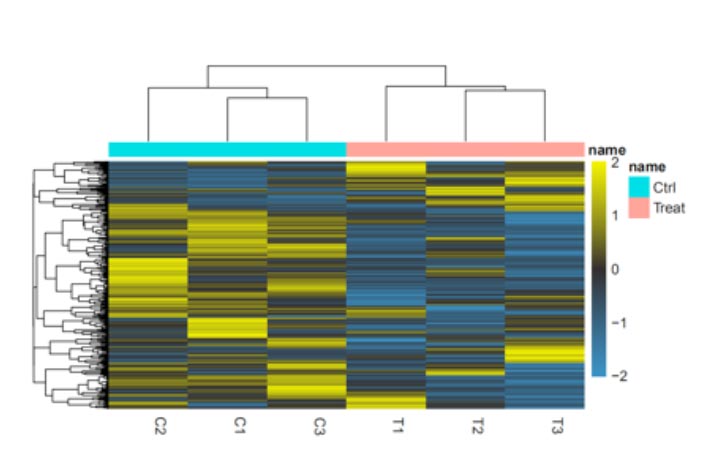
图5. 差异RNA修饰水平聚类热图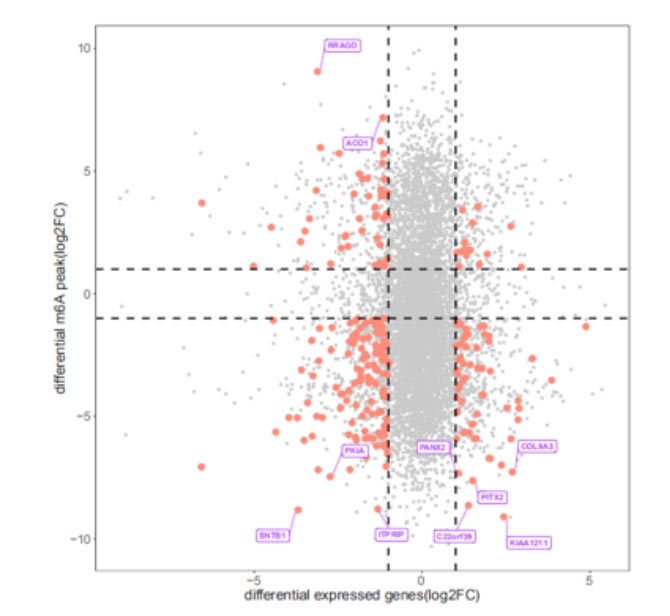
图6. 多组学关联分析四象限图(RNA-seq与meRIP-seq)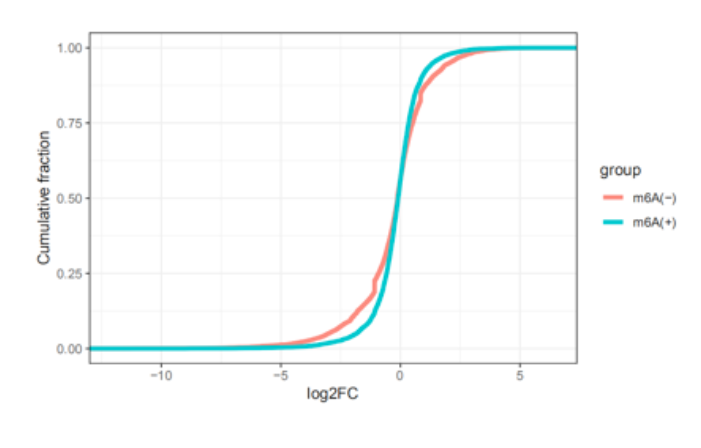
图7. 多组学关联分析累计分布图(RNA-seq与meRIP-seq)
图8. UCSC导出peak序列(与单位点修饰预测分析一致)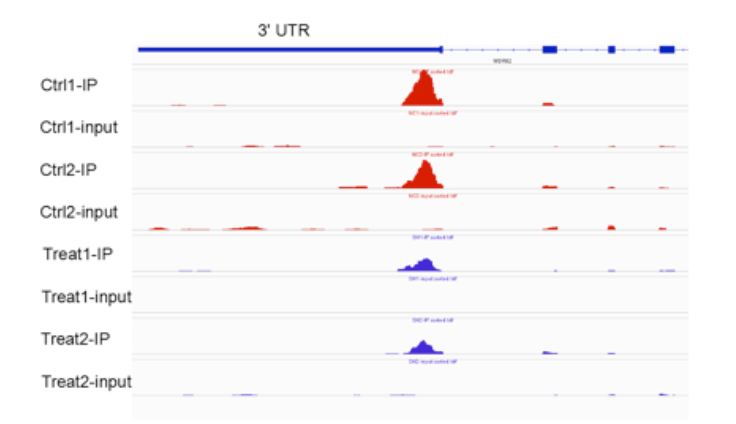
图9. IGV基因峰图
案例
Cell Stem Cell: m6A阅读器YTHDF2下调可抑制白血病 meRIP联合SLAM-seq、Ribo-seq,深入RNA修饰调控机制
本研究发现m6A阅读蛋白YTHDF2在AML患者身上广泛过表达,在小鼠和人类AML发生和发展过程中起着关键的作用。研究者敲除白血病前期细胞的YTHDF2,再进行meRIP-seq,发现敲除组中表达水平显著上调的mRNA具有丰富的m6A修饰。为了研究带有m6A修饰的mRNA的上调机制,研究者利用SLAM-seq检测mRNA的半衰期,证实m6A修饰使这些基因的半衰期延长了。
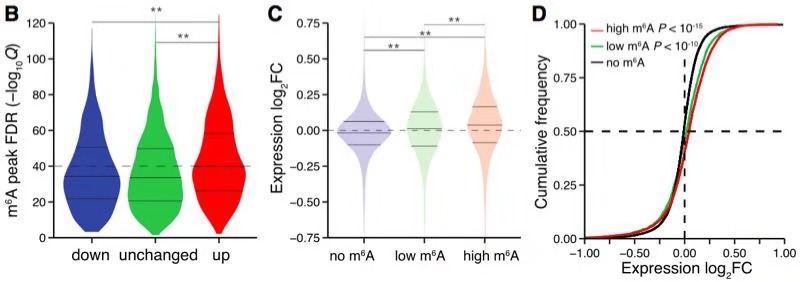
利用meRIP-seq分析Ythdf2敲除组和对照组的转录组m6A甲基化修饰图谱。B, Ythdf2敲除后mRNA表达减少、不变和增加的3个分组的m6A峰的错误发现率FDR。C, Violin图显示敲除组和对照组的表达变化,分了无m6A修饰、有m6A修饰和丰富m6A修饰的3个组做对比。D, 敲除组和对照组mRNA表达变化的累积分布曲线,敲除组有m6A修饰的mRNA表达上升。
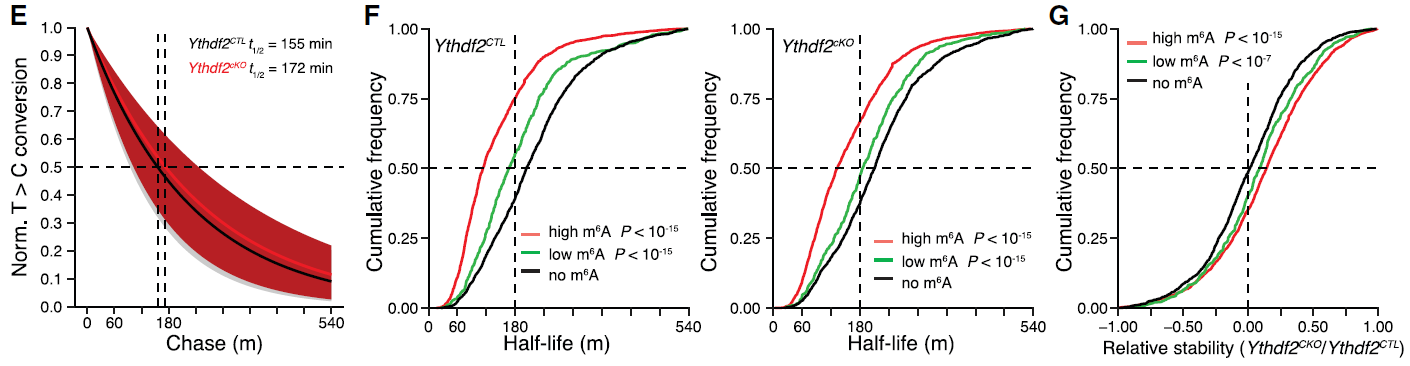
利用SLAM-seq分析Ythdf2敲除组和对照组的转录组半衰期。E,衰减曲线显示敲降组的mRNA半衰期稍微增长。G,YTHDF2的缺失,使含m6A修饰mRNA半衰期进一步增长。
随后,研究者利用RIBO-seq检测两个组的蛋白翻译效率,发现YTHDF2的缺失后,无论是有m6A修饰的还是没有m6A的mRNA,翻译效率都没有显著改变。这些结果表明YTHDF2在白血病中促进m6A mRNA的降解,而这个降解受m6A的指导。
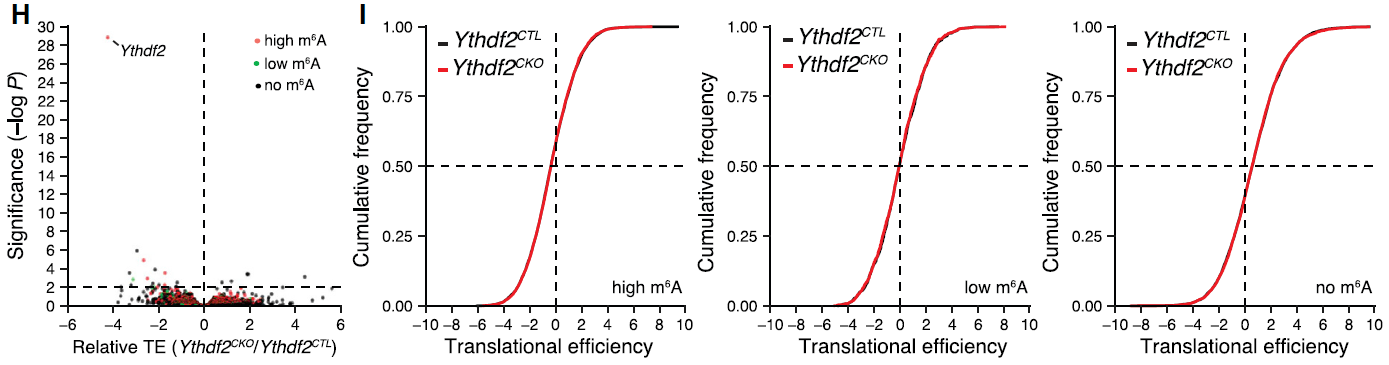
利用RIBO-seq分析Ythdf2敲除组和对照组的翻译效率。H, 火山图显示两组的翻译效率变化。I, 两组mRNA,m6A高水平、m6A低水平和无m6A修饰的mRNA分别的累计分布曲线。
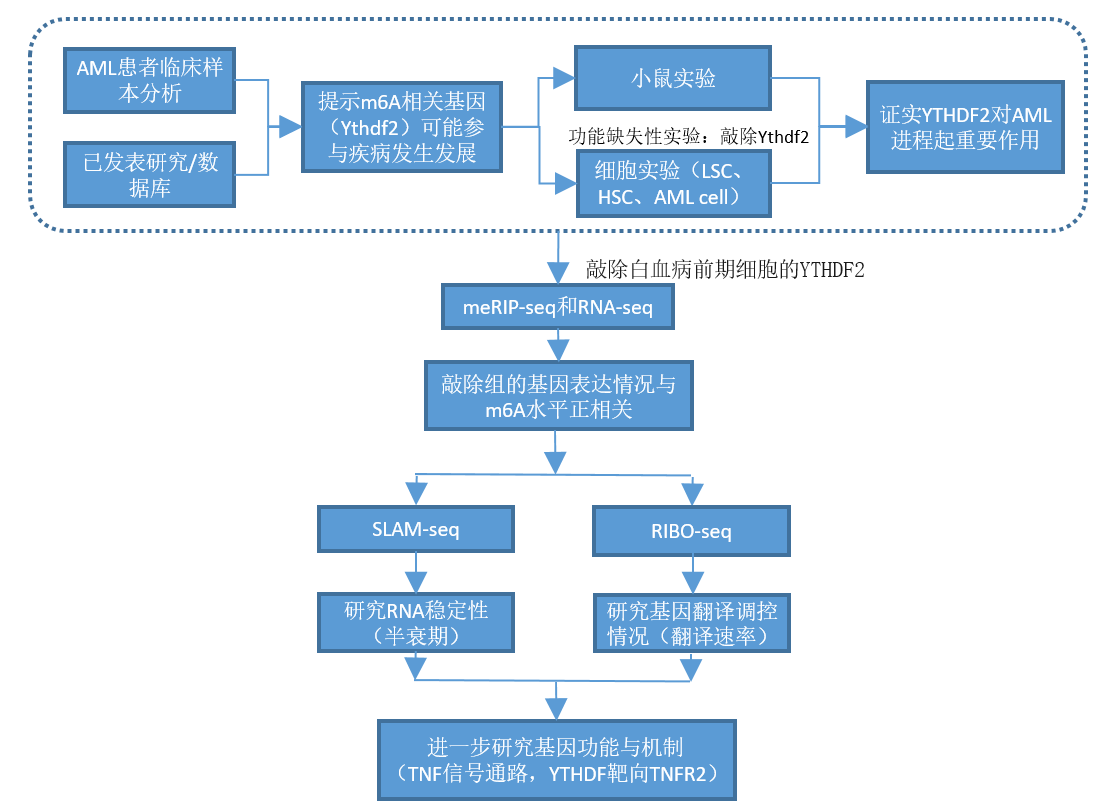
原文:Paris J, Morgan M, Campos J, et al. Targeting the RNA m6A Reader YTHDF2 Selectively Compromises Cancer Stem Cells in AcuteMyeloid Leukemia. Cell Stem Cell. 2019 Jul 3;25(1):137-148.e6.
客户文章
图10.周龄小鼠肝脏 mRNA m6A 修饰 metagene 图
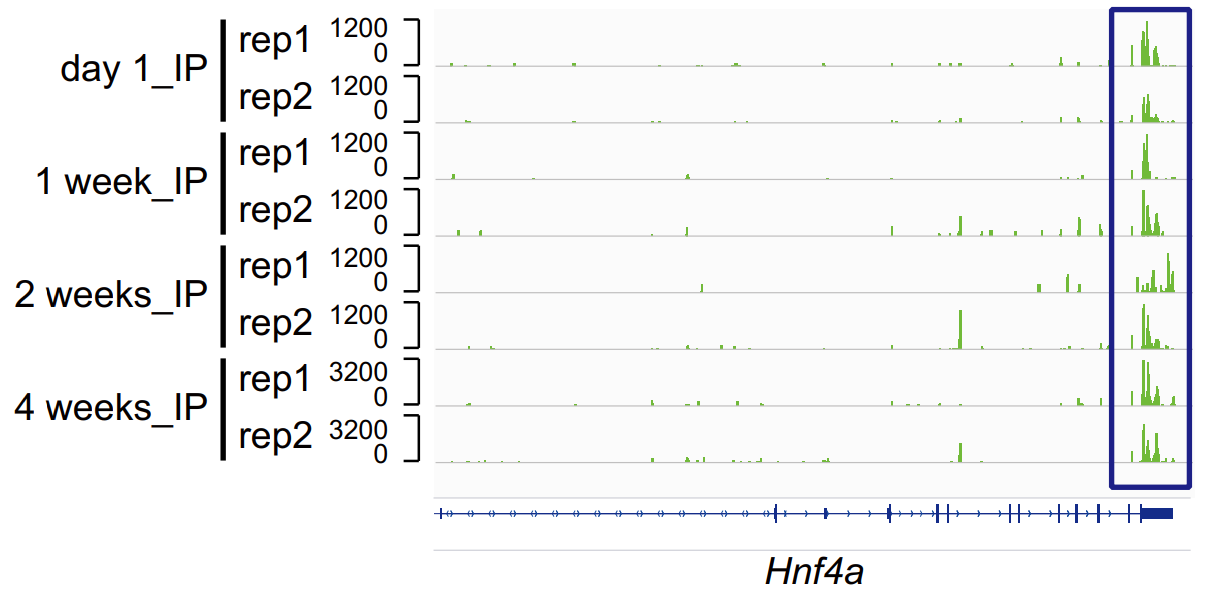
图11. 出生后 4 个不同时间点样本在 Hnf4a 位点的 m6A 修饰峰图

图12. m6A 修饰 Motif 分析
质量承诺:meRIP-seq motif分析结果须有显著的RRACH特征,如无可以申请全额退款或者免费重测!
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
- 作者
- 内容
- 询问日期
 文献和实验
文献和实验[1] Xu Y, Zhou Z, Kang X, et al. Mettl3-mediated mRNA m6A modification controls postnatal liver development by modulating the transcription factor Hnf4a. Nat Commun. 2022 Aug 5;13(1):4555. (meRIP-seq 客户文章)
[2] Jie Lai, Zhiyong Zhou, Kan Hu, et al. (2024) N6-methyladenosine methylation analysis of long noncodingRNAs and mRNAs in 5-FU-resistant colon cancer cells, Epigenetics, 19:1, 2298058 (meRIP-seq 客户文章)
[3] Zhang Y, Yan C, Xie Q, et al. Exposure to bisphenol A affects transcriptome-wide N6-methyladenine methylation in ovarian granulosa cells. Ecotoxicol Environ Saf. 2024 Mar 1;272:116071. (meRIP-seq 客户文章)
[4] Shi Y, Niu Y, Yuan Y, et al. PRMT3-mediated arginine methylation of IGF2BP1 promotes oxaliplatin resistance in liver cancer. Nat Commun. 2023 Apr 6;14(1):1932. (meRIP-seq 客户文章)
[5] Xu H, Lin X, Li Z, et al. VIRMA facilitates intrahepatic cholangiocarcinoma progression through epigenetic augmentation of TMED2 and PARD3B mRNA stabilization. J Gastroenterol. 2023 Sep;58(9):925-944. (meRIP-seq 客户文章)
[6] Li Y, He X, Lu X, et al. METTL3 acetylation impedes cancer metastasis via fine-tuning its nuclear and cytosolic functions. Nat Commun 2022 Oct 26;13(1). (meRIP-seq 客户文章)
[7] Liu L, Wu Y, Li Q, et al. METTL3 Promotes Tumorigenesis and Metastasis through BMI1 m6A Methylation in Oral Squamous Cell Carcinoma, Molecular Therapy (2020).(meRIP-seq 客户文章)
[8] Han D, Zhou T, Li L, et al. AVCAPIR: A Novel Procalcific PIWI-Interacting RNA in Calcific Aortic Valve Disease. Circulation. 2024;149(20):1578-1597. (客户文章)
Nat Commun:肖东/孙妍课题组合作揭示 m6A 修饰调节细胞自噬的新机制
27a 与 METTL3 结合减弱显著抑制 METTL3 泛素化降解,是导致饥饿时 METTL3 蛋白稳定性和丰度增加的主要原因。 图 2 YTHDF3 需要 METTL3 介导的 m6A 修饰来促进自噬 (图源:Hao WC, et al., Nat Commun, 2022) 为探究 YTHDF3 识别 mRNA m6A 修饰诱导自噬的具体机制,研究者运用饥饿诱导的 MEFs 细胞模型通过 RIP 和 MeRIP 测序(图 3a-f),并运用 RNA EMSA 等实验(图 3 g-k),证实 METTL3
K4me3 信号,发现 CUT&Tag 信号在分辨率和灵敏度上均优于 ChIP-Seq 信号。 图 5. CUT&Tag 与 ChIP-Seq IGV 视图比较【3】 该研究成果为进一步研究植物中的表观基因组学提供了一个有前途的策略。在操作简单性和所需的时间方面,CUT&Tag 优于传统染色质分析方法。更重要的是 CUT&Tag 需要更少的起始材料,更少的测序数据得到更高质量的数据。 参考文献: 【1】 Kaya-Okur HS, Wu SJ
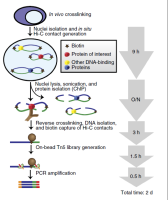
原理: 将染色质和与之相互作用的转录因子和组蛋白通过甲醛等物质交联起来,然后通过超声将染色质打碎成小片段,加入针对特定转录因子或特殊修饰的组蛋白抗体,通过 Protein A/Protein G 微球或磁珠将抗体-转录因子-染色质复合物拖下来,通过 PCR 或测序的方法检测与目的蛋白相结合的 DNA 序列,进而研究这些转录因子在细胞发育或者生长中的作用位点。 ✦ ChIP-seq: ChIP-seq 将 ChIP 技术与二代测序相结合,将 ChIP 下来的 DNA 进行二代测序文库构建,能够获取
 技术资料
技术资料