相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 询价记录
- 文献和实验
- 技术资料
- 提供商:
表观生物
- 服务名称:
m6Am-Exo-seq测序服务
项目简介:
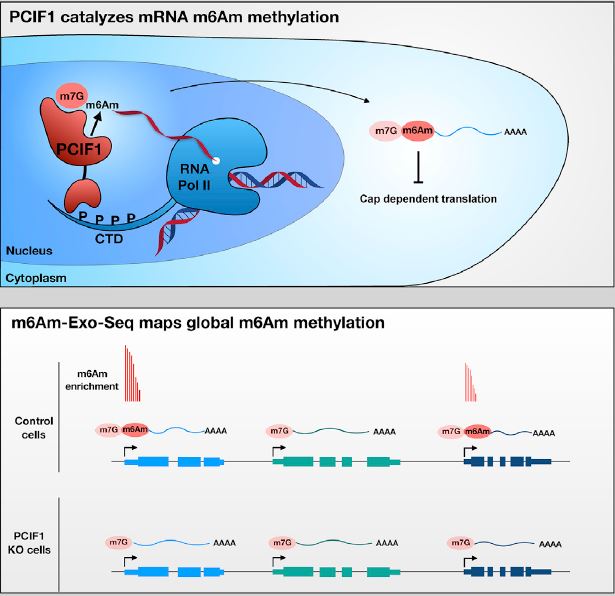
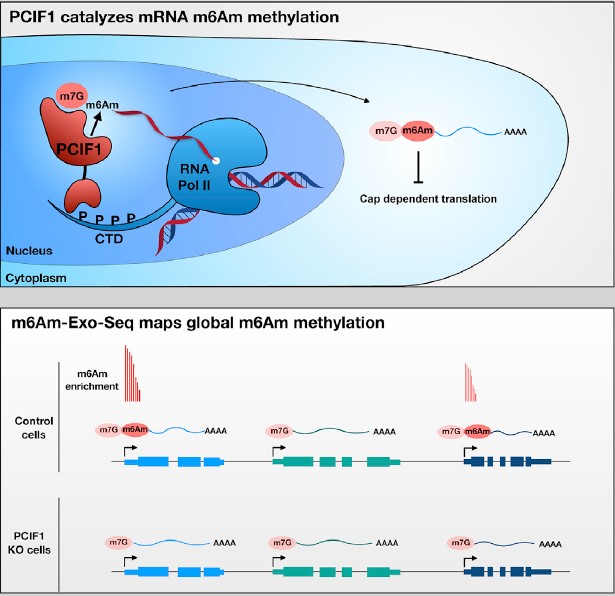
RNA上存在一百多种化学修饰,其中N6-甲基腺嘌呤(m6A)作为真核生物mRNA上含量最普遍的化学修饰,参与调控了很多重要的生物学过程。不同于m6A,m6Am主要位于真核生物mRNA 5'端帽子之后的第一个碱基。早在1975年,科学家就鉴定到了m6Am的存在。m6Am也是一个动态、可逆的修饰,但其修饰酶PCIF1近两年才发现。m6Am修饰主要位于5’m7Gppp帽子后的第一个碱基,主要通过影响mRNA的蛋白翻译效率发挥作用。
为推动表观转录修饰m6Am的研究,表观生物新开发m6Am-Exo-seq测序技术,大幅降低样本要求量,可以在全转录组范围(mRNA及lincRNA)高效准确的鉴定m6Am的修饰位点及丰度变化。
技术流程:
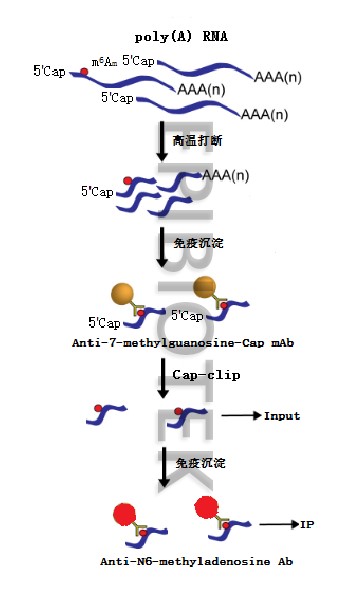
送样要求:
样本类型:
1. 总RNA,100ug
2. 细胞,数量为5x107
3. 组织,质量为100mg
样本物种:
仅限人、大小鼠物种,其他物种需评估
分析内容
基本分析
1.测序原始reads去接头,质量控制(QC)
2.参考基因组比对(Mapping)
3.富集区域鉴定(PeakCalling)
4.富集区域注释(PeakAnno)
5.lncRNA修饰分析、mRNA修饰分析
6.富集区域metagene plot图、pie plot图、venny图
7.Motif分析
8.Peak基因GO和KEGG分析
9.差异Peak分析(即差异RNA修饰mRNA、lncRNA)
10.差异Peak基因GO和KEGG分析
高级分析
1.单位点修饰预测分析(SingleSite)
2.差异RNA修饰热图(Heatmap)
3.累计分布曲线分析(Cumulative Distribution Fraction Analysis)
4.关联分析(与RNA-seq 关联分析或多组学关联分析)
5.关联分析四象限图
6.关联分析热图(Heatmap)
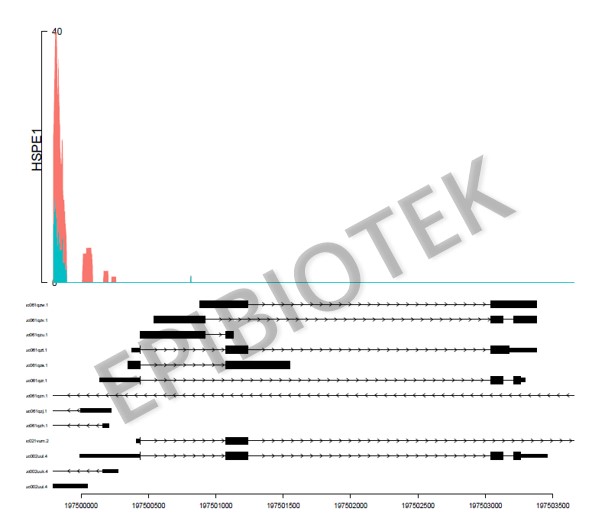
7.IGV峰图(附5个基因)
表观生物实测数据:

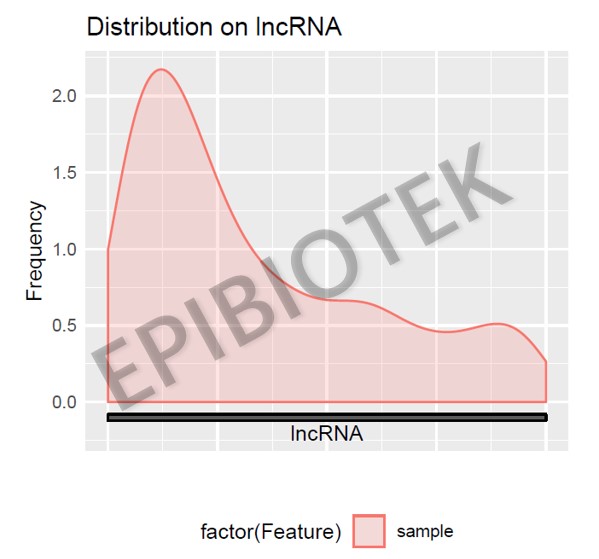
图4. Peak在lincRNA结构上的分布metageneplot图

图5. motif分析结果(CA特征,不同于m6A修饰的RRACH)
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
- 作者
- 内容
- 询问日期
 文献和实验
文献和实验1.Sendinc E, Valle-Garcia D, Dhall A, et al. PCIF1 catalyzes m6Am mRNA methylation to regulate gene expression[J]. Molecular cell, 2019, 75(3): 620-630. e9.
2.Mauer J, Sindelar M, Despic V, et al. FTO controls reversible m 6 Am RNA methylation during snRNA biogenesis[J]. Nature chemical biology, 2019, 15(4): 340-347.
3.Akichika S, Hirano S, Shichino Y, et al. Cap-specific terminal N6-methylation of RNA by an RNA polymerase II–associated methyltransferase[J]. Science, 2019, 363(6423): eaav0080.
4.Sun H, Zhang M, Li K, et al. Cap-specific, terminal N 6-methylation by a mammalian m 6 Am methyltransferase[J]. Cell research, 2019, 29(1): 80-82.
5.Li K, Cai J, Zhang M, et al. Landscape and regulation of m6A and m6Am methylome across human and mouse tissues[J]. Molecular cell, 2020, 77(2): 426-440. e6.
之前剪切底物。但 ACA 序列的第一个 A 发生 m6A 甲基化之后将无法被 MazF 所识别。MazF RNA 酶作用示意图根据这一原理,作者将纯化后的 mRNA 进行 MazF 酶处理,然后再对打断的 RNA 进行逆转建库测序。对于无修饰的位点,ACA 处被完全剪切,测序 reads 正好分布于该位点上下游而且比对至该处的上下游 reads 数是相同的。而甲基化位点的 reads 会包含上下游的序列。作者开发了 MAZTER-MINE 软件包专门进行分析。MAZTER-Seq 实验
魔高一尺,道高一丈!清华大学徐萌团队发现能量代谢与肿瘤免疫逃逸的关系
人员对 B16-OVA 肿瘤细胞进行 M6A-seq。通过计算每个峰 m6A 比率,研究人员发现 Fto 敲降导致 m6A 甲基化整体增加。研究人员进一步挑选了 83 个 FTO-m6A 直接调控基因进行 GO 分析,发现 DNA 结合和转录激活显著富集,包括 bZIP 转录因子 Jun 和 Cebpb。为了分析这些 mRNA 中 m6A 增加是否与 FTO 直接相关,研究人员在肿瘤细胞中过表达了 CRISPR-dCas13 融合蛋白,发现在 gRNA-Cebpb-JunB 和 dCas13b-FTO 共转
Mol Cell:浙大王青青团队等合作揭示乳酸化调控 RNA m⁶A 修饰促进肿瘤浸润髓系细胞的免疫抑制功能
削弱其促进肿瘤的能力。RNA-seq 分析提示 JAK-STAT 可能是 METLL3 调控的下游通路,TIMs 的 JAK-STAT 通路对其促肿瘤作用具有枢纽性作用(2022 年是 JAK-STAT 通路发现 30 周年 [6])。作者利用 m6A 甲基化免疫共沉淀测序分析,证实 TIMs 中 Jak1 的 mRNA 上 m6A 修饰增加,以 m6A-YTHDF1 依赖形式提高其在多聚核糖体的翻译效率。JAK1 促进转录因子 STAT3 的磷酸化,启动 STAT3 下游免疫抑制效应分子 IL