相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 询价记录
- 文献和实验
- 技术资料
- 提供商:
湖北佰莱博科技有限公司
- 服务名称:
分子动力学模拟技术服务
- 规格:
1次
佰莱博分子动力学模拟(Molecular Dynamics Similation)技术服务
佰莱博生物专业的计算生物学研究团队,配置有多套高性能计算机设施用于计算生物学服务,可以提供分子动力学模拟、分子对接、虚拟筛选,基于AI的蛋白质结构设计等“干”实验服务;同时还提供基于SPR、 MST、BLI、ITC等生物物理技术、生物化学及细胞生物学分子相互作用等“湿”实验服务。为大学、科研院所及生物制药企业提供全流程分子互作研究与创新药物开发解决方案。
1.分子动力学模拟简介
分子动力学模拟是一种以分子的经典力学(分子力场)模型,通过数值求解分子体系的运动方程,研究分子体系的结构与性质的计算机模拟方法。分子动力学模拟的本质就是通过宏观性质来研究微观粒子的一种方法,这种方法使用牛顿运动定律来追踪分子体系中每个原子随时间的运动。分子动力学模拟是分子模拟中最接近实验条件的模拟方法,能够从原子层面给出体系的微观演变过程,直观地展示实验现象发生的机理与规律,促使我们的研究向着更高效,更经济,更有预见性的方向发展。
在分子动力学模拟中,分子体系内的原子被赋予初始位置和速度,然后根据它们之间的相互作用力(这些力可以基于经验势函数或更复杂的量子力学计算),计算出每个原子的加速度。通过数值积分方法(如Verlet算法或其变种),逐步推进时间,可以计算出原子在下一时刻的位置和速度,这样就可以模拟出整个体系随时间的演化过程。
常用的分子动力学模拟软件包括AMBER、CHARMM、GROMACS等,其中GROMACS是一款开源软件,可以用于上百万个粒子体系的分子动力学模拟研究,适合大型系统和长时间尺度。
2.分子动力学模拟的应用
1) 配体-受体相互作用:模拟配体和受体的结合模式,研究分子识别的机制,评估配体和受体的亲和力,从而指导新药的设计。
2) 药物动力学:模拟药物分子在体内的吸收、分布、代谢等的过程,预测药物的生物利用度和代谢。
3) 蛋白质结构和功能:研究蛋白质的动态行为、构象变化和功能机制。
3.技术流程(通用步骤,具体按照项目微调)
1) 获取蛋白的3D结构(PDB格式),如没有蛋白的3D结构,需要对蛋白质进行结构预测
2) 受体-化合物体系需对化合物进行结构优化,对受体蛋白进行活性位点预测,随后进行分子对接,从分子对接结果出发,进行动力学模拟。
3) 将蛋白进行预处理,并生成拓扑结构(如果是受体配体体系,也需将配体进行预处理,生成拓扑结构)
4) 依次进行模拟体系溶剂化(如水溶剂体系)、能量最小化(找到局部势能最小值)、温度平衡(NVT控温)、压强平衡(NPT控压)
5) 启动分子动力学模拟
6) 根据客户需求进行数据分析
图1 体系溶剂化后图片
4.结果分析
1) 根均方偏差(Root Mean Square Deviation,RMSD):衡量模拟过程中结构相对于初始结构的平均偏离程度,用于判断结构是否稳定。
2) 根均方波动(Root Mean Square Fluctuation,RMSF):评估每个原子或残基在模拟过程中的波动幅度,可以揭示哪些区域最灵活或者最稳定。
3) 回旋半径(Radius of Gyration):反映了分子的整体紧凑型或展开程度,用于研究蛋白的折叠状态。
4) 氢键分析:分析蛋白质内部或蛋白质与其他分子之间形成的氢键数量和稳定性。
5) SASA(溶剂可及表面积):测量了蛋白质可被水溶剂接触的比例,可用于预测相互作用中发生构象变化的幅度。
6) MMPBSA(分子力学/泊松-玻尔兹曼表面积):用于计算蛋白与配体的结合自由能,绝对值越大,二者的结合能力越强。
5.结果交付
示例:磷酸化修饰对蛋白结构的影响
5.1 RMSD分析
均方根偏差(Root mean square deviation, RMSD)表示某一时刻的构象与目标构象所有原子偏差的加和,是衡量体系是否到达稳定的重要依据,可用于量化蛋白质三级结构的稳定性。具体而言,RMSD 值较小且稳定表明分子体系在模拟过程中保持了与参考构象相似的结构,显示其三级结构的稳定性。相反,RMSD 值的显著变化则可能指示分子发生了构象转变或解折叠过程。一般情况下,RMSD 值的差值(ΔRMSD)<0.2nm 时,认为结果是平稳的状态。
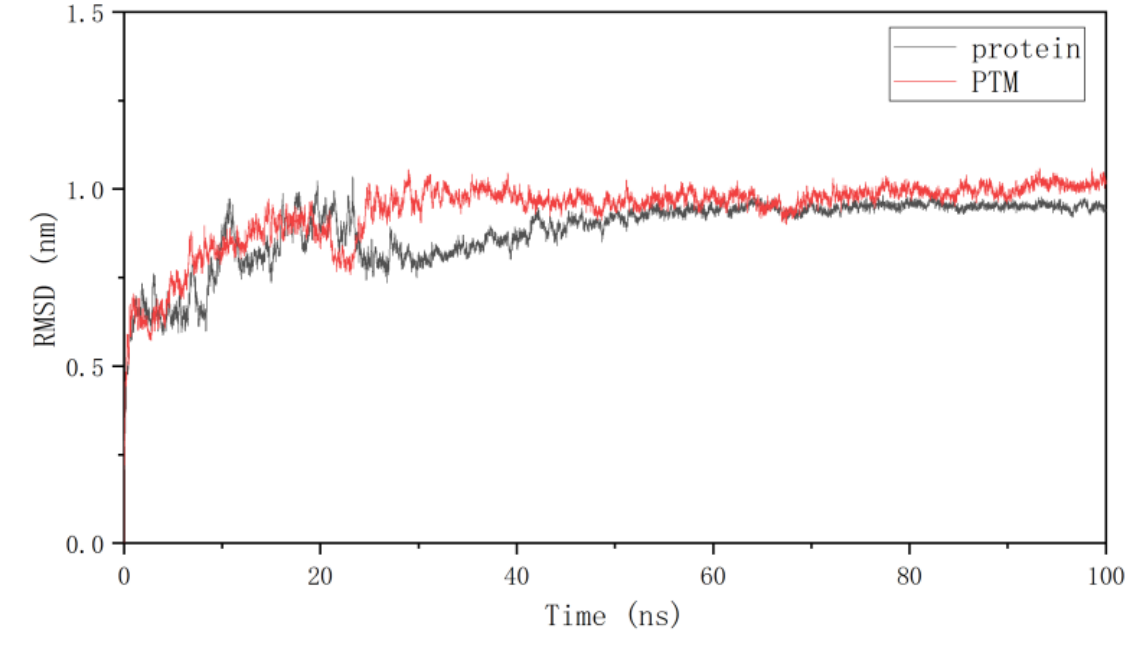
图2 蛋白的RMSD随时间变化图
5.2 RMSF分析
均方根波动曲线(Root Mean Square Fluctuation, RMSF)代表蛋白质中氨基酸在动力学模拟过程中的波动程度。通过计算每个残基在整个模拟过程中相对于其平均位置的波动,RMSF 提供了一个反映局部动态行为的度量。
较高的 RMSF 值代表该氨基酸具有较大的波动,相反,较低的 RMSF 值代表该氨基酸波动小。通过分析 RMSF 曲线,可以了解蛋白质中哪些区域具有较高的动态性,哪些区域较为稳定。这对于理解蛋白质的功能和动力学行为具有重要意义。例如,高 RMSF 值区域可能对应于蛋白质的柔性环或无规则区,而低 RMSF 值区域可能对应于结构较为稳定的二级结构元素,如 α-螺旋和 β-折叠。
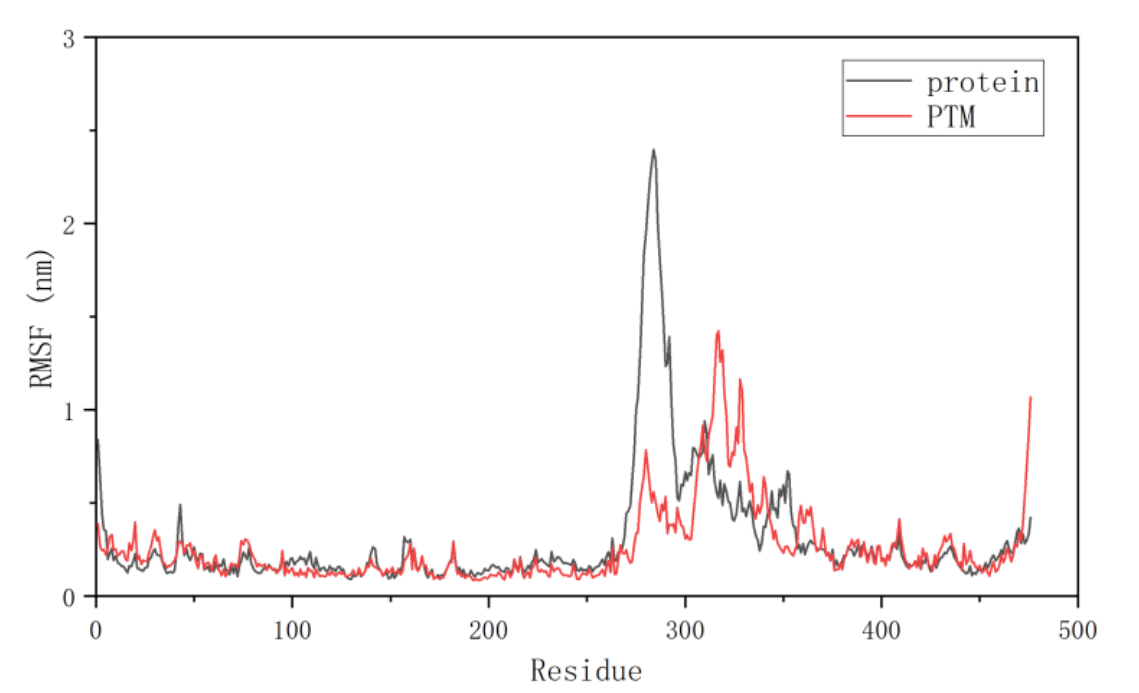
图3 蛋白每个残基的RMSF值
5.3 Rg分析
回转半径(Radius of Gyration, Rg)是衡量蛋白质结构紧密度的重要参数,表示蛋白质中所有原子的质量中心与其几何中心之间的平均平方距离。Rg 值越小,说明蛋白质结构越紧密;相反,Rg 值越大,说明蛋白质结构越松散。
通过计算和分析 Rg 值,可以了解蛋白质在动力学模拟过程中整体构象的变化。较低的 Rg 值通常对应于较为紧密和稳定的结构,如折叠良好的蛋白质核心区域;较高的 Rg 值则可能表明蛋白质结构较为松散或展开。Rg 的变化趋势能够反映出蛋白质在不同条件下的构象稳定性。例如,在蛋白质折叠过程中,Rg 值会逐渐减小,表明蛋白质逐渐形成紧密的折叠结构。而在蛋白质解折叠过程中,Rg 值则会逐渐增大,表明蛋白质结构逐渐展开。
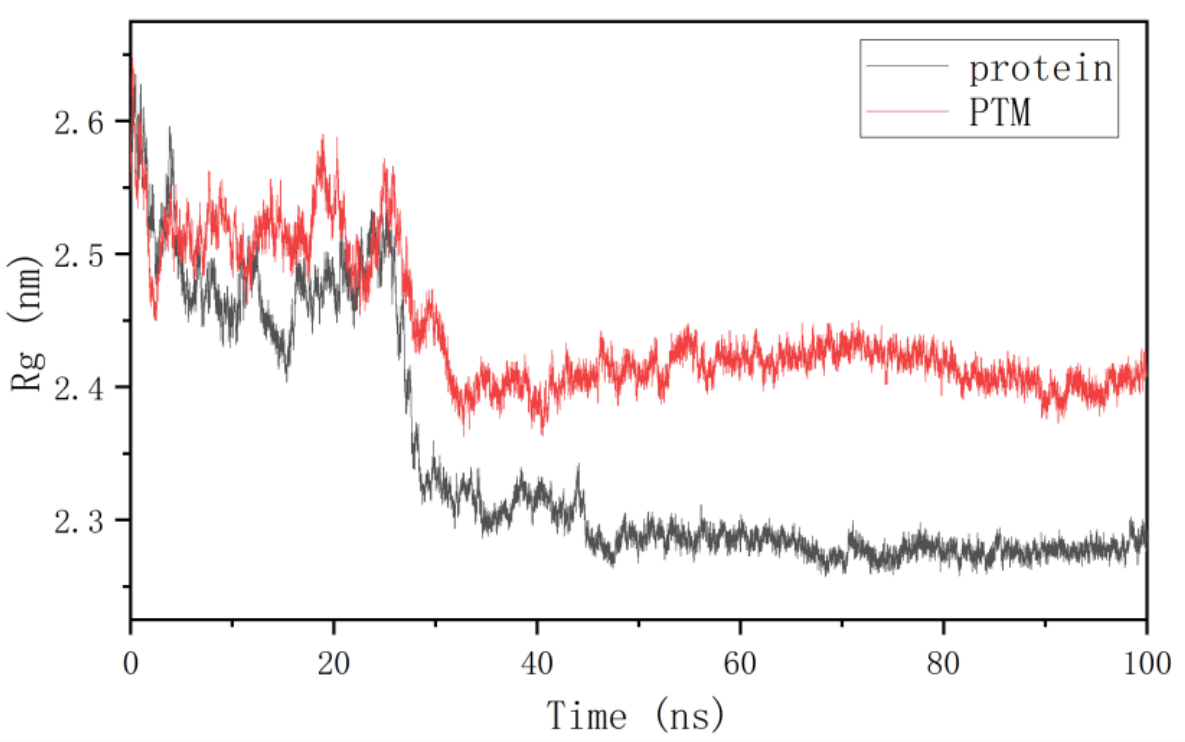
图4 蛋白的Rg随时间变化图
5.4 自由能地貌图
自由能地貌图(Free Energy Landscape,简称 FEL)是用于描述分子体系在不同构象或状态之间自由能变化的图形化工具。通过将 Rg 和 RMSD 值这两个参数作为坐标,并通过统计方法计算对应的自由能分布,可以揭示分子在不同构象和状态下的稳定性和动力学信息。
根据图中的能量图可以看到,蓝色区域为体系中具有稳定构象的能量区域。这个区域表示体系在这些构象下自由能最低,显示了 Hmgcs1 蛋白结构在这些状态下的高稳定性。通过分析这个蓝色区域,可以识别出最稳定的蛋白构象以及可能的折叠路径和关键中间态,这对于理解蛋白质的功能和动力学行为具有重要意义。
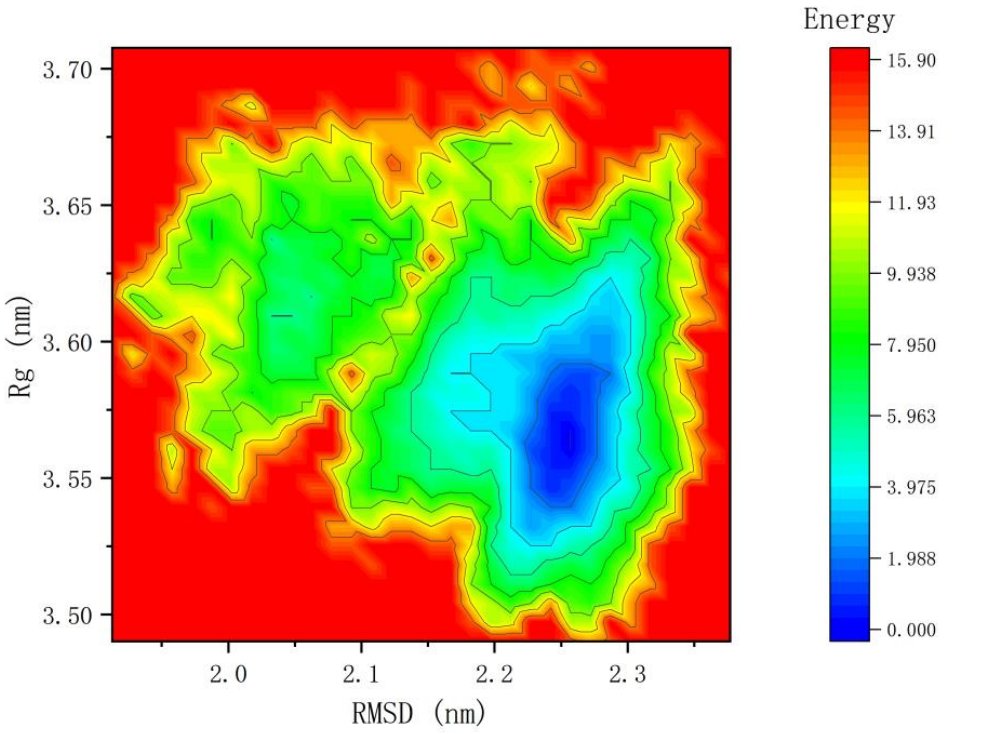
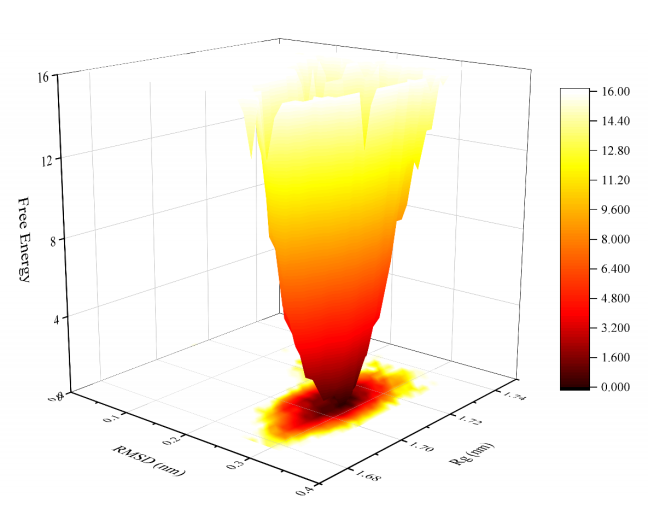
图6 自由能地貌图(2D和3D)
个性化分析部分(根据具体的项目以及预期结果需要进一步沟通)
A、 小分子与蛋白结合情况(MM/PBSA、残基分解)
对模拟轨迹进行周期消除后,结合前述 RMSD 等分析,提取蛋白复合物于 20 ns - 100 ns 时间段的运动轨迹进行 MM/PBSA 结合自由能计算,共选取 1000 帧结构进行计算。
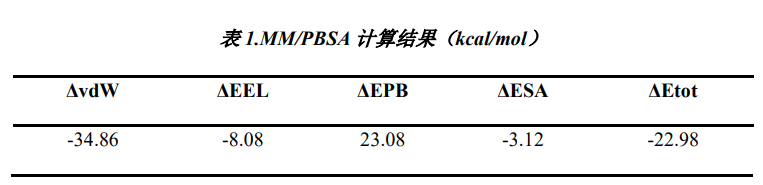
注:各项解释
ΔvdW (Van der Waals Energy):范德华相互作用能量,反映了分子之间的非极性相互作用;
ΔEEL(Electrostatic Energy):电荷之间的静电相互作用的能量;
ΔEPB(Polar Solvation Energy):极性溶剂化能,利用 Poisson-Boltzmann 方程来计算分子在极性溶剂中的溶剂化能;
ΔESA(Non-Polar Solvation Energy):非极性溶剂化能,通常通过溶剂可接触表面积乘以一个经验参数得到;
ΔEtot (ΔGbind):结合自由能总值,用于评估分子之间的结合自由能。
为更深入地分析小分子与蛋白的相互作用及强度,本节计算了每个残基对结合自由能的贡献。
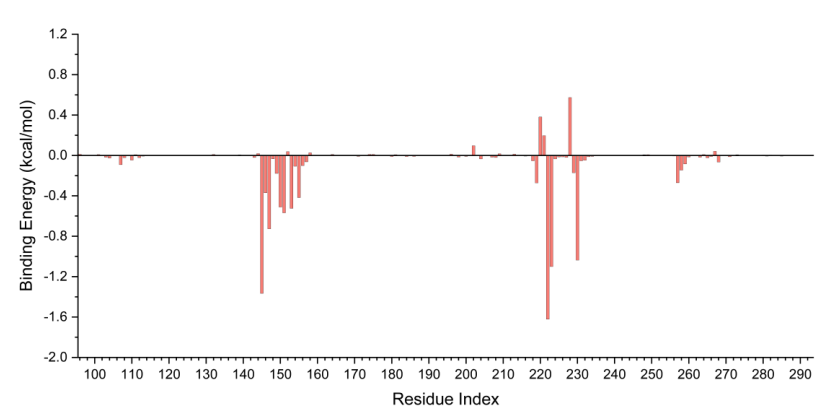
图7 结合自由能残基分解
B、 小分子化合物与蛋白结合情况指纹图谱
在药物设计过程中,往往需要对化合物的结构进行修饰改造,可以通过这个结合情况图谱来进一步分析化合物各部分的结合情况,从而进一步对化合物结构进行改造优化。
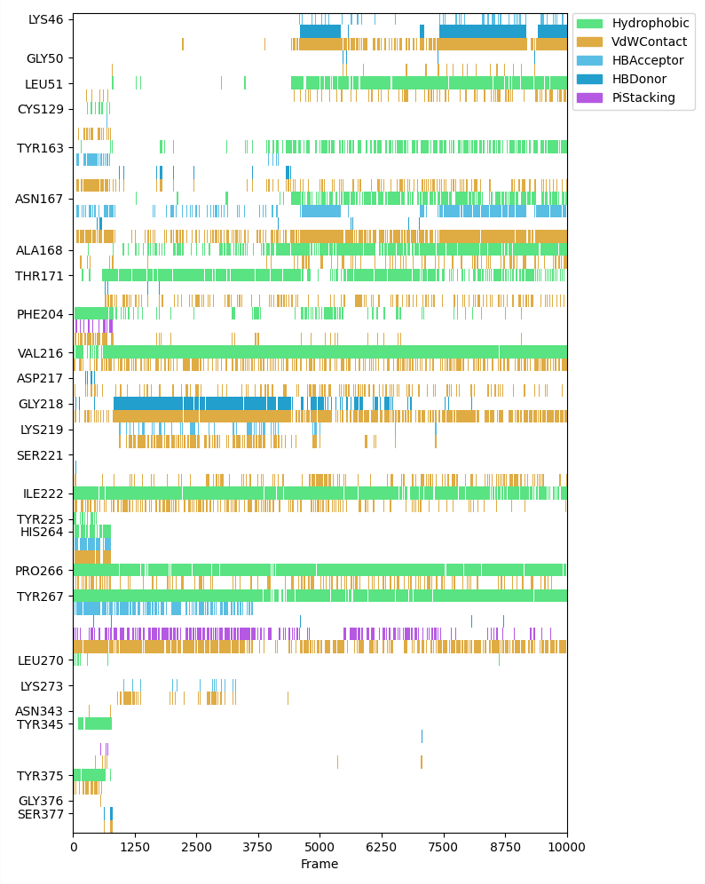
图8 小分子化合物与蛋白结合指纹图谱
C、SAPT能量分解
以对称匹配微扰理论(Symmetry-Adapted Perturbation Theory, SAPT)为基础,将分子间相互作用能分解为不同物理项,从能量角度更深入地理解分子间相互作用的本质。
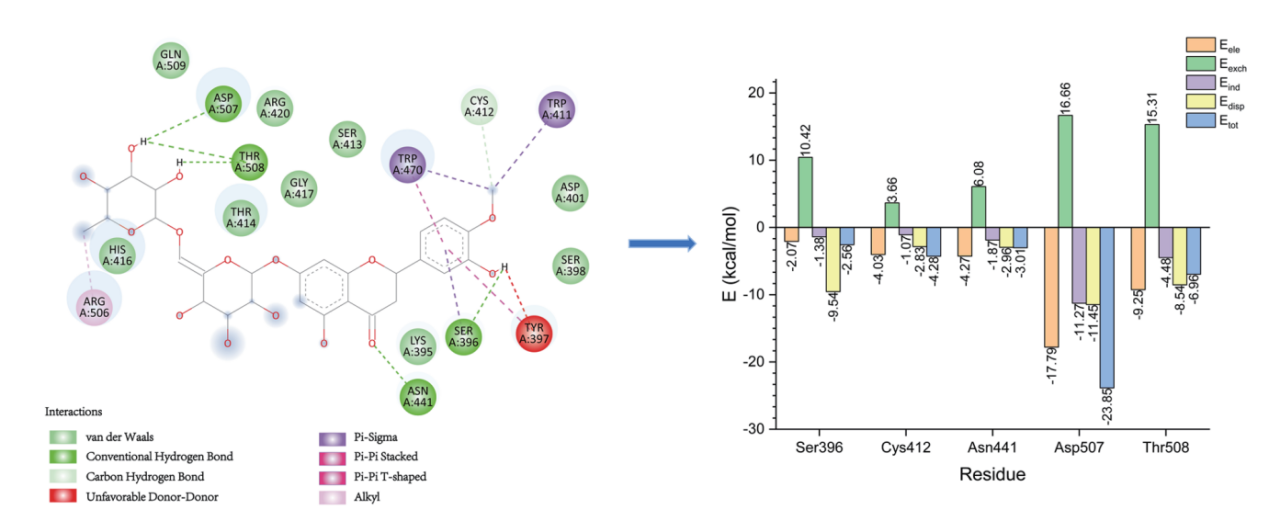
图9 小分子与蛋白弱相互作用分析
D、 二级结构变化
在分子动力学模拟中,二级结构分析用于研究和监测蛋白质或核酸等生物大分子中的二级结构变化情况。通过不同的颜色表示每个残基在动力学过程中形成的二级结构变化。
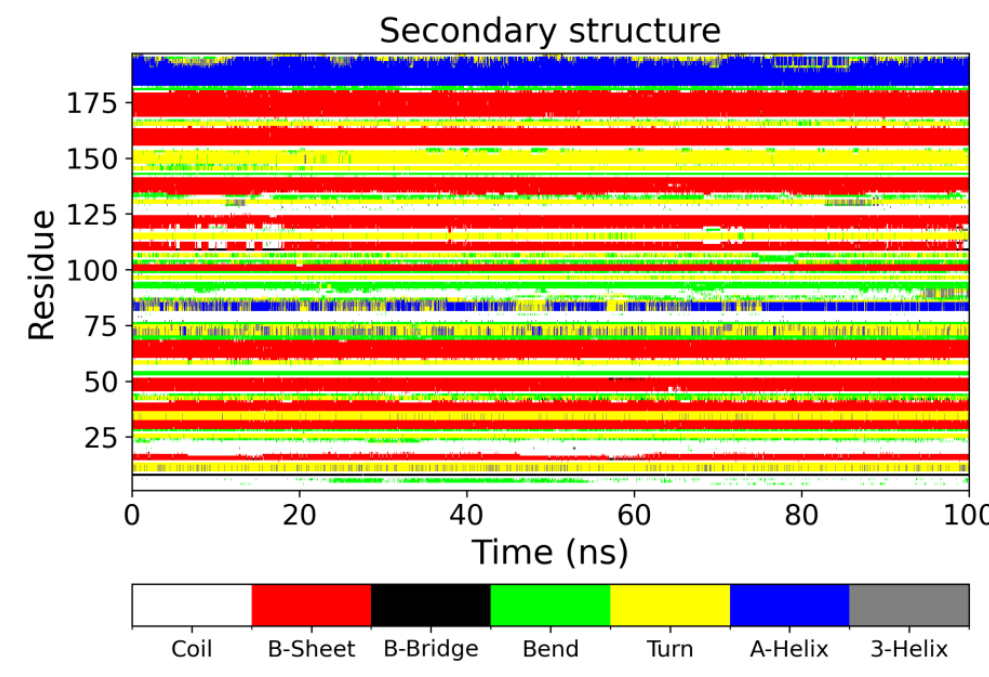
图10 蛋白二级结构变化
动力学项目类型分类
|
动力学模拟1 基础动力学模拟,常规的模拟体系+基础分析 |
|
动力学模拟2 含非标残基模拟,小分子化合物、PTM修饰基团等+基础分析 |
|
动力学模拟3 恒定pH模拟,不同pH梯度+基础分析 |
|
动力学模拟4 含膜模拟+基础分析 |
基础分析:RMSD、RMSF、Rg、SASA、Hbond、自由能计算、自由能地貌图
进阶分析1:FEP自由能微扰、SAPT能量分解
进阶分析2:指纹图谱、二级结构变化
6. 服务项目
分子动力学模拟技术服务:
根据客户提供的小分子与蛋白进行分子动力学模拟,分析蛋白配体复合物在100 ns中的动态变化,可以更深入地分析小分子与蛋白之间的相互作用,通过残基自由能分解可辅助验证关键氨基酸位点。
分子对接技术服务:
根据客户提供的小分子与蛋白进行对接,得到蛋白配体不同构象亲和力排序并作图,可同时进行SPR等体外实验验证。
虚拟筛选技术服务:
小分子化合物库与蛋白的对接,筛选得到化合物亲和力排序,可以挑选排名靠前的做SPR等体外分子互作技术高通量验证。
反向找靶技术服务:
根据客户提供的小分子化合物,在人、小鼠、大鼠靶点库中找到相匹配的蛋白靶点,给出蛋白打分列表,后期再做分子对接和亲和力检测实验验证。
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
- 作者
- 内容
- 询问日期
 文献和实验
文献和实验相关专题 分子生物学技术已经成为生命科学领域研究有力工具,但是很多实验的重复性和复杂性常会耗费很多时间,严重影响了实验的进度。所以现在很多课题组将实验中某些费事的实验环节外包给生物学技术服务 商。 1.哈尔滨海基生物科技有限公司 现代分子生物学技术已经成为科学研究领域有力的工具。但是,繁杂和重复性的工作花费了科研人员太多不必要的宝贵时间。将这些工作交给专业团队就能够大大节省实验人员的时间,提高
以蛋白分子为靶目标的噬菌体肽库模拟抗原的筛选 (1)将单克隆抗体 用包被液CBS稀释,加入酶标板,一般抗体包被浓度为10ug/孔,包被体积为100ul/孔,稍加晃动使孔表面变湿,置于4℃下包被过夜(或37℃慢摇孵育2h)。第二天倾去板中包被液,每孔加入200ul/孔洗板液(PBST05),静置5rain,甩去洗板液,于无菌干净的滤纸上拍打,以尽量除去剩余的液体,重复洗5次,以下的洗板操作都与此相同。加入封阻液(PBSTOS-1%BSA)200ul,于37℃
埃博拉病毒(EBOV)复制周期中的关键步骤会涉及到病毒糖蛋白 2(GP2)的构象改变,从而促进宿主 - 病毒膜融合,释放病毒基因组的过程。这个蛋白的作用和 HIV 的 GP41 的作用机制类似。这篇文章针对 EBOV 的 GP2 蛋白为靶点进行虚拟筛选,所选用的分子库包含大约 170 万个分子,使用传统的 DOCK 软件进行对接,从排名前列的化合物中,选出 165 种购买并进行生物活性检测,发现了 4 个良好的候选化合物,其 IC50 在 3 - 26uM 之间。在随后的分子动力学模拟中,发现
 技术资料
技术资料暂无技术资料 索取技术资料









