相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 保存条件:
-20℃
- 英文名:
NanoHema Panel v2.0
- 库存:
1
- 供应商:
纳昂达(南京)
NanoHema Panel v2.0 靶向 481 个血液肿瘤相关基因,全面覆盖碱基替换、插入/缺失、基因扩增及融合基因等多种变异类型。该 Panel 由 DNA 和 RNA 两部分组成:DNA 部分覆盖 1.7 Mb 基因组区域,包含 436 个基因的全部编码区、12 个基因的部分内含子区及 IGH 基因 Switch 区;RNA 部分覆盖 146 个基因,优化融合基因检测。NanoHema Panel v2.0 通过多组学整合、高靶向性设计及临床实用性优化,为血液肿瘤的精准诊断、治疗决策及全程管理提供了高效、经济的分子检测方案。
产品特色
● 多类型变异覆盖更全面
涵盖 481 个血液肿瘤相关基因,全面解析碱基替换、插入/缺失、扩增及融合基因等多种变异类型。
● 双流程协同结果更准确
RNA 流程有效弥补 DNA 流程的局限,不仅精准识别融合基因伴侣,还能验证融合事件的功能性影响,双流程多维互补保障检测结果的全面性和准确性。
● 个性化定制服务更灵活
支持 Panel 灵活拆分或增加目标基因,精准适配不同类型血液肿瘤的检测需求,满足科研探索或临床诊断的多元场景应用。
产品表现
NanoHema Panel v2.0_DNA——捕获表现
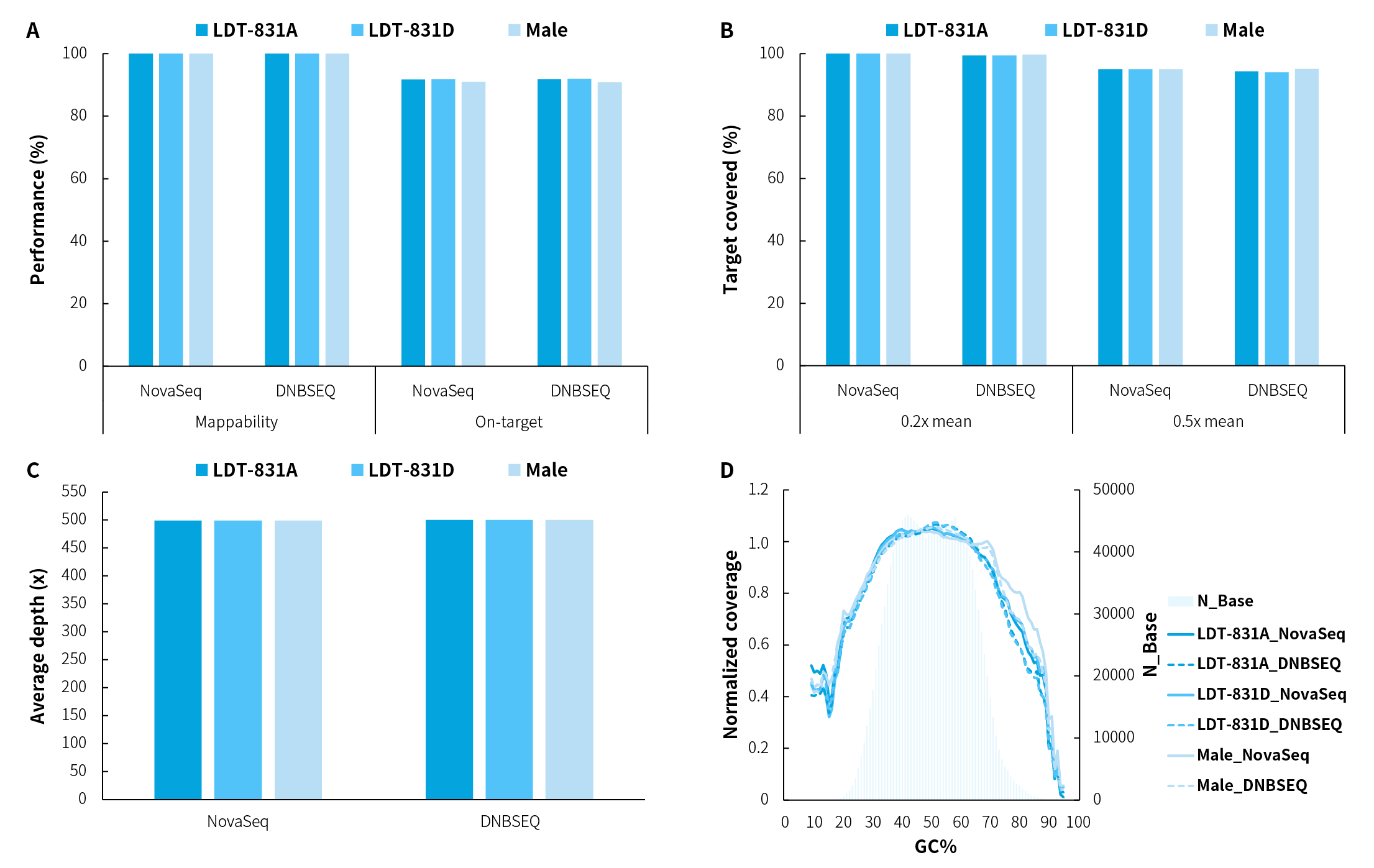
图 1. NanoHema Panel v2.0_DNA 对不同样本的捕获表现。A. 比对率&中靶率;B. 靶区域覆盖度;C. 平均测序深度 (去重后);D. GC 偏好性。50 ng 不同类型 gDNA 样本利用 NadPrep® 快速 DNA 酶切文库构建试剂盒搭配 NadPrep® Universal Stubby Adapter (UDI) Module 进行预文库构建,以 NanoHema Panel v2.0_DNA 搭配 NadPrep® Hybrid Capture Reagents 完成杂交捕获。测序模式分别为 Novaseq 6000,PE150 和 DNBSEQ-T7,PE150;随机选取 1.3 Gb 进行数据分析。
注:LDT-831 样本为血液肿瘤 gDNA 质控品 (LDT Bioscience,LDT-831A:AF = 5%;LDT-831D:AF = 0%);Male 样本为人类基因组 DNA 标准品 (Promega, G1471)。
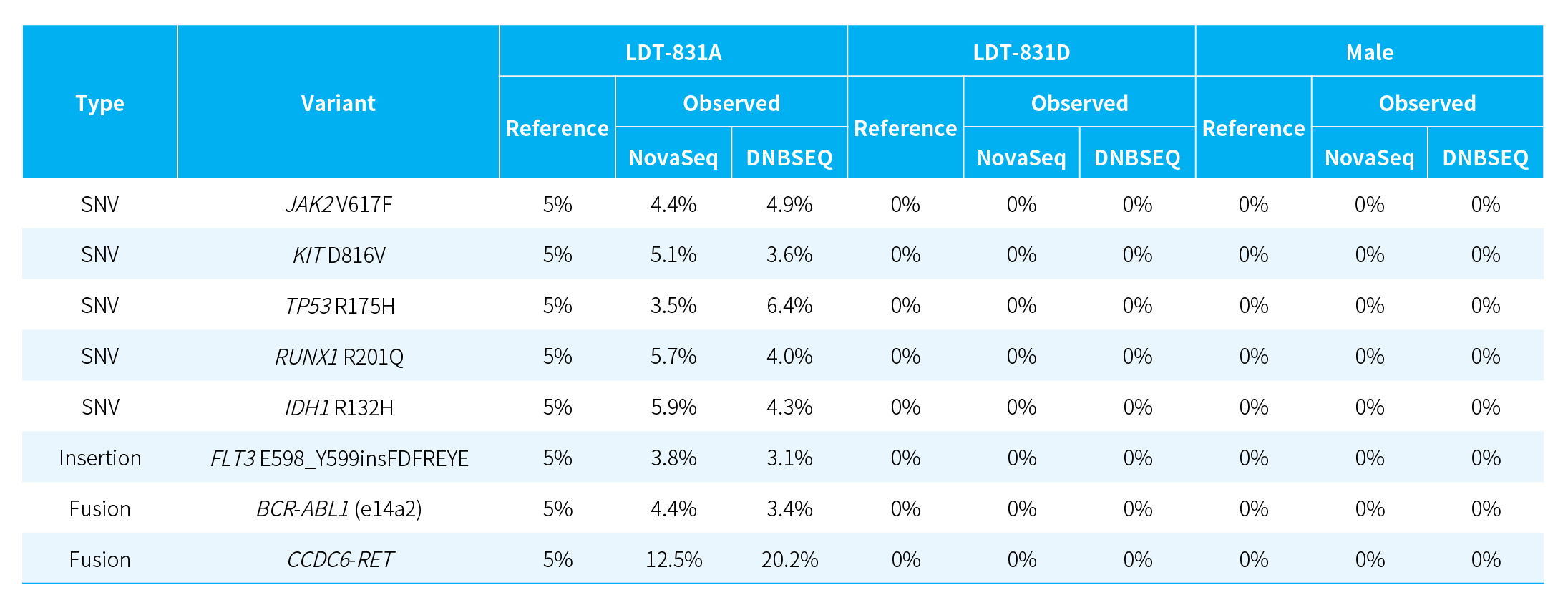
NanoHema Panel v2.0_DNA——多类型变异稳定检出
表 1. NanoHema Panel v2.0_DNA 对不同标准品中多类型变异的检出情况。
NanoHema Panel v2.0_RNA——捕获表现
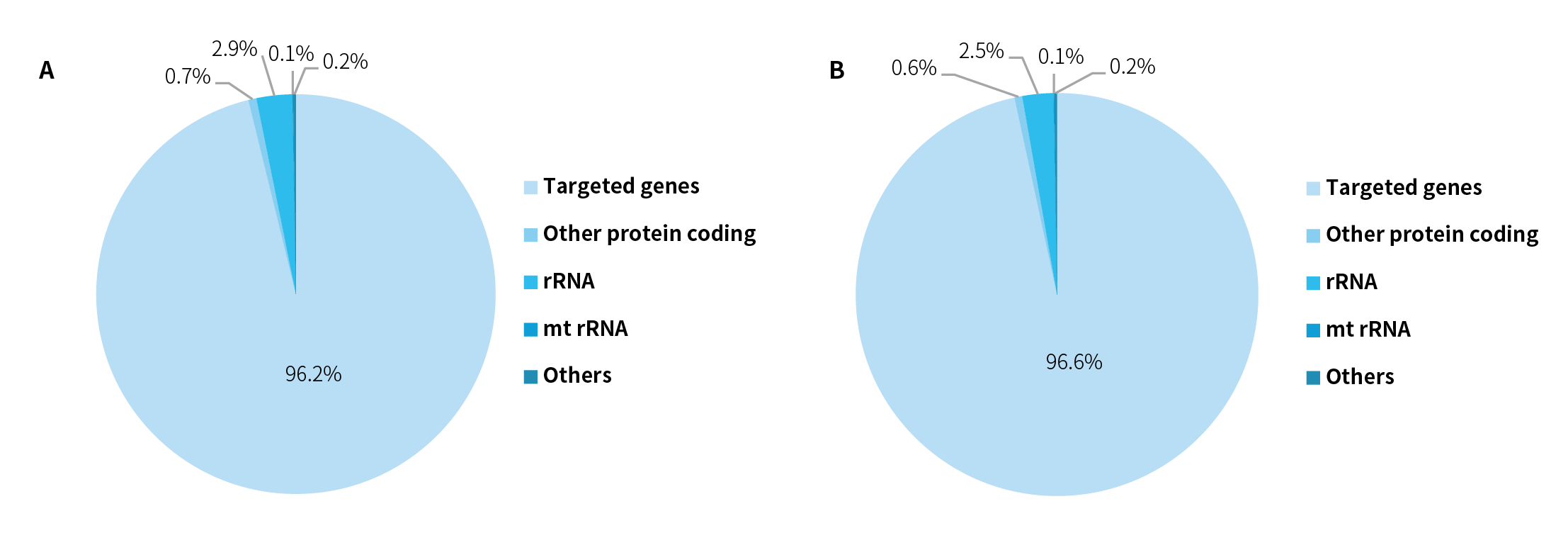
图 2. NanoHema Panel v2.0_RNA 捕获测序数据组成。50 ng K562 细胞系 RNA 以 NadPrep® Total RNA-To-DNA Module 搭配 NadPrep® DNA Library Preparation Kit v2 进行预文库构建,以 NanoHema Panel v2.0_RNA 搭配 NadPrep® Hybrid Capture Reagents 完成杂交捕获。测序模式分别为 Novaseq 6000,PE150 (A) 和 DNBSEQT7, PE150 (B)。以 Salmon 进行分析。
NanoHema Panel v2.0_RNA——融合基因检出情况
表 2. NanoHema Panel v2.0_RNA 对 K562 细胞系 RNA 中融合基因的检出。
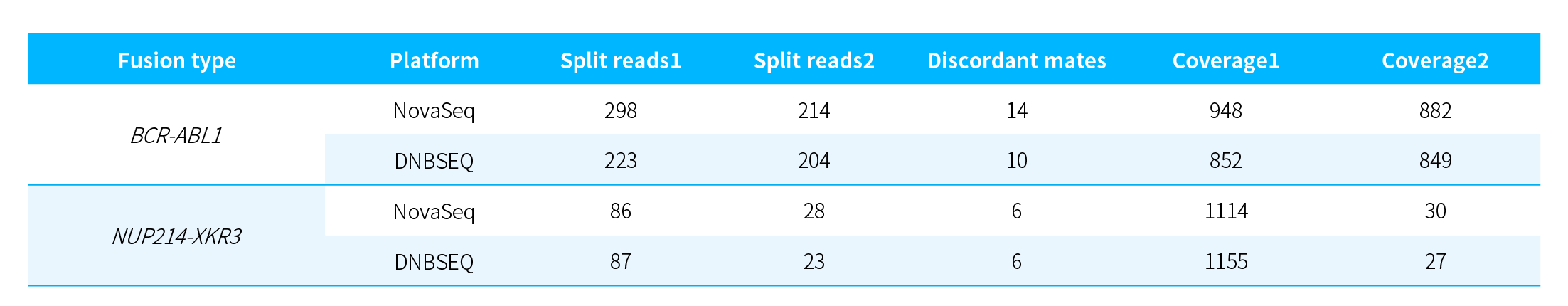
注:每百万 read pair。测序数据以 Arriba 进行分析。
基因列表
| NanoHema Panel v2.0_DNA_436 genes_Full CDS | ||||||||||||||
| ABL1 | ABL2 | ACTB | AKT1 | AKT2 | AKT3 | ALK | AMER1 | ANKRD26 | AP3B1 | APC | AR | ARAF | ARHGAP26 | ARID1A |
| ARID1B | ARID2 | ARID5B | ASXL1 | ASXL2 | ASXL3 | ATG5 | ATM | ATR | ATRX | ATXN1 | AURKA | AURKB | AXIN1 | AXL |
| B2M | BAALC | BAP1 | BARD1 | BCL10 | BCL11A | BCL11B | BCL2 | BCL6 | BCL7A | BCOR | BCORL1 | BIRC3 | BLM | BLNK |
| BRAF | BRCA1 | BRCA2 | BRD4 | BTG1 | BTG2 | BTK | BTLA | CALR | CARD11 | CBFB | CBL | CBLB | CCND1 | CCND2 |
| CCND3 | CCNE1 | CCR4 | CCR7 | CCT6B | CD22 | CD27 | CD274 | CD28 | CD58 | CD70 | CD79A | CD79B | CD83 | CDC42 |
| CDC73 | CDH1 | CDK12 | CDK4 | CDK6 | CDK8 | CDKN1A | CDKN1B | CDKN1C | CDKN2A | CDKN2B | CDKN2C | CEBPA | CEP57 | CHD2 |
| CHD8 | CHEK1 | CHEK2 | CIITA | CKS1B | CREBBP | CRLF2 | CSF1R | CSF3R | CTCF | CTLA4 | CTNNB1 | CUX1 | CXCR4 | CYLD |
| DAXX | DDR2 | DDX3X | DDX41 | DHX15 | DNM2 | DNMT3A | DOT1L | DTX1 | DUSP2 | DUSP22 | EBF1 | ECT2L | EED | EGFR |
| EP300 | EPCAM | EPHA2 | EPHA3 | EPHA5 | EPHA7 | EPOR | ERBB2 | ERBB3 | ERBB4 | ERCC4 | ERG | ESR1 | ETNK1 | ETS1 |
| ETV1 | ETV4 | ETV6 | EZH2 | FAM46C | FANCA | FANCC | FANCD2 | FANCE | FANCF | FANCG | FANCI | FANCJ | FANCL | FANCM |
| FAS | FAT1 | FAT4 | FBXO11 | FBXW7 | FGFR1 | FGFR2 | FGFR3 | FGFR4 | FH | FLCN | FLT1 | FLT3 | FLT4 | FOXO1 |
| FOXO3 | FOXP1 | FYN | GADD45B | GATA1 | GATA2 | GATA3 | GNA11 | GNA13 | GNAQ | GNAS | GNB1 | GRIN2A | HACE1 | HBA1 |
| HBA2 | HBB | HDAC1 | HDAC4 | HDAC7 | HGF | HIST1H1A | HIST1H1C | HIST1H1D | HIST1H1E | HNF1A | HRAS | ID3 | IDH1 | IDH2 |
| IFNAR2 | IGF1R | IGH | IGK | IGL | IGLL5 | IKBKE | IKZF1 | IKZF2 | IKZF3 | IL3 | IL7R | INPP4B | INPP5D | IRF1 |
| IRF4 | IRF8 | ITK | ITPKB | JAK1 | JAK2 | JAK3 | JARID2 | JUN | KAT6A | KDM2B | KDM5A | KDM5C | KDM6A | KDR |
| KIT | KLF2 | KLHL5 | KLHL6 | KMT2A | KMT2B | KMT2C | KMT2D | KRAS | LEF1 | LMO2 | LRP1B | LTB | LYN | LYST |
| MAF | MAFB | MALT1 | MAP2K1 | MAP2K2 | MAP2K4 | MAP3K1 | MAP3K14 | MAPK1 | MCL1 | MDM2 | MDM4 | MED12 | MEF2B | MEF2D |
| MEN1 | MET | MFHAS1 | MGA | MITF | MLH1 | MPL | MSH2 | MSH3 | MSH6 | MTOR | MUTYH | MYB | MYC | MYCL |
| MYCN | MYD88 | NBN | NCKAP1L | NCSTN | NF1 | NF2 | NFE2L2 | NFKB1 | NFKB2 | NFKBIA | NFKBIE | NKX2-1 | NLRC4 | NOTCH1 |
| NOTCH2 | NPM1 | NRAS | NSD1 | NT5C2 | NTRK1 | NTRK3 | NUP98 | P2RY8 | PAG1 | PAK3 | PALB2 | PAX5 | PBRM1 | PC |
| PDCD1 | PDCD1LG2 | PDGFRA | PDGFRB | PDK1 | PHF6 | PIK3CA | PIK3CD | PIK3R1 | PIK3R2 | PIM1 | PLCG1 | PLCG2 | PML | PMS2 |
| POLE | POT1 | POU2AF1 | PPM1D | PPP2R1A | PRDM1 | PRF1 | PRKCB | PRPF8 | PTCH1 | PTEN | PTPN1 | PTPN11 | PTPN2 | PTPN6 |
| PTPRD | PTPRK | PTPRO | RAB27A | RAD21 | RAD50 | RAD51 | RAF1 | RARA | RASGEF1A | RB1 | RECQL4 | REL | RELN | RET |
| RHOA | RICTOR | RNF43 | ROS1 | RPL10 | RPS15 | RPTOR | RUNX1 | SAMD9 | SAMD9L | SBDS | SDHA | SDHB | SDHC | SDHD |
| SETBP1 | SETD2 | SF3B1 | SGK1 | SH2B3 | SH2D1A | SLC19A1 | SLC7A7 | SMAD2 | SMAD4 | SMARCA2 | SMARCA4 | SMARCB1 | SMC1A | SMC3 |
| SMO | SOCS1 | SOX11 | SOX2 | SPEN | SPOP | SRC | SRP72 | SRSF2 | STAG2 | STAT3 | STAT4 | STAT5A | STAT5B | STAT6 |
| STK11 | STX11 | STXBP2 | SUFU | SUZ12 | SYK | TBL1XR1 | TCF3 | TCL1A | TERC | TERT | TET1 | TET2 | TET3 | TGFBR2 |
| TNFAIP3 | TNFRSF11A | TNFRSF14 | TNFRSF17 | TNFRSF1B | TOP1 | TOP2A | TP53 | TP63 | TP73 | TRAF2 | TRAF3 | TRAF5 | TSC1 | TSC2 |
| TSHR | TYK2 | U2AF1 | UNC13D | VAV1 | VHL | WHSC1 | WT1 | XIAP | XPC | XPO1 | ZAP70 | ZBTB7A | ZEB2 | ZMYM3 |
| ZRSR2 | ||||||||||||||
| NanoHema Panel v2.0_DNA_12 genes_Selected Intron | ||||||||||||||
| ALK | BCL6 | BCR | BRAF | ETV6 | JAK2 | KMT2A | MYC | PDGFRA | PDGFRB | RARA | RET | |||
| NanoHema Panel v2.0_RNA_146 genes | ||||||||||||||
| ABL1 | ABL2 | ALK | ARHGAP26 | ARID1A | ASXL1 | ATG5 | BCL10 | BCL11A | BCL11B | BCL2 | BCL3 | BCL6 | BCL7A | BCL9 |
| BCOR | BCR | BIRC3 | BRAF | BTG1 | CBFA2T3 | CBFB | CBL | CCND1 | CCND2 | CCND3 | CD274 | CDK6 | CEBPE | CIITA |
| CREBBP | CRLF2 | CTNNB1 | DEK | DUSP22 | DUX4 | EGFR | EP300 | EPOR | ERBB2 | ERG | ETS1 | ETV1 | ETV4 | ETV6 |
| EWSR1 | FGFR1 | FGFR2 | FGFR3 | FOXO1 | FOXO3 | FOXP1 | GLIS2 | HLF | IGH | IGK | IGL | IKZF1 | IL3 | IRF4 |
| ITK | JAK1 | JAK2 | JAK3 | KAT6A | KIF5B | KMT2A | LMO1 | LMO2 | LYN | MAF | MAFB | MALT1 | MECOM | MEF2D |
| MLLT1 | MLLT10 | MLLT3 | MN1 | MTCP1 | MYB | MYC | MYH11 | NCOA2 | NCOR1 | NF1 | NF2 | NFKB2 | NOTCH1 | NPM1 |
| NSD1 | NTRK1 | NTRK3 | NUP214 | NUP98 | NUTM1 | P2RY8 | PAX5 | PBX1 | PDCD1LG2 | PDGFRA | PDGFRB | PICALM | PIM1 | PML |
| POU2AF1 | PRDM1 | PTCH1 | RAF1 | RARA | RARB | RARG | RBM15 | RET | ROS1 | RUNX1 | RUNX1T1 | STAT6 | STIL | SYK |
| TAF15 | TAL1 | TBL1XR1 | TCF3 | TCL1A | TCL1B | TET1 | TLX1 | TLX3 | TNFRSF11A | TOP1 | TP63 | TPM3 | TPM4 | TRA |
| TRB | TRD | TRG | TYK2 | VAV1 | WHSC1 | ZBTB16 | ZEB2 | ZNF292 | ZNF362 | ZNF384 | ||||
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验V2R, β2AR, and β‐arrestin2 coding sequences 8‐arginine‐vasopressin (8‐AVP) Isoproterenol
Predicting Functional Effect of Human Missense Mutations Using PolyPhen‐2
?2 (Polymorphism Phenotyping v2), available as software and via a Web server, predicts the possible impact of amino acid substitutions on the stability and function of human proteins using structural and comparative evolutionary considerations. It performs
Sleep and EEG Phenotyping in Mice
Figure 7. EEG delta power during NREMS (upper panel) and distribution of NREMS during 48‐hr baseline, 6‐hr sleep deprivation (SD), and 18‐hr recovery (lower panel). Delta power increases during the dark










