相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 询价记录
- 文献和实验
- 技术资料
- 提供商:
江苏集萃药康生物科技股份有限公司
- 服务名称:
Rosa26-CAG-LSL-Cas9-tdTomato
Rosa26-CAG-LSL-Cas9-tdTomato 验证报告 I
-
构建方案
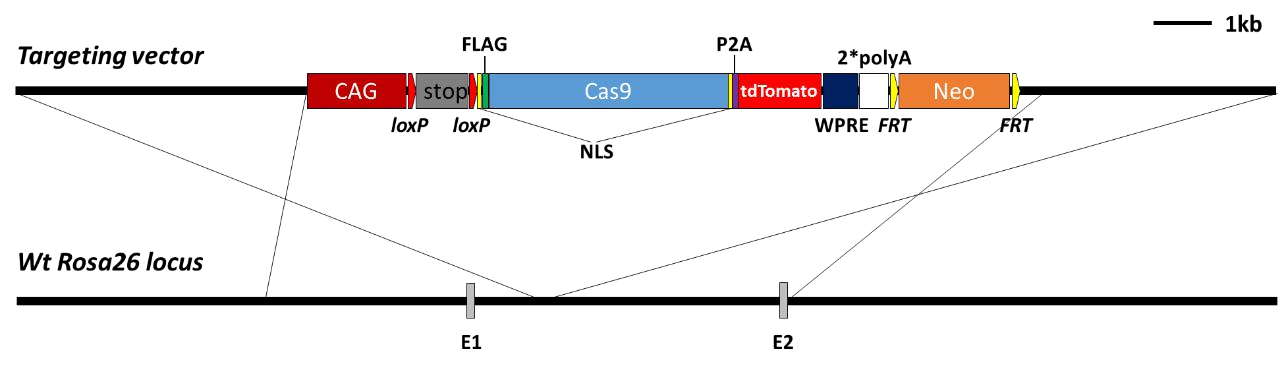
图一 Rosa26-LSL-Cas9-tdTomato 小鼠构建方案
小鼠的 Rosa26 基因座(Friedrich and Soriano, 1991)被证明能够稳定表达整合到此位点的外源基因,已被广泛地用于制作各种敲入及 Gene Trap 小鼠。我们将 CAG 启动子(Niwa et al., 1991) 驱动的 Cas9 表达元件插入到 Rosa26 位点,并在 CAG 与 Cas9 之间放置两侧带有 loxP 的 STOP 元件,以阻止 Cas9 表达,另外在 Cas9 后面还融合了红色荧光标记 tdTomato,以方便检测Cas9 表达情况。当存在 Cre 重组酶时,Cre/loxP 重组切除 STOP 后即可开启 Cas9 表达,并可以通过红色荧光检测到。
-
表达检测
为测试 Rosa26-LSL-Cas9-tdTomato 小鼠的 Cas9 表达情况及切割活性,我们分离了 MEF 细胞进行体外转染 Cre+sgRNA 测试。将基因型为 Rosa26-LSL-Cas9-tdTomato KI/flper 的 515#雌鼠及 397#雄鼠交配,取 E13.5 天胚胎分离 MEF 细胞,经鉴定确认分离得到的 MEF 细胞基因型为 KI/flper。
我们使用 Lonza Nucleofector™ 2b Device 进行 MEF 转染,转染使用的质粒为 pCAG-Cre:GFP 及U6-GPR50-S51,由于 Cre 与 GFP 融合,Cas9 与 tdTomato 融合,转染后通过观察绿色荧光和红色荧光即可获知 Cre 及 Cas9 的表达情况。转染后,收集细胞,提取基因组 DNA 进行 PCR 检测,通过 PCR 产物 T7E1 酶切实验可以获知 Cas9 对 sgRNA 靶点的切割情况。
转染实验分 5 组(TABLE 1),组 1A-1C 为 Cas9+sgRNA 共转染组,转染的 Cre 及 sgRNA 质粒总量分别为 2ug+4ug, 1ug+2ug 及 3ug+6ug;组 2 只转染 2ug Cre 质粒;组 3 只转染 4ug sgRNA 质粒。
TABLE 1 MEF 电转染实验分组及结果
| 实验分组 |
转染质粒 |
转染结果 |
|||
| pCAG-Cre-GFP |
U6-GPR50-S51 |
绿色荧光 |
红色荧光 |
DNA 切割 |
|
| 组 1A |
2ug |
4ug |
有 |
有 |
有 |
| 组 1B |
1ug |
2ug |
有 |
有 |
有 |
| 组 1C |
3ug |
6ug |
有 |
有 |
有 |
| 组 2 |
2ug |
0 |
有 |
有 |
无 |
| 组 3 |
0 |
4ug |
无 |
无 |
无 |
结果显示(图二~七),组 1A-1C 均能观察到绿色荧光和红色荧光,并且,T7E1 检测显示,Cas9
Page 2
对 sgRNA靶点进行了明显的切割;组2 能观察到绿色荧光和红色荧光,但未检测到Cas9 对 sgRNA
靶点切割;组3 未检测到绿色或红色荧光,也未检测到Cas9 对 sgRNA靶点的切割。
我们选择18h, 24h,48h 和66h 这4 个时间点观察荧光,发现绿色荧光在18h 最强,随着时间延长渐渐变弱,到66h 已经几乎观察不到;而红色荧光在则在18h 不可见,24h才开始出现,48h最强,66h开始变弱,绿色和红色荧光的变化与Cre 及Cas9 的表达顺序一致,当质粒通过转染进入细胞后,Cre被表达出来即可观察到绿色荧光,Cre切除rosa26 位置的loxP-STOP-loxP,开启Cas9的表达,才可以看到红色荧光,因此,红色荧光出现的时间应当比绿色荧光出现时间晚。T7E1切割检测显示,组2 及组3 都未发生Cas9 切割,证明在缺少Cre 或sgRNA 时,均不会发生Cas9切割。表达检测结果与BROAD 研究所的张锋博士实验室制作的Rosa26-LSL-Cas9-EGFP 小鼠一致。
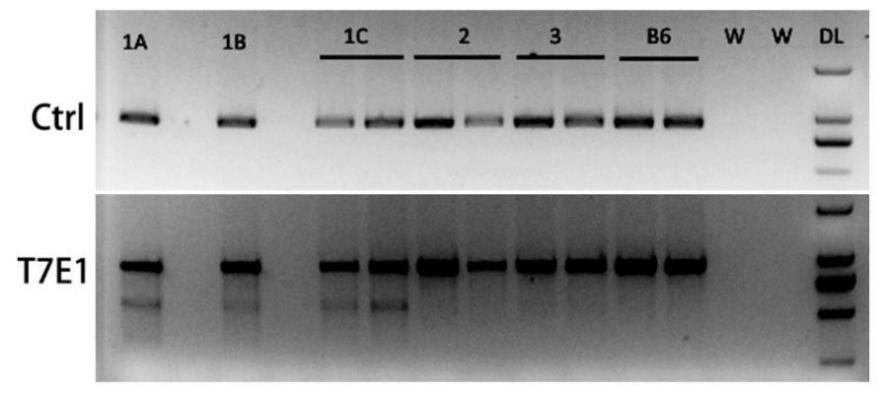
图二 T7E1酶切鉴定
T7E1 酶切鉴定结果显示,共转染了Cre和sgRNA质粒的1A1B 1C三组DNA都发生了Cas9对sgRNA靶点的切割,而单独转染Cre 质粒(组2)或单独转染sgRNA 质粒(组3)的两组与B6(阴性对照)一样,未检测到Cas9 切割。
B6: C57BL/6J genomic DNA, W: blank control, DL: DL2000 DNA Marker
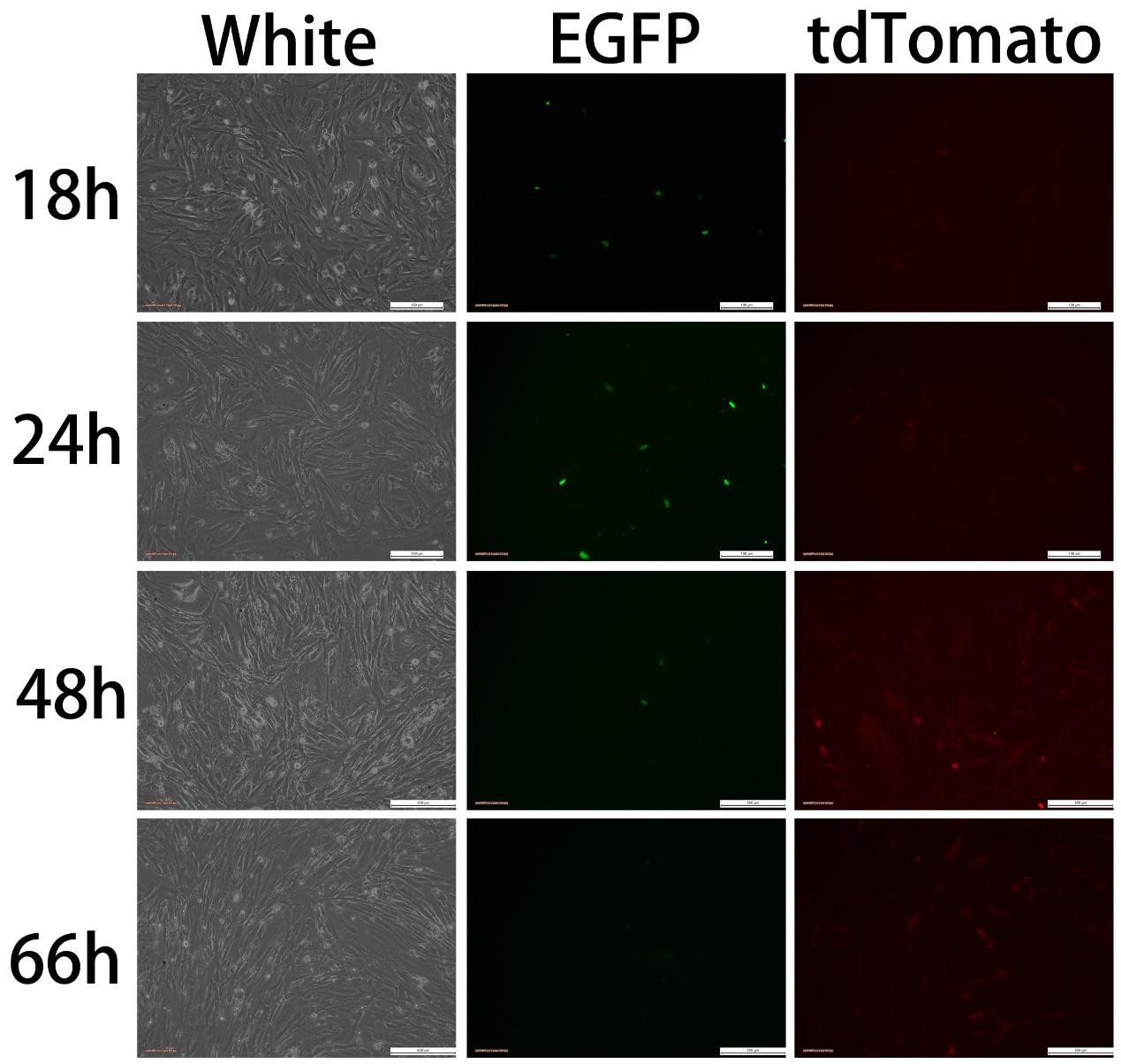
图三 组2 Transfectedwith pCAG-Cre:GFP
转染质粒为pCAG-Cre:GFP(2ug),Cre-GFP表达后可观察到绿色荧光,而Cas9-tdTomato则需要Cre/loxP反应切除STOP元件后才能开启表达,出现红色荧光,因此,红色荧光出现的时间应当比绿色荧光出现的时间要晚。实验结果与预期相符,绿色荧光18h 即出现,随着时间推移,绿色荧光渐渐消失,至66h 已不可见;24h 开始出现红色荧光,48h荧光增强,66h红色荧光减弱。
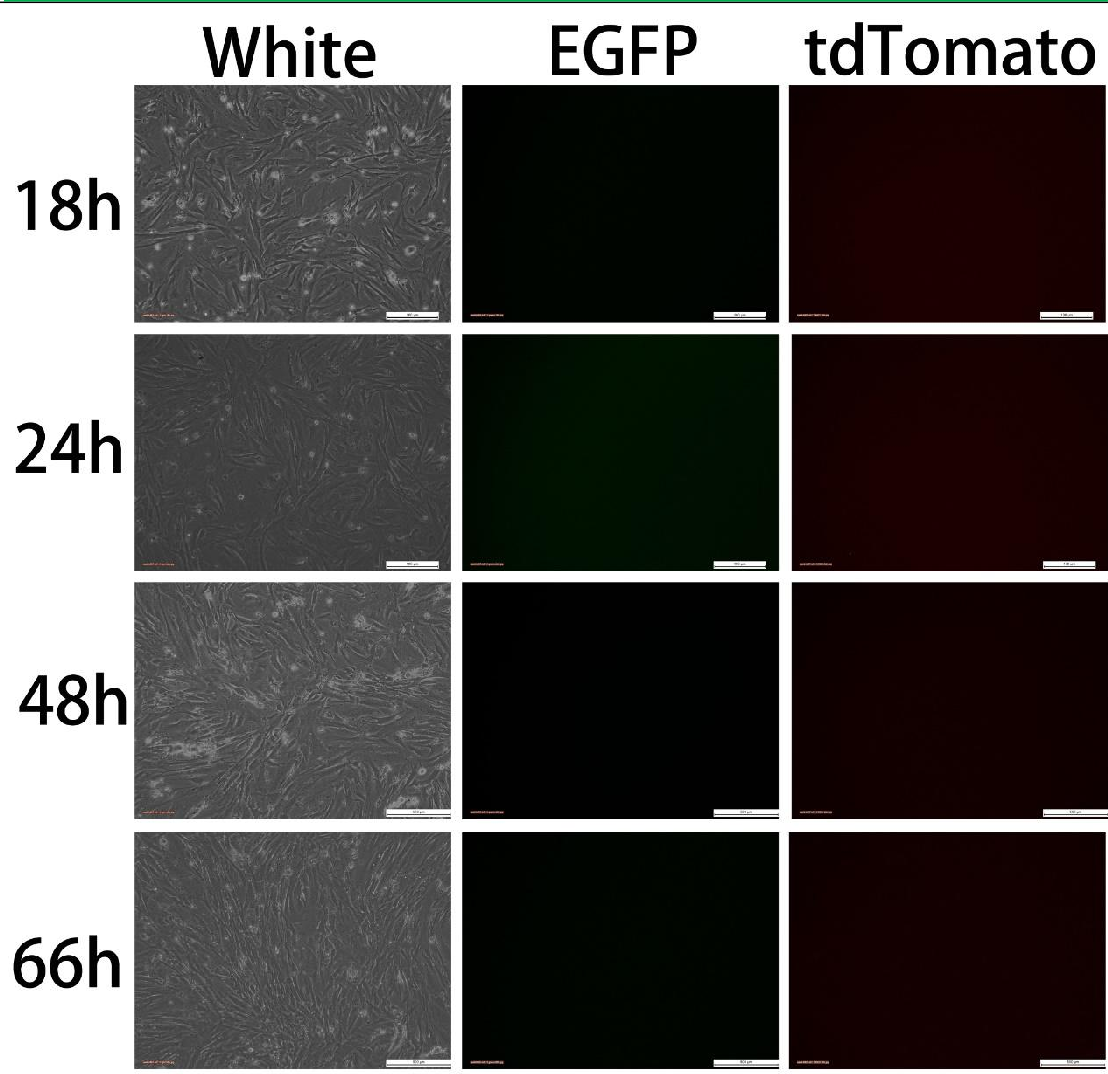
图四 组3 Transfectedwith U6-GPR50-S51
转染质粒U6-GPR50-S51(4ug),由于无Cre表达,观察不到绿色荧光,不能切除loxP-STOP-loxP,无法开启Cas9 表达,观察不到红色荧光。实验结果与预期相符,未观察到荧光。
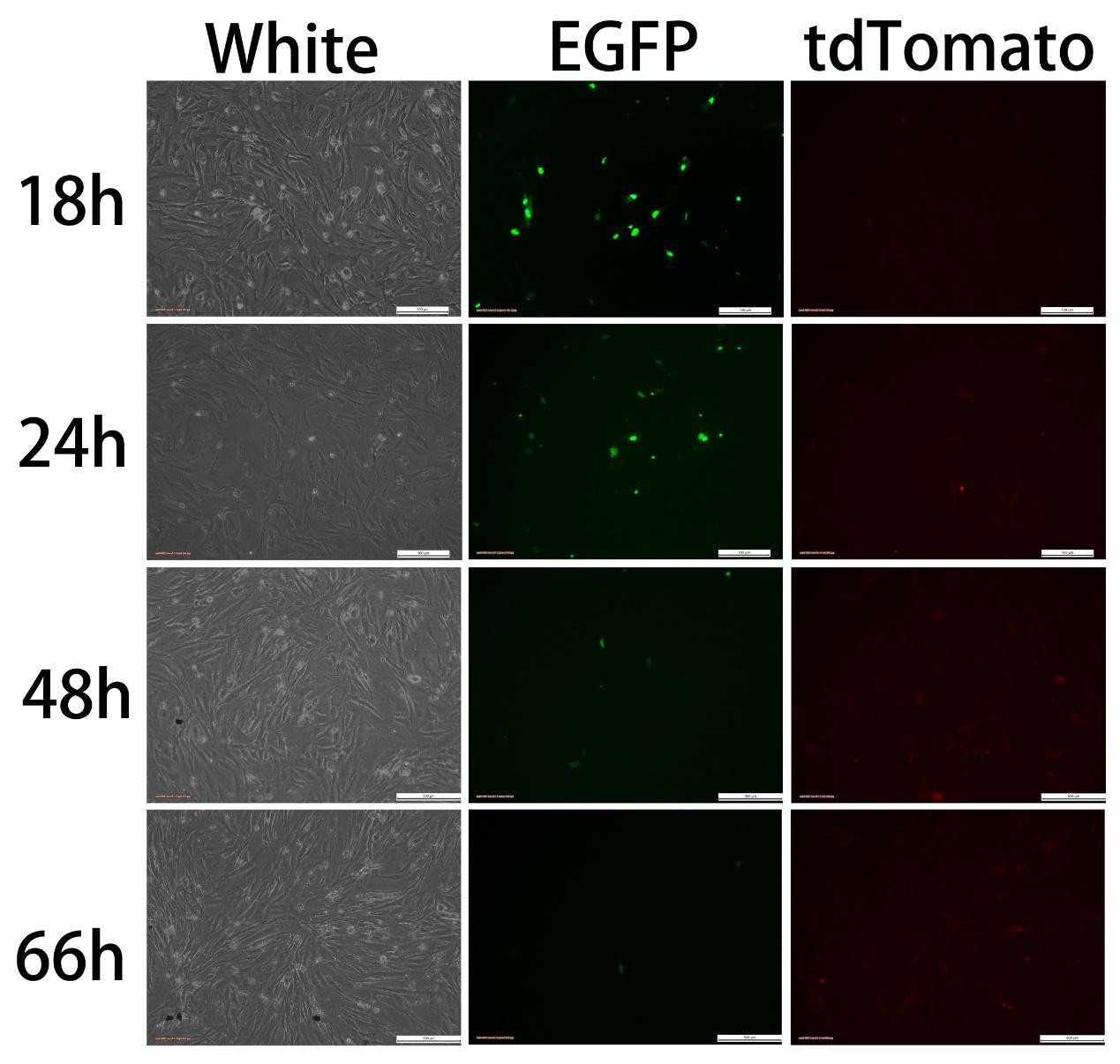
图五 组1A Transfectedwith pCAG-Cre:GFP(2ug)+U6-GPR50-S51(4ug)
转染质粒为pCAG-Cre:GFP(2ug)+U6-GPR50-S51(4ug),Cre-GFP表达后可观察到绿色荧光,而Cas9-tdTomato则需要Cre/loxP反应切除STOP元件后才能开启表达,出现红色荧光,因此,红色荧光出现的时间应当比绿色荧光出现的时间要晚。实验结果与预期相符,绿色荧光18h 即出现,随着时间推移,绿色荧光渐渐消失,至66h 已不可见;24h开始出现红色荧光,48h荧光增强, 66h红色荧光减弱。

图六 组1B Transfectedwith pCAG-Cre:GFP(1ug)+U6-GPR50-S51(2ug)
转染质粒为pCAG-Cre:GFP(1ug)+U6-GPR50-S51(2ug),Cre-GFP表达后可观察到绿色荧光,而Cas9-tdTomato则需要Cre/loxP反应切除STOP元件后才能开启表达,出现红色荧光,因此,红色荧光出现的时间应当比绿色荧光出现的时间要晚。实验结果与预期相符,绿色荧光18h 即出现,随着时间推移,绿色荧光渐渐消失,至66h 已不可见;24h开始出现红色荧光,48h荧光增强, 66h红色荧光减弱。
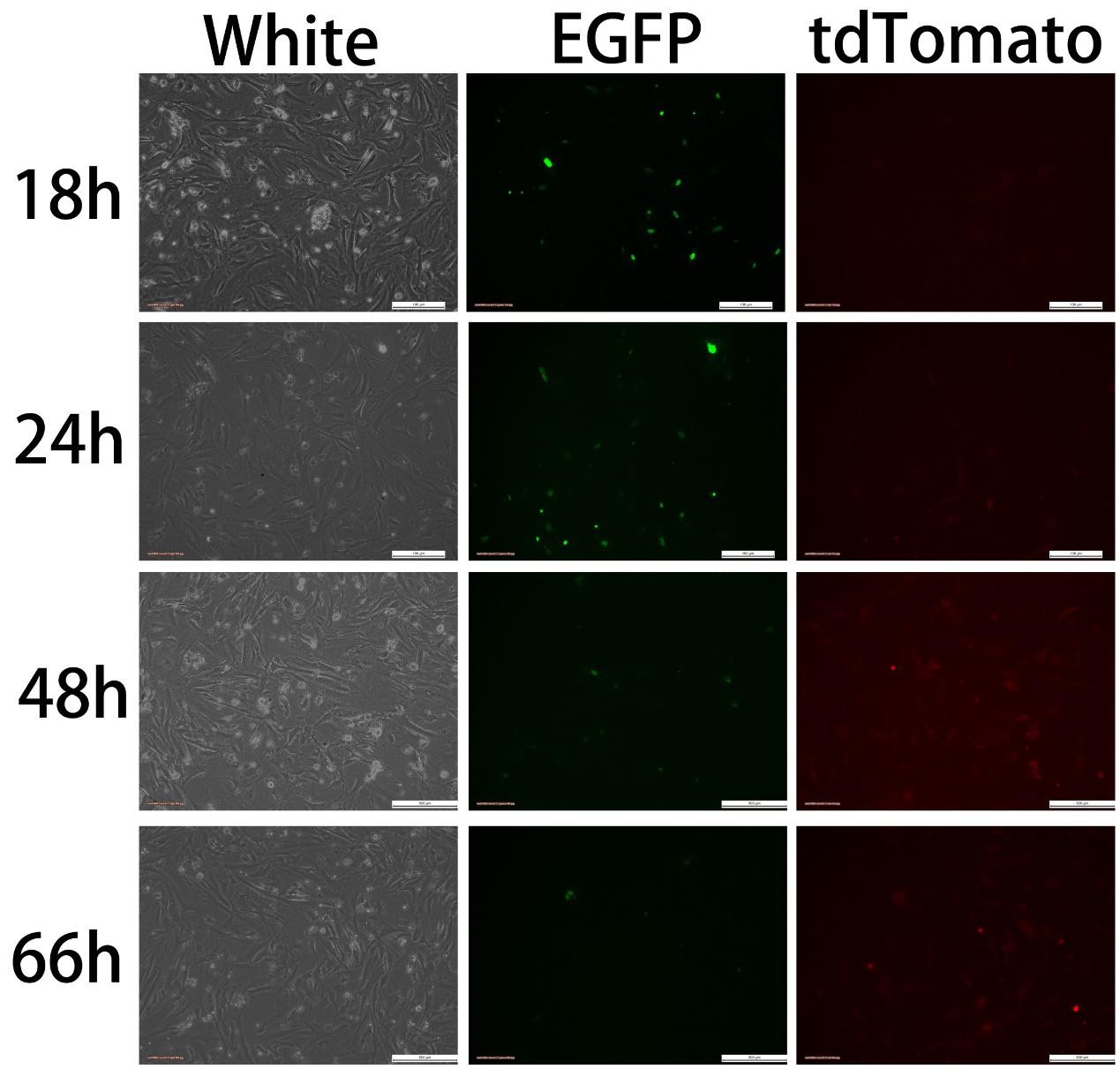
图七 组1C Transfectedwith pCAG-Cre:GFP(3ug)+U6-GPR50-S51(6ug)
转染质粒为pCAG-Cre:GFP(3ug)+U6-GPR50-S51(6ug),Cre-GFP表达后可观察到绿色荧光,而Cas9-tdTomato则需要Cre/loxP反应切除STOP元件后才能开启表达,出现红色荧光,因此,红色荧光出现的时间应当比绿色荧光出现的时间要晚。实验结果与预期相符,绿色荧光18h 即出现,随着时间推移,绿色荧光渐渐消失,至66h 已不可见;24h开始出现红色荧光,48h荧光增强, 66h红色荧光减弱。
-
结论
分离rosa26-LSL-Cas9-tdTomato小鼠的MEF 细胞在体外转染Cre 表达质粒,该模型可在Cre/loxP切除STOP 元件后开启Cas9 及tdTomato表达;在共转染Cre 表达质粒及guide RNA表达质粒时,可同观察到红色荧光,并检测到靶基因的切割。根据实验结果,可判定该模型制备成功。但是,由于转染效率较低,本次实验观察到的荧光信号较弱。
参考文献
Cong, L., Ran, F.A., Cox,D., Lin, S., Barretto, R., Habib, N., Hsu, P.D.,Wu, X., Jiang, W., Marraffini, L.A., et al. (2013). Multiplex genomeengineering using CRISPR/Cas systems. Science 339,819-823.
Flotte, T.R. (2000). Sizedoes matter: overcoming the adeno-associated virus packaging limit. Respir Res 1, 16-18.
Friedrich, G., and Soriano, P. (1991). Promoter traps in embryonicstem cells: a genetic screen to identify and mutate developmentalgenes in mice. Genes Dev 5, 1513-1523.
Jinek, M., Chylinski, K., Fonfara, I., Hauer,M., Doudna, J.A., and Charpentier, E. (2012). A programmabledual-RNA-guided DNA endonuclease in adaptive bacterial immunity.Science 337, 816-821.
Kumar, M., Keller,B., Makalou, N., and Sutton, R.E. (2001). Systematicdetermination of the packaging limit of lentiviral vectors. Hum GeneTher 12, 1893-1905.
Liu, F., Song, Y.,and Liu, D. (1999). Hydrodynamics-based transfection inanimals by systemic administration of plasmid DNA. Gene Ther 6,1258-1266.
Mali, P., Yang, L., Esvelt, K.M., Aach, J., Guell, M., DiCarlo, J.E.,Norville, J.E., and Church, G.M. (2013). RNA-guided human genomeengineering via Cas9. Science 339, 823-826.
Nishikawa, M., Yamauchi, M., Morimoto, K., Ishida, E., Takakura, Y., and Hashida, M. (2000). Hepatocyte-targeted in vivo gene expressionby intravenous injection of plasmid DNA complexed with syntheticmulti-functional gene delivery system. Gene Ther 7, 548-555.
Niwa, H., Yamamura, K., and Miyazaki, J. (1991). Efficient selectionfor high-expression transfectants with a novel eukaryotic vector.Gene 108, 193-199.
Platt, R.J., Chen, S., Zhou, Y., Yim, M.J., Swiech, L., Kempton,H.R., Dahlman, J.E., Parnas, O., Eisenhaure, T.M., Jovanovic, M.,et al. (2014). CRISPR-Cas9 knockin mice for genome editing andcancer modeling. Cell 159, 440-455.
Song, Y.K., Liu, F.,Zhang, G., and Liu, D. (2002). Hydrodynamics-basedtransfection: simple and efficient method for introducing andexpressing transgenes in animals by intravenous injection of DNA.Methods Enzymol 346,92-105.
Wells, D.J. (1993). Improved gene transfer by direct plasmidinjection associated with regeneration in mouse skeletal muscle. FEBSLett 332, 179-182.
Wu, Z., Yang, H., and Colosi, P. (2010). Effect of genome size on AAVvector packaging. Mol Ther 18, 80-86.
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
- 作者
- 内容
- 询问日期
 文献和实验
文献和实验在最新出版(2016.02)的冷泉港实验指南的头版,详细阐述了 CRISPR-Cas9 系统在小鼠基因编辑中应用。 在现代生物学中,实现对小鼠基因进行编辑或许是最重要的进步之一。然而,传统基因工程手段都很费时费力。可编程核酸酶的使用极大的提高了基因编辑技术的准确性(如,锌指核酸酶、转录激活效应核酸酶),但是这些核酸酶的设计和使用仍然纷繁复杂成本高昂。随着下一代可编程核酸酶系列--CRISPR-Cas9 系统的出现,使得基因编辑的能力更加高效准确,与此同时,该系统所涉及的试剂
我们大多习惯把所有的基因编辑小鼠都称为「转基因小鼠」,其实细品下便知这是一种错误的说法。来来来,师兄帮你们科普下! 一、基因编辑小鼠的分类 基因编辑小鼠可分为三大类、7 小类: (1)转基因(Tg)首先构建表达载体,包含启动子、增强子、编码区、polyA 等元件,然后将表达载体注入到受精卵的细胞核内,外源表达载体将插入到受精卵的基因组内,再将该受精卵移植到受体母鼠输卵管内,发育成为小鼠个体,该小鼠就会表达转入的外源基因。 (2)基因全身性敲除(KO)利用 Crispr/Cas9
基因编辑再次升级!领域大牛刘如谦 Cell 发文开发新工具,可安全高效进行体内基因编辑
4 BE-eVLPs 支持有效、高效的基因编辑,并且显示出最小的脱靶编辑效率和 DNA 整合风险。 图片来源:Cell v4 BE-eVLPs 有效地编辑人类原代细胞和小鼠细胞 为了进一步探索 v4 BE-eVLPs 的效用,他们评估了其在体外靶向和编辑多种原代人类或小鼠细胞的能力。在转导含有纯合子 COL7A1(R185X)突变的原代人类成纤维细胞中,他们观察到高于 95% 的修复效率。这种高效率在小鼠模型的原代成纤维细胞中也得到了重现。 这些结果验证了 v4 BE-eVLPs 的活性并不局限于细胞
 技术资料
技术资料暂无技术资料 索取技术资料









