【干货】多种循环肿瘤DNA 突变检测方法详细解读
2024-07-05 07:53点击次数:1537
关键词:ctDNAARMS-PCR,液体活检,迈迪安NGS建库分子酶循环肿瘤DNA(Circulating Tumor DNA,ctDNA)含有肿瘤的遗传信息,与肿瘤组织具有高度的一致性,可代替肿瘤组织用于肿瘤的早期诊断,预后监测和药物疗效监测,是一种具有良好临床应用前景的液体活检标记物。
但血液中的ctDNA 片段化程度高,含量稀少,且与野生型DNA 混合存在(约1%甚至更低),并会随着患者肿瘤分期等情况动态变化,造成了ctDNA 检测困难,需要高灵敏度高特异性的突变检测方法,才能够从大量的野生型DNA 中检测出微量突变型ctDNA。
目前,灵敏度和特异性能够满足ctDNA 检测需求的方法主要有:扩增阻滞突变系统PCR(Amplification Refractory Mutation System PCR,ARMS-PCR)、钳制PCR(Clamping-PCR)、数字化PCR(Digital-PCR)和高通量测序技术。
本文主要对这些方法的原理、特点、进展和应用前景进行了综述,为研究人员选择合适的ctDNA 检测方法提供理论依据。

当肿瘤发生发展到一定阶段,肿瘤细胞会通过凋亡、坏死和分泌等途径将肿瘤特异的DNA 释放入血液,形成循环肿瘤DNA。血液中的ctDNA 携带有肿瘤特异性的单核苷酸变异、拷贝数变异、插入和缺失突变、序列的结构变异等遗传学改变,与肿瘤组织具有高度的一致性。
大量报道显示,ctDNA 可代替肿瘤组织用于肿瘤的早期诊断,预后监测和药物疗效监测。与难获取且存在异质性的肿瘤组织相比,ctDNA来自外周血,易获得,更均一,更能反映肿瘤的发生发展的情况。此外,ctDNA 还可通过多次采取外周血的途径获得,可以实现对疾病或药物疗效的实时监测。因此,基于ctDNA 的液体活检方式已逐渐成为肿瘤研究领域的新热点。
尽管目前针对ctDNA 的研究报道越来越多,但要将ctDNA作为分子标记物应用于临床仍然存在很多技术上的难题。
这主要是由于,与正常体细胞DNA 相比,ctDNA 具有以下独特的生物学特性:
① 含量低:每毫升血浆样本中仅含有几十拷贝的ctDNA,且不同癌症及个体间ctDNA 含量存在显著差异
② 与大量野生型DNA 共存:血液中突变的ctDNA 拷贝数仅为野生型DNA 拷贝数的1%甚至更低,仅一个碱基差异的野生型DNA 会严重干扰突变型ctDNA 的检测
③ 片段化程度高:长度主要集中在166bp 左右
④ 半衰期短:一般为2h
⑤ 处于一个动态的实时变化状态:ctDNA 含量会随着患者的肿瘤分期,手术和化疗等情况动态变化
这些特点造成了ctDNA 检测比体细胞检测更加困难,需要具有高灵敏度和特异性的突变检测方法,才能够从大量的野生型DNA 中扩增并检测出微量的突变型ctDNA。
临床常用的直接测序法(包括Sanger 测序法和焦磷酸测序法),通常只能检测5%~20 %的基因突变,难以满足血浆ctDNA 基因突变检测的需求。
目前,灵敏度和特异性能够满足ctDNA 检测需求的方法主要有ARMS-PCR、钳制PCR(Clamping-PCR)、数字化PCR和高通量测序技术。
- ARMS-PCR
等位基因特异性PCR(Allele Specific PCR,AS-PCR),又称ARMS-PCR,是Newton等首先建立起来用于检测已知突变的方法。
其基本原理为:DNA 聚合酶缺少3'→5' 外切酶活性,因此PCR 引物的3' 端末位碱基必须与其模板DNA 互补才能有效扩增。根据DNA 聚合酶的此种特性,研究者可以针对不同类型的已知突变设计特异性的引物来实现突变检测。
该法在设计引物时,针对ctDNA 分别设计突变型上游引物(MP-F),通用型下游引物(Primer-R)和荧光探针(Probe),在合适的反应条件下,MP-F只能与ctDNA互补发生扩增产生荧光信号,而与野生型DNA 3' 端存在错配,无法扩增产生荧光信号(如图1)。
但在实际ARMS-PCR 过程中,即使引物3' 端碱基存在错配,突变型特异的PCR 引物也可能与野生型DNA 发生延伸,进而产生假阳性的结果。
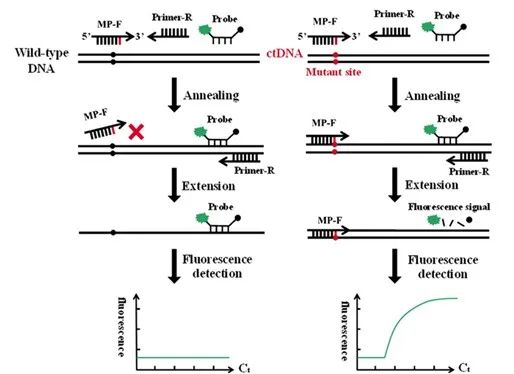
图1 ARMS-PCR 法检测ctDNA 原理图
为解决这一问题,对此方法进行了改进:即在引物3' 端附近掺入一些错配的碱基,使得突变型PCR 引物与野生型DNA 模板的结合力进一步减弱,引物错配的发生率大大降低,进而降低假阳性的发生率。此法敏感性可达到1%。
为了进一步提高检测灵敏度,在ARMS-PCR 基础上,研究者们引用了一种新型探针即蝎形(Scorpions)探针,构成蝎形探针扩增阻滞突变系统(Scorpions ARMS,SARMS)。
蝎形探针的特点是带有一段特异性引物,可与相应的靶核酸结合,进行扩增反应。而蝎形探针的尾能和此扩增子相互杂交,使得淬灭基团与荧光基团分开,产生荧光。因此其相互杂交是一种分子内的相互作用,与Taqman 探针不同,它并不依赖于Taq 和外切酶活性。SARMS的优点是反应迅速,产生的荧光信号更强,且具有更好的特异性,其敏感性可达0.1%。
ARMS-PCR 技术操作简单,仪器要求低,灵敏度较直接测序法有较大的提高。但ARMS-PCR目前仍存在一些缺陷。
首先,ARMS-PCR 只能检测已知突变,对未知突变难以检测;其次,虽然有了各种改进办法,但ARMS-PCR 假阳性率仍然较高,这便限制了其突变检测灵敏度的进一步提高,难以实现对低于0.1 %突变的检测。
此外,ARMS-PCR 在使用前期需要对反应条件进行严格的优化,包括ARMS 引物的筛选,引物、探针、Mg2+ 和Taq 酶的最佳浓度的选取,退火温度的摸索等等。另外临床肿瘤患者血浆样本成分复杂,极易影响ARMS-PCR扩增效率从而对实验结果造成偏差。
- Clamping-PCR
钳制聚合酶链式反应(Clamping-PCR) 是1993 年由意大利学者Henrik Φ rum 等首次提出的一种新型的突变检测技术。
Clamping-PCR根据探针类别主要包括三种:
1)肽核酸( Polyamide Nucleic Acid,PNA)介导的Clamping-PCR 技术
2)锁核酸(Locked Nucleic Acid,LNA)介导的Clamping-PCR 技术
3)反向引物介导的Clamping-PCR技术
以PNA介导的Clamping-PCR技术为例,PNA是一类合成的寡核苷酸类似物,它能够与互补的单链核酸杂交但不能被DNA 聚合酶识别而无法进行扩增;另外,PNA/ DNA结合物对碱基错配敏感,一个碱基的错配可使Tm值降低8℃~20℃(平均15℃)。
基于PNA的特点,设计ctDNA突变检测思路如图2所示:针对ctDNA设计一条上游引物(MP-F)、一条通用的下游引物(Primer-R)、一条荧光探针(Probe) 和一条能与野生型DNA 互补的PNA 探针(PNA-F),在严格的反应条件下,PNA 探针与野生型DNA高亲和性结合,进而阻止突变型引物与野生型DNA结合。
由于PNA探针无法作为引物延伸,则野生型DNA扩增被阻止。另外,PNA探针与ctDNA之间存在碱基错配,Tm值大幅降低,PNA探针无法与ctDNA结合,对ctDNA扩增不会造成影响。最后,通过荧光信号的有无就能检测出ctDNA的有无。
但在实验操作过程中,因为背景荧光等影响因素,限制了其灵敏度的进一步提高。为此,很多研究者对PNA-PCR方法进行改进,例如:利用核酸类似物LNA 做荧光探针,进一步降低荧光背景,提高检测灵敏度和特异性。
这种PNA-LNA qPCR方法常被用来检测ctDNA中B-RAF、EGFR、Kras 等基因片段的突变情况。
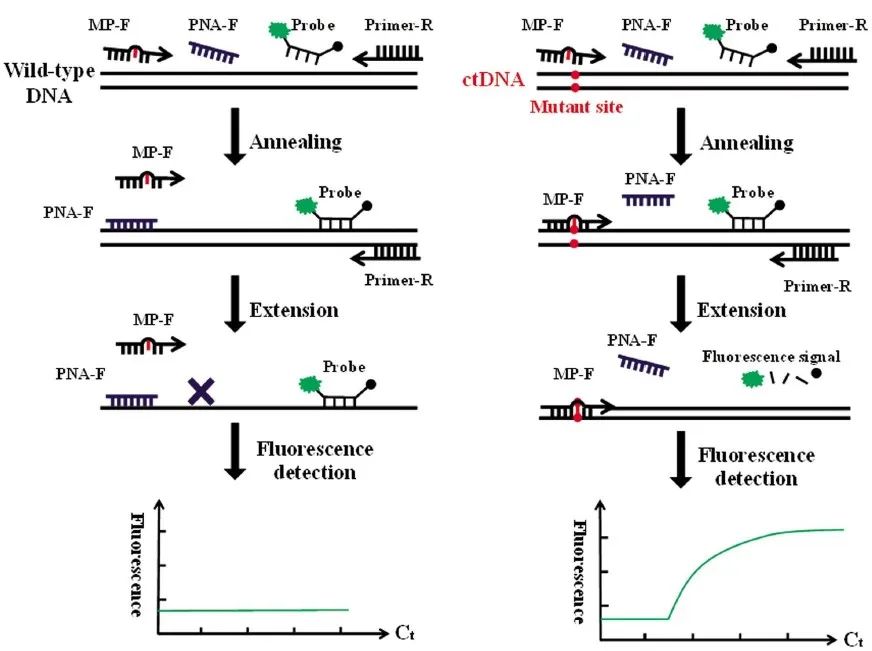
图2 PNA介导的Clamping-PCR 法检测ctDNA 原理图
钳制PCR的灵敏度可达到0.1 % ~1 % , 与普通的ARMS-PCR相比,有一定程度的提高,并且操作简便,对仪器也无特殊要求。
虽然Clamping-PCR检测ctDNA的突变具有诸多优势,但仍存在一些不足。首先,这种方法前期PNA探针和普通PCR引物设计、验证过程比较复杂,既要保证PNA探针不影响突变性样本扩增,又要保证野生型样本扩增被钳制,只有对Tm值的合理设置,才能实现高灵敏度、高特异性的要求;其次,PNA、LNA探针价格昂贵,国内应用较少。
- 数字PCR
数字PCR(Digital-PCR,dPCR) 是近几年迅速发展起来的一种新兴技术,基于单分子PCR方法来进行计数的一种核酸绝对定量方法。
目前,dPCR主要分为微滴式(Droplet dPCR,ddPCR)和芯片式(Chip dPCR,cdPCR)两种:ddPCR 主要以Biorad 公司的QX200 系统以及Raindance Technologies 公司的Raindrop 系统为代表;Cd-PCR 主要以Fluidigm 公司的BioMark HD 系统以及Life Technology公司的Quant Studio 3D 系统为代表。
目前ddPCR 应用较为广泛,其原理是将含有核酸分子的反应体系分割成数万个(甚至几十万个)纳升级的油包水型微滴反应器,核酸分子随机地进入微滴反应器中。经有限的PCR循环扩增,有模板的微滴反应器发出荧光,无模板的不发荧光。然后对每个微滴逐个进行荧光检测,有荧光信号的微滴反应器判读为1,没有荧光信号的微滴反应器判读为0,最后根据泊松分布的原理和阳性微滴的个数与比例即可得出靶分子的起始拷贝数或浓度。
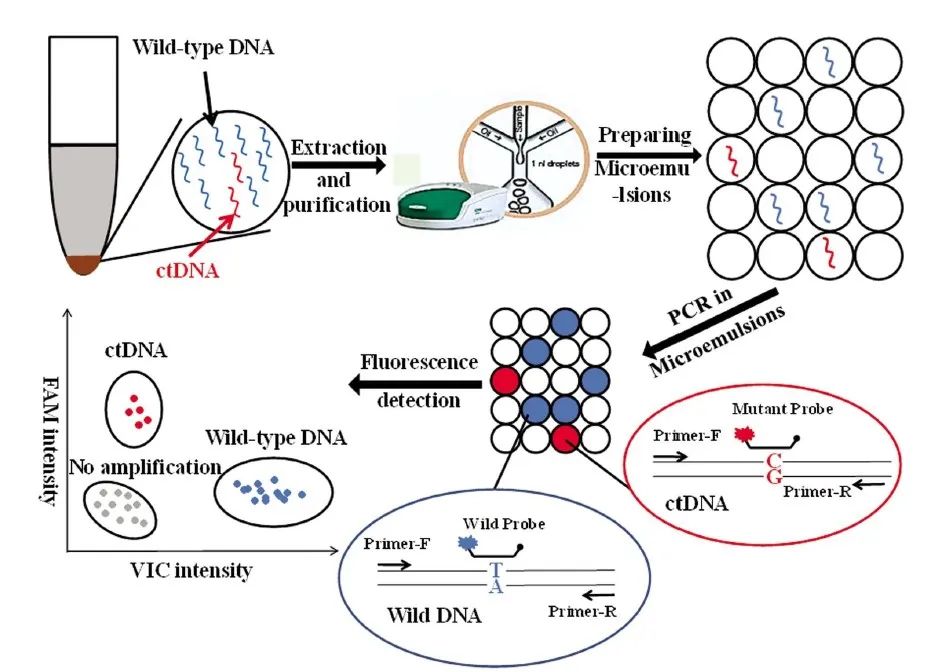
图3 ddPCR 法检测ctDNA 原理图
DdPCR不依赖Ct 值或内参基因,即可确定低至单拷贝的待检靶分子的绝对数目,实现了对核酸样本的绝对定量。同时,ddPCR能够检测出100000个野生型分子中的一个突变,检测极限低至1/100000,具有ARMS-PCR和Clamping-PCR无法比拟的超高灵敏度。
此外,ddPCR与普通PCR相比具有更高的耐受性。这是因为ddPCR技术第一步反应体系的分配过程,它可以使背景序列和PCR反应抑制物被均匀分配到每个微滴反应器中,但大部分反应单元中却并不含有目的序列,从而使得低丰度的目的序列被相对富集于某些反应单元中,显著地降低了这些反应单元中背景序列和抑制物对反应的干扰。
ddPCR的这些优点使其常被用来检测ctDNA的突变情况。尽管如此,目前两种类型的dPCR技术仍然存在着一些不足。
首先,cdPCR 和ddPCR 都只针对单一样本进行分析,这一技术特点决定了其一次性检出率低,检测时间长。其次,荧光检测技术的局限性导致其不能同时检测多种不同类型的遗传学变异,因此该技术的分析通量低,对实验进度有一定影响。最后,精密仪器维护费用和相关配套的试剂耗材成本高昂,这也是限制其广泛应用的另一个重要原因。
- 高通量测序
高通量测序(High-Throughput Sequencing)又名下一代测序(Next-generation Sequencing,NGS), 是相对于传统Sanger测序而言的,指能同时对几十万到几百万个核酸分子进行序列测定的新技术。
高通量测序技术均采用边合成边测序的基本原理,仅测序方法略有区别,主要包括:Illumina/Solexa四色荧光和聚合酶合成原理,Life Technologies/Ion Torrent H+浓度变化和半导体芯片原理以及Roche/454 焦磷酸测序原理等。而目前市场占有率最高的依旧是Illumina 高通量测序技术。
Illumina Miseq测序的核心技术是一种基于单分子簇和专有的可逆终止化学反应的边合成边测序技术。测序时将不同的基因组DNA 随机片段固定到光学透明的玻璃表面(即Flow cell),这些DNA随机片段经过有限循环次数的延伸和桥式扩增后,在Flow cell上形成了数以亿计的不同种类的簇(即Cluster),每个Cluster是具有数千份相同模板的单分子簇。然后使用带荧光基团的四种特殊脱氧核糖核苷酸与分子簇反应,通过利用可逆性终止的边合成边测序原理对待测的模板DNA 进行测序。
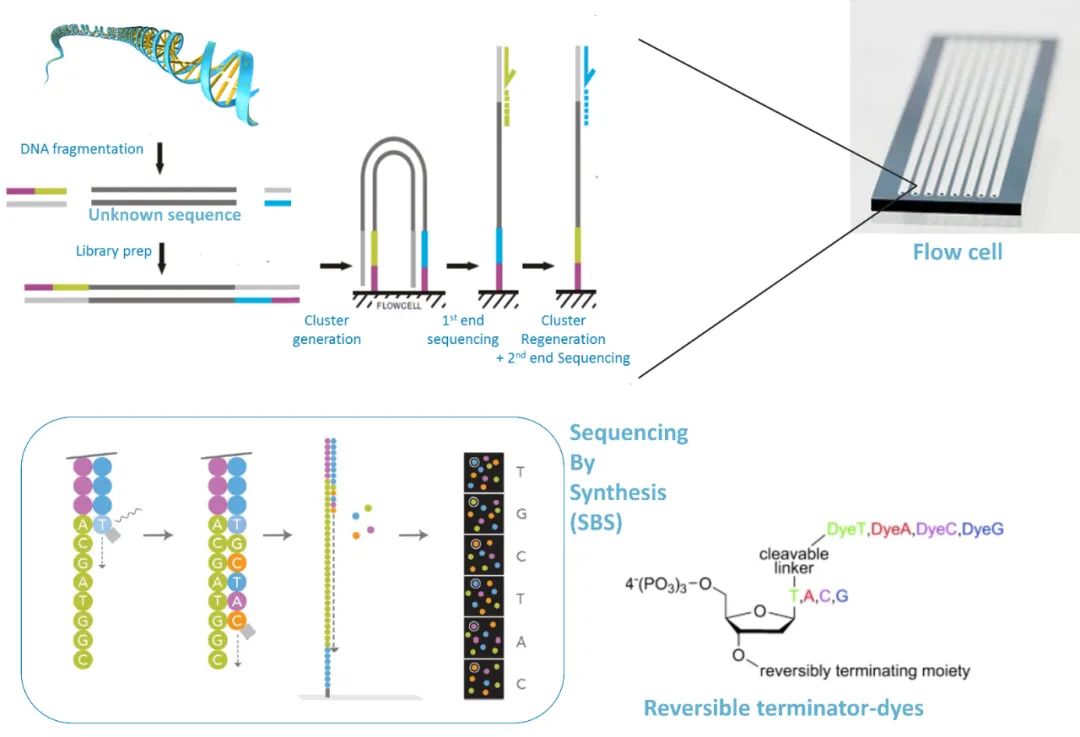
图4 Illumina测序原理图
2014 年,斯坦福大学的研究者们利用Illumina 测序技术平台研究出一种经济且具有高灵敏性的定量检测ctDNA方法,并将其命名为肿瘤个体化分析深度测序(Cancer Personalized Profiling by deep Sequencing,CAPP-Seq)法。
此方法首先利用肿瘤基因突变数据库(Catalogue of Somatic Mutation in Cancer,COSMIC)、肿瘤基因图谱库(The Cancer Genome Atlas, TCGA)和其他数据库中寻找出非小细胞肺癌(NSCLC)患者最常突变的基因和外显子区域,然后再纳入突变外显子区及ALK、ROS1、RET等基因中频发融合断点旁边的外显子和内含子区域,应用一种最大化迭代算法(在尽量减小筛选体系中突变总数量的前提下,使每个病例所携带的或体系内包含的突变数量最大化),最后设计出NSCLC ctDNA 的" 筛选器"(Selector),其能识别139个复发突变基因中的521个外显子及12个内含子,长度约为125kb,平均能够识别4个单核苷酸突变,经过方法优化后选用Illumina HiSeq 2000这种高通量测序方法进行突变检测。
Illumina 测序技术可以在芯片的数百万甚至上千万个点上同时阅读测序,不但测序通量高,而且测序速度快。同时,高通量测序技术还具有定量功能,即样品中DNA被测序的次数反映了样品中这种DNA的丰度。此外,Illumina测序能得到待测样本的所有序列信息,既能对已知突变进行检测也能对未知突变进行检测,且一次检测能得到所有突变位点的检测结果。因此,Illumina测序技术已是ctDNA检测的常用方法。
但目前国内实验室拥有高通量测序仪的并不多, 依托此技术的CAPP-Seq方法较难推广。同时,虽然高通量测序的价格已大幅降低(样本量越大单个样本的费用越低),但一个样本(测序长度约1Mb,测序深度10000× )的检测费用还是需要1000-2500人民币,价格依然昂贵。
此外,目前已知的与肿瘤相关驱动基因数量有限,对于疾病表型和基因型的关系还有赖于生物信息的解读,而高通量测序应用于肿瘤细胞突变检测的标准化和质量控制尚未形成共识。
因此,如何解读二代测序得到的海量序列信息,是制约高通量测序技术应用于临床的瓶颈因素。
总结与展望
循环肿瘤DNA作为新型的肿瘤治疗标志物已成为国内外科研工作者研究的热点,目前已广泛应用于肺癌的早期诊断、EGFR突变检测、靶向药物继发性耐药监测和乳腺癌化疗药物疗效监测等领域。但由于ctDNA自身的生物学特性,ctDNA对检测方法的灵敏度和特异性要求很高。
本文所综述的ARMS-PCR、Clamping-PCR、ddPCR和高通量测序技术等方法与传统的Sanger测序法相比,其灵敏度及特异性均得到很大的提高,且各有优缺点,均能实现对ctDNA 的准确检测(表1)。研究人员可根据自身实验条件和实验目的选择合适的检测方法。
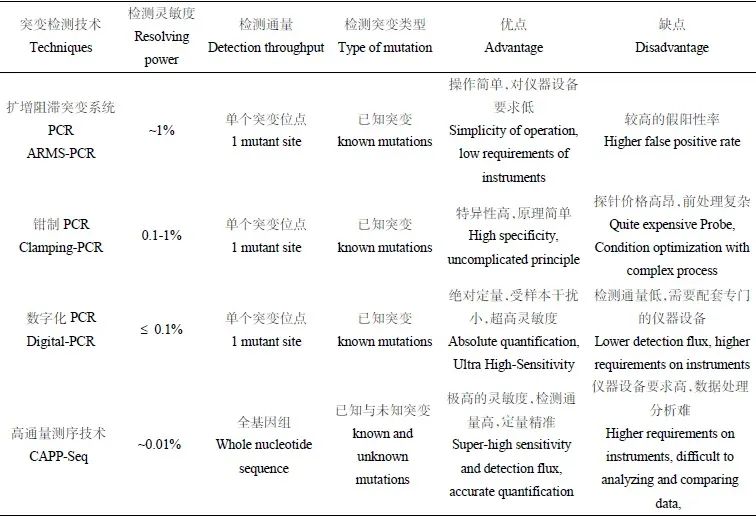
表1 主要ctDNA 突变检测技术比较
除了检测方法本身的特性外,ctDNA的检测结果还受到样本质量、样本类型(唾液、尿液、胸腹水、脑脊液和血浆/ 血清等)、ctDNA含量、ctDNA的提取方法、ctDNA样本的临床病理特征如病理类型、肿瘤分期、肿瘤分化程度等因素的影响。
因此,除了选择合适的检测方法,还需选择标准化的ctDNA处理程序以降低非检测因素对实验结果的影响。只有在样本处理程序标准化和样本检测方法得当的情况下,才能保证ctDNA检测结果的准确性和重复性。
相信随着技术的进步和精准医疗的发展,ctDNA将在不远的将来应用于临床,为医生和患者服务。
迈迪安为肿瘤ctDNA检测的解决方案
| ARMS-PCR分子酶/预混液推荐 | |
|
产品编号 |
产品名称 |
| MDX008 | 抗体热启动Taq聚合酶 |
| MDX020 | Fast qPCR Mix 预混液 |
| MDX009 | 化学热启动Taq聚合酶 |
|
产品编号 |
产品名称 | 作用 |
| MDX207 |
无甘油T4 DNA聚合酶 |
末端修复 |
| MDX208 |
无甘油T4 DNA聚合酶I |
末端修复 |
| MDX206 |
无甘油T4多核苷酸激酶 |
磷酸化 |
| MDX011 |
无甘油Taq DNA聚合酶 |
A尾 |
| MDX200 |
无甘油T4 DNA连接酶 |
链接 |
| MDX203 |
无甘油高保真Pfu聚合酶 |
文库富集 |
| MDX041 | NGS建库磁珠 |
清洗& |
| MDX039 | NGS文库定量试剂盒 | 文库定量 |
本文信息来源:doi: 10.13241/j.cnki.pmb.2018.23.040
↓现在赶紧联系我们订购样品吧↓
进入-》迈迪安丁香通商铺
咨询详情订购样品:
电话:010-85660600
Meridian迈迪安官网:meridianbioscience.com/lifescience
邮箱:info.cn@meridianlifescience.com


Meridian迈迪安致力于提供最高质量的分子原料酶制剂,全球研发生产基地均通过了 ISO 13485认证。我们始终坚持以客户为中心,以高质量的服务和产品为合作伙伴提供价值。
迈迪安产品主要优势包括:
-
一致性:单批次量产,保证可靠的检测
-
优质性能:通过严格质控,减少批间差及污染

美国迈迪安生物科技是一家大规模生产各种抗体,各种病毒抗原,各种重组蛋白质,各类 PCR 多聚酶,各种核苷酸和体外诊断关键试剂的大型领军企业。45年以来,迈迪安生命科学公司始终坚持以提供创新的生命科学解决方案和建立值得信赖的合作伙伴关系为我们的宗旨。我们的重点是为体外诊断试剂公司提供优质的原料和服务,使其有助于推动这些领域的发展。


本周
本月
本年
单个细胞也能提取核酸?超全干货教你微量样本发高分(含完整电子版宝典资料))
LetPub发布最新SCI影响因子查询及期刊投稿分析系统
应用gentleMACS™灌流技术从脂肪肝小鼠模型高效分离肝细胞与非实质细胞
草甘膦(glyphosate)酶联免疫分析(ELISA)试剂盒使用说明书
ELISpot试剂盒限时特惠!一口价低至500元,加赠免费读板服务
LetPub完整SCI影响因子、期刊分区查询系统
Elabscience® 从原料到标记,打造属于中国自己的流式抗体品牌
链脲佐菌素 (Streptozotocin,STZ)-糖尿病动物模型造模
淋巴细胞百分比偏低原因解析
11月Cell重磅:新线粒体细胞死亡方式!或将引爆国自然新热点!先天免疫和代谢信号通过线粒体周膜裂解诱导线粒体依赖性膜裂解
- 促销公告
- 更多 ›


