2 个回答
Eason老歌迷
有帮助
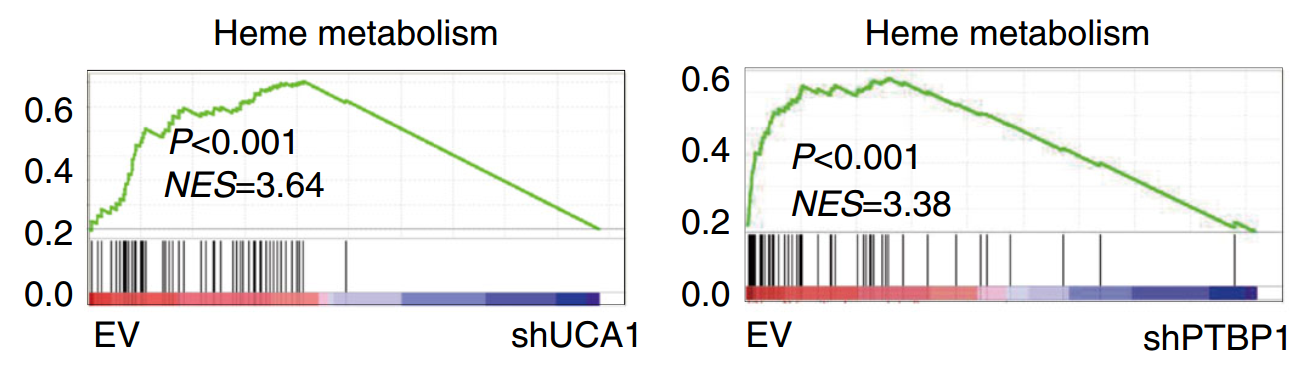
由于ES是根据分析的数据集中的gene是否在一个功能gene set中出现来计算的,但各个功能gene set中包含的gene数目不同,且不同功能gene set与data之间的相关性也不同 较data set在不同功能gene set中的富集程度要对ES进行标准化处理,也就是NES,NES=某一功能gene set的ES/数据集所有随机组合得到的ES平均值。
值为正,表示某一功能gene集富集在排序序列前方。值为负,表示某一功能gene集富集在排序序列后方。
天一湖医者
有帮助
1、图最上面部分展示的是富集分数(ES,enrichment score)值计算过程,从左至右每到一个基因,计算出一个ES值,连成线。在最左侧或最右侧有一个特别明显的峰值就是基因集表型上的ES值。图中间部分每一条线代表基因集中的一个基因,及其在基因列表中的排序位置。
2、最下面部分展示的是基因与表型关联的矩阵,红色为与第一个表型(class A)正相关,在class A中表达高,蓝色与第二个表型(class B)正相关,在class B中表达高。
3、Leading-edge subset 对富集得分贡献最大的基因成员。若富集得分为正值,则是峰左侧的基因;若富集得分为负值,则是峰右侧的基因。
4、FDR GSEA默认提供所有的分析结果,并且设定FDR<0.25为可信的富集,
相关产品推荐
相关问答
提问
扫一扫

实验小助手

扫码领资料
反馈
TOP
打开小程序