GSEA 系列教程 | 快速学会特定基因集的 GSEA 分析,让你文章的水平更高一级!
在前三期的教程中,我们分别学习了如何安装及使用 GSEA,并学习了怎样利用 GSEA 分析数据库中的数据(还不学没关系,文末有传送门)。
不过,在高水平的文章中,我们还会看到针对特定基因集的 GSEA 分析。
也就是说,我们先假设 Case 组和 Control 组的差异是由于某种生物学过程的差异造成的,为了验证我们的假设,我们可以通过对特定的基因集进行 GSEA 分析,以验证我们的假设是否正确。
这样的分析不仅仅能够解释说明具体的科学问题,还会使文章显得高大上,如下图:
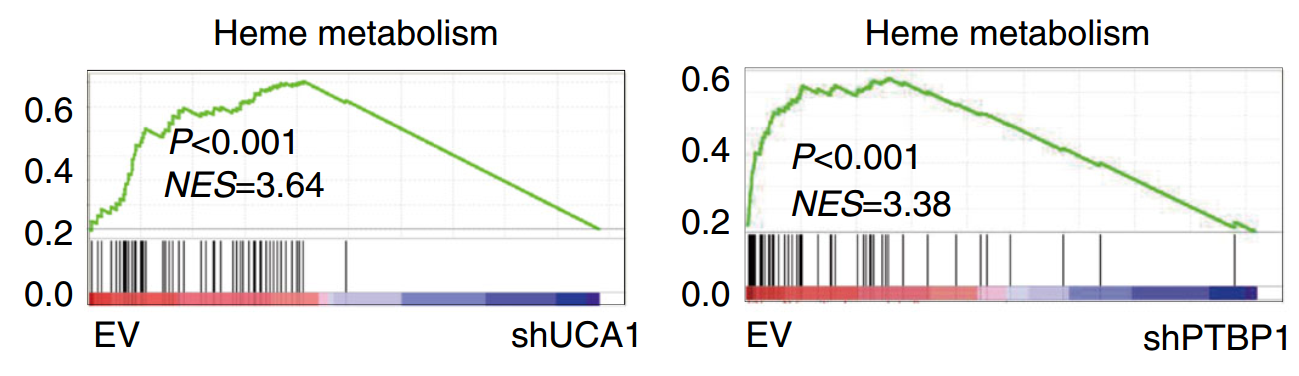
图片来源:Nature Communications
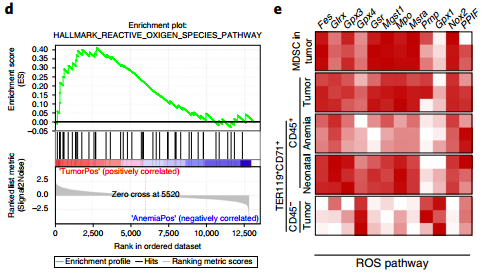
图片来源:Nature Medicine
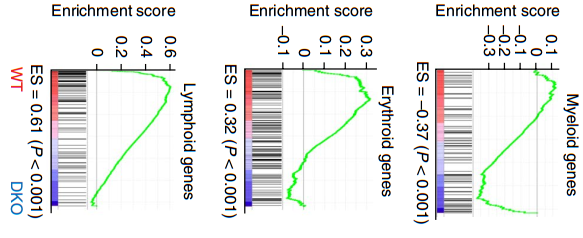
图片来源:Nature Immunology
今天,我们就系统地教大家如何对特定基因集进行 GSEA 分析。
我们选择的数据和上一期教程中的一样,仍是安秀丽老师团队最近在 Blood 上发表的一篇文章中的转录组数据 --「Identification and transcriptome analysis of erythroblastic island macrophages」。
PS:原文链接:
https://doi.org/10.1182/blood.2019000430

数据的具体信息大家可以通过阅读文献原文或者参考我们上一期的教程获得。
准备好输入数据之后,打开 GSEA 软件,点击左上角的「Load data」来导入我们的数据。
和上期教程一致,导入表达矩阵数据和样本类型解释文件,此时我们可以看到以下界面:成功导入 2 个文件,无错误。
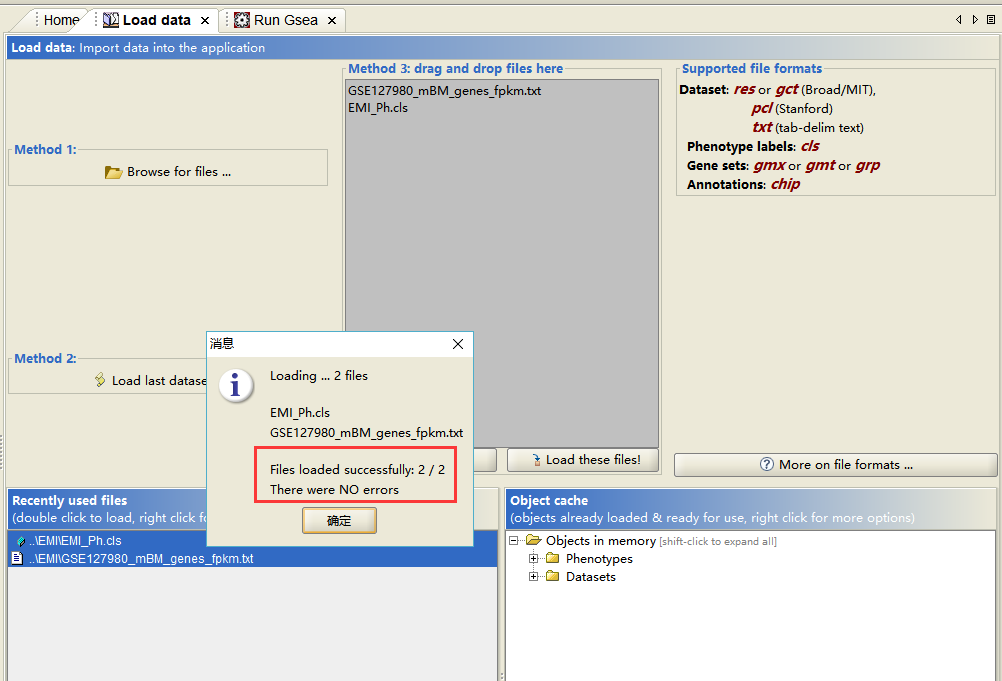
数据导入成功之后,接下来就是今天的重点:针对特定基因集的 GSEA 分析。
由于我们使用的转录组数据是血岛中的巨噬细胞(GFP+)和非血岛中的巨噬细胞(GFP-),因此我们首先假设:相比于 GFP - 的细胞,GFP + 的细胞会更加富集与红细胞发育相关的通路。
接下来就是来验证我们假设的时候啦!
1、首先,前往 GSEA 官网,点击进入「Molecular Signatures Database」。
该数据库中包含了几乎所有的生物学相关通路及生物学过程(Biological Process),对我们做生物的人来说是非常的好的一个数据库。
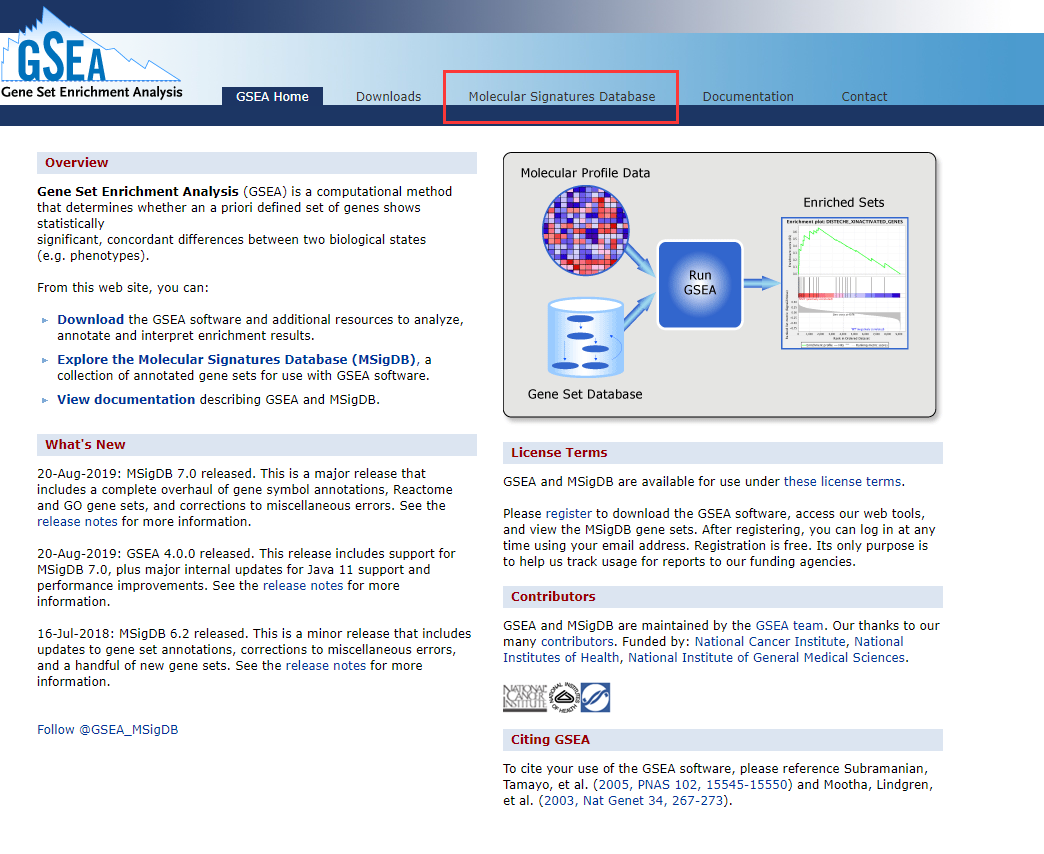
由于我们是带有目的性去寻找相关基因集,所以说就不用逐个浏览了。
2、我们直接选择「Search Gene Sets」,然后在搜索框中输入「erythrocyte development」,之后就是点击「search」。如下图:
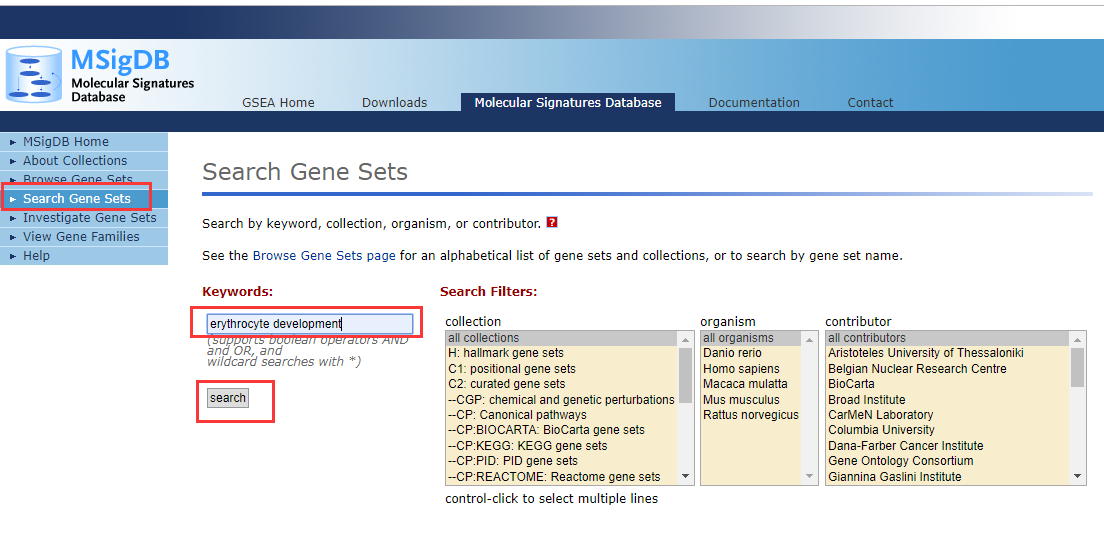
我们看到共搜到 968 条与「erythrocyte」和「development」相关的基因集。
经过简单筛选之后,我们选择了「GO_ERYTHROCYTE_DEVELOPMENT」这个基因集,点击该基因集。
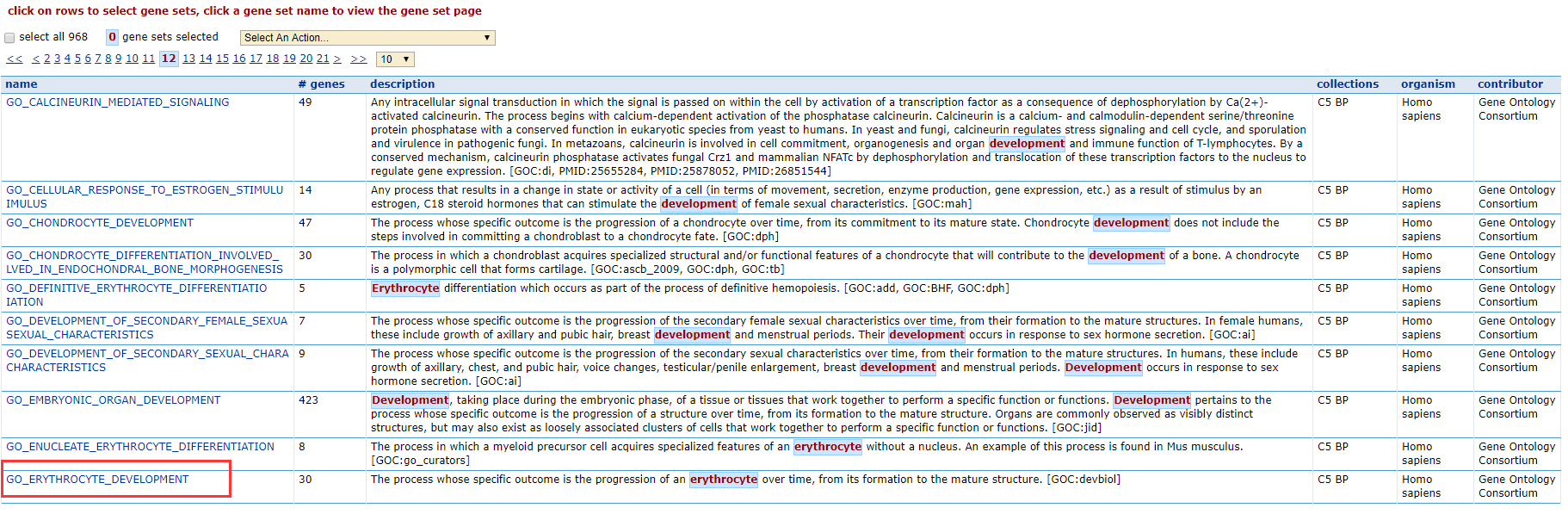
3、然后就会跳到如下图的界面,包括了该基因集的一些详细信息。我们需要做的是下载该基因集,在下载行有多种格式,我们选的格式为「grp」格式。
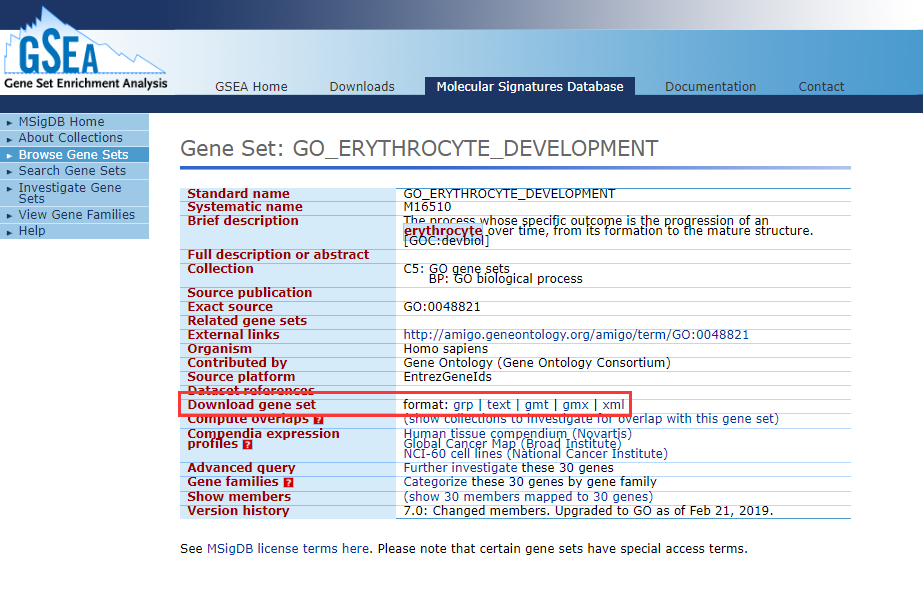
4、成功下载该数据集之后,和导入表达矩阵相似,我们在 GSEA 软件中导入该基因集。
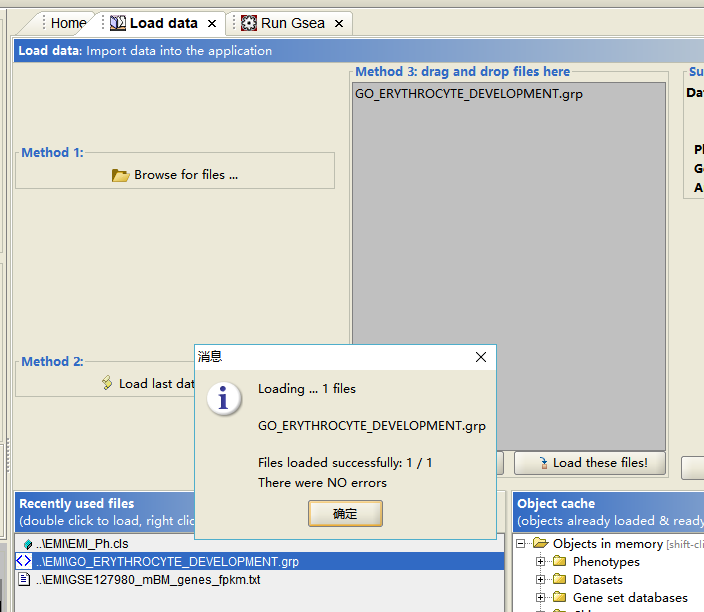
5、和上一期的教程不同,在「Run Gsea」时,我们选择「Gene sets database」中的「Gene sets (grp)」,然后选择我们导入的目标基因集。
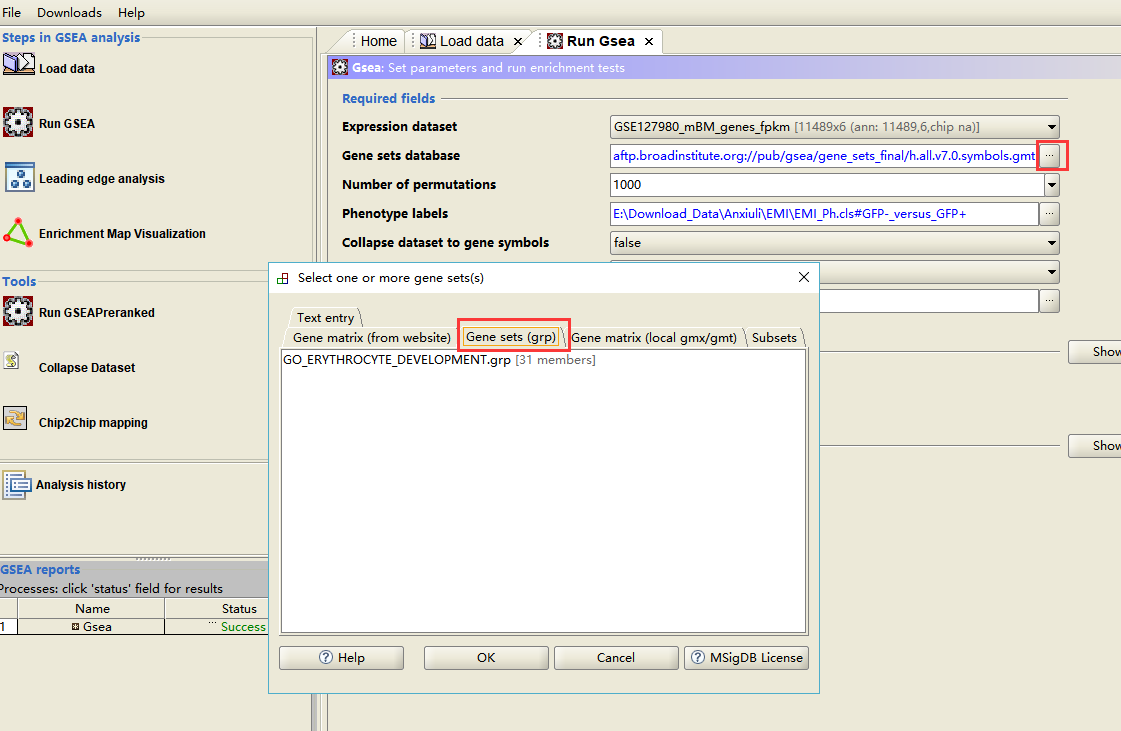
6、选中之后,就直接运行,然后看到左下角的成功状态之后,点开。
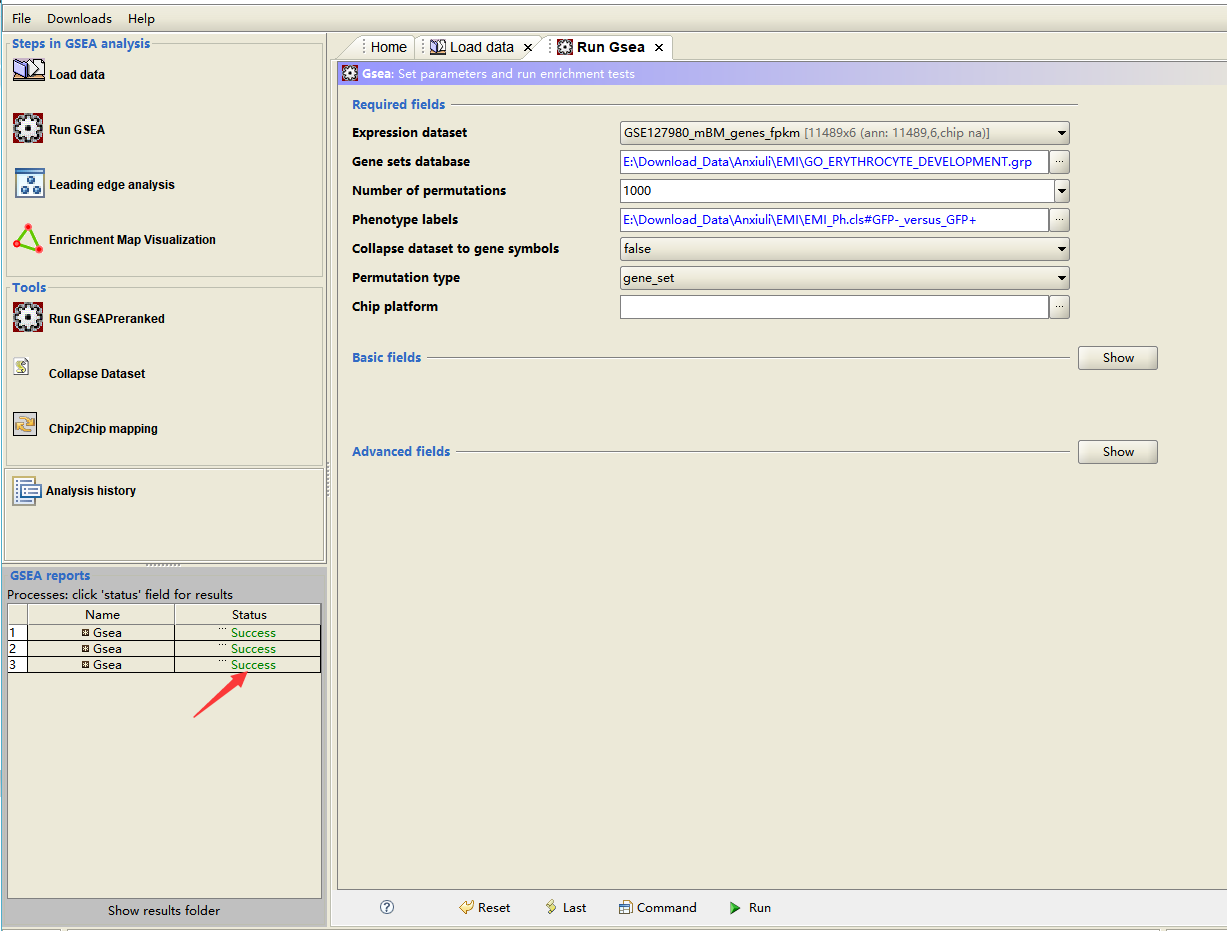
7、在网页版的分析结果中,我们看到,该基因集在 GPF + 组(血岛中的巨噬细胞)高表达。这是和预期相符的,也就是说初步验证了我们的假设。
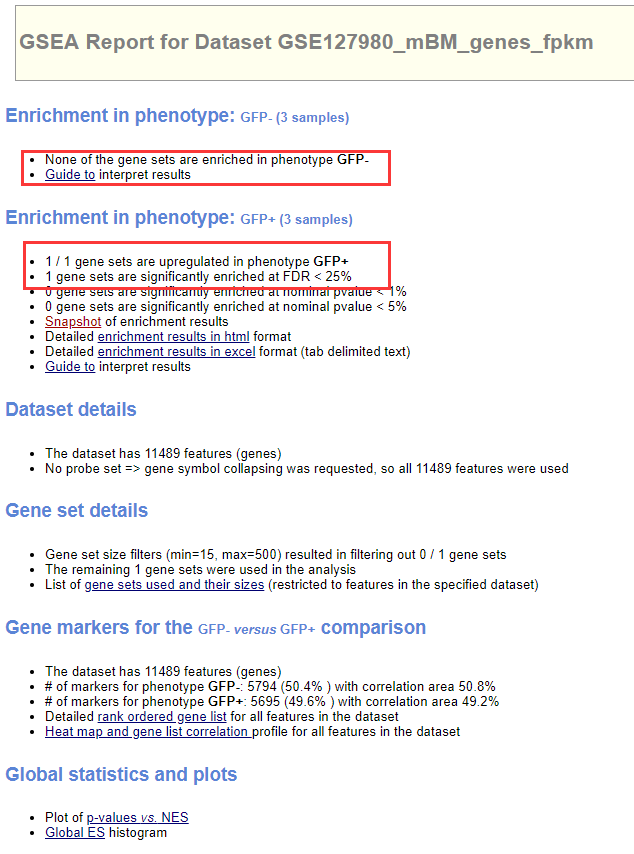
8、点击「Snapshot」查看更加详细的富集信息。
我们看到该基因集在 GFP + 组上调,尽管其 P 值和 FDR 没有小于常规的标准 0.05,但我们仍然看到了其在 GFP + 组上调的这种趋势。
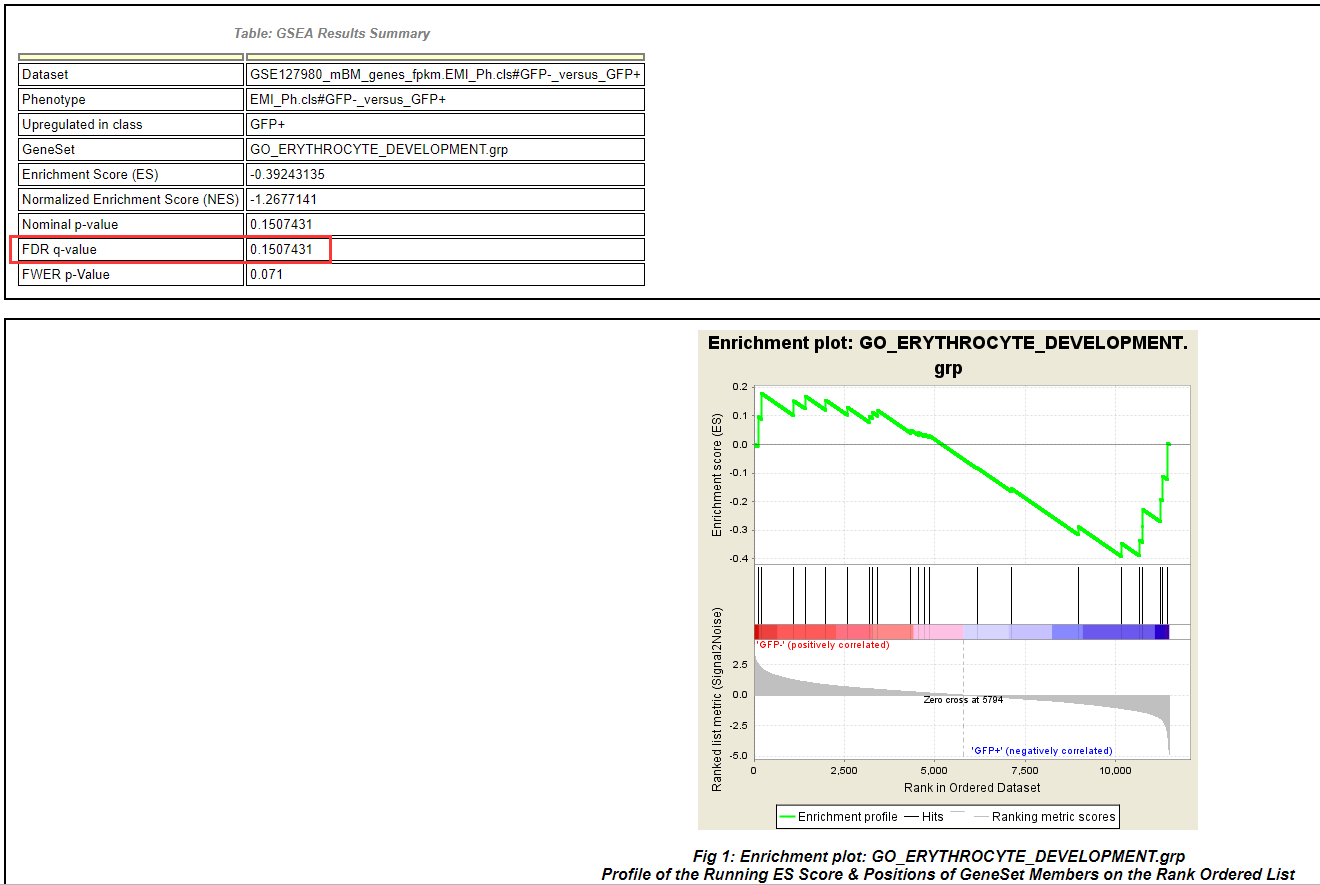
至此,我们就完成了对特定基因集的 GSEA 分析。
通过系统学习这四期的教程,相信你一定能够熟练掌握 GSEA 的各种分析方法。
不过,学会了如何进行 GSEA 固然重要,但要做出高质量的、达到发表水平的文章,仅仅这些学习还是不够充分的。
在做 GSEA 分析时如何选择数据库?
如何选择合适的基因集进行分析?
有哪些技巧可以使你的分析结果更加漂亮呢?
持续关注丁香学术,敬请期待下期教程!
参考文献:
1、Liu, J. et al. Long non-coding RNA-dependent mechanism to regulate heme biosynthesis and erythrocyte development. Nature Communications 9, doi:10.1038/s41467-018-06883-x (2018)
2、Zhao, L., et al. (2018). "Late-stage tumors induce anemia and immunosuppressive extramedullary erythroid progenitor cells." Nat Med 24(10): 1536-1544.
3、Kato, H., et al. (2018). "Infection perturbs Bach2- and Bach1-dependent erythroid lineage 'choice' to cause anemia." Nat Immunol 19(10): 1059-1070.
图片来源:作者提供