MEGA遗传距离矩阵怎么看
dxy_iitpuzy7
组间分析结果为什么只有竖行有分组的名称,还是说抬头的横行的1,2,3,4...和竖行的分组名称是对应的,还有个疑问就是用核酸测序结果做遗传距离那种模型更正确呢?我看有用 Tajima-Nei 也有k2P,p-distance的,用那种好点呢,谢谢大家!!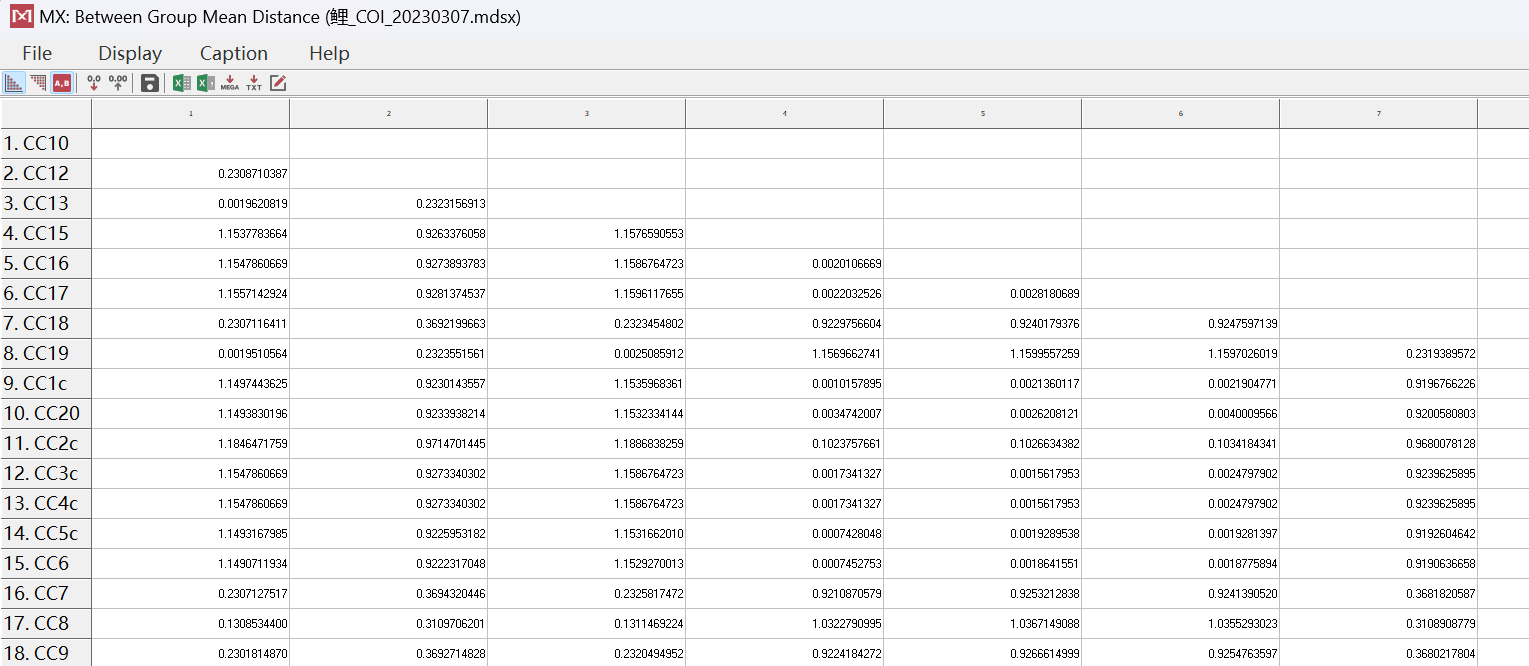
分享
2 个回答
土井挞克树
有帮助
是对应的,模型选择方面p-distance的成功率比较高
loveliufudan
有帮助
关于您的第一个问题,组间分析的结果中通常只在竖行上标注分组名称,这是因为竖行代表的是各个样本或处理组的数据,而横行则是对应的各个检测指标或基因。横向的1、2、3、4...表示的是样本或处理组的编号,而竖向的分组名称则是对应着各个样本或处理组所属的实验条件或分类信息。因此,竖向的分组名称和横向的编号是一一对应的。
至于您的第二个问题,不同的遗传距离模型适用于不同类型的遗传数据和研究目的。Tajima-Nei模型适用于基因频率较为平均的情况,如多态性较高的基因或微卫星等。而k2P(Kimura 2-parameter)和p-distance模型适用于比较基因或序列之间的差异性,其中k2P模型更加适用于较为保守的序列,而p-distance模型则适用于基因差异较大的情况。
因此,在选择遗传距离模型时,需要根据具体的实验数据和研究目的进行选择和优化。如果您不确定选择哪种模型,可以同时使用多种模型进行分析,以获取更加全面和准确的结果。
dxy_iitpuzy7![]()
好的谢谢!
相关产品推荐
相关问答
提问
扫一扫

实验小助手

扫码领资料
反馈
TOP
打开小程序