肿瘤研究的核心科学问题:挑战与复杂性交织的生命谜题
百奥智汇
前言:
肿瘤研究的核心科学问题:挑战与复杂性交织的生命谜题
肿瘤,这一古老而顽固的生命之敌,其发生、发展与演化的复杂性远超想象。理解肿瘤,本质上是解析一个在时空维度上动态演化的异常生态系统。其核心科学问题构成了一座座亟待征服的科学高峰:
肿瘤异质性的本质与根源:肿瘤绝非单一克隆的简单堆积。其内部存在惊人的遗传变异(驱动基因突变、拷贝数变异、染色体不稳定性)、表观遗传重塑(DNA 甲基化、组蛋白修饰改变)以及由此驱动的细胞状态(干性、分化、间质化、代谢状态)和功能(增殖、侵袭、迁移、治疗抵抗)的多样性。这种异质性如何在肿瘤进化树中产生(分支进化 vs. 线性进化)?微环境压力(如免疫攻击、缺氧、营养剥夺)如何塑造和选择不同的克隆或亚群?肿瘤干细胞在维持异质性和驱动复发转移中扮演何种角色?理解异质性层次(瘤内、瘤间、原发灶与转移灶间)及其动态变化,是破解肿瘤可塑性、治疗抵抗和转移扩散的关键。
肿瘤微环境(TME)的构成、功能及其双向互作:肿瘤并非孤立存在,而是深嵌于一个由免疫细胞(T 细胞、B 细胞、NK 细胞、巨噬细胞、髓系抑制细胞等)、基质细胞(成纤维细胞、内皮细胞、周细胞)、神经细胞、细胞外基质(ECM)以及复杂信号分子(细胞因子、趋化因子、生长因子、代谢物)共同构成的动态微环境中。TME 如何从抑制肿瘤转变为促进肿瘤生长、侵袭、血管生成、免疫逃逸和转移的「帮凶」?不同细胞组分间如何通过直接接触或分泌因子进行复杂通讯(crosstalk),形成支持肿瘤的生态位?免疫细胞在 TME 中为何功能失调或耗竭?如何打破 TME 的免疫抑制屏障?TME 的空间结构(如三级淋巴结构的存在与位置)如何影响抗肿瘤免疫应答和预后?
免疫逃逸机制的多样性:肿瘤如何巧妙地躲避或抑制宿主的免疫监视和清除(免疫编辑理论)?是下调抗原呈递(MHC-I/II 丢失)、表达免疫检查点分子(PD-L1, CTLA-4 等)、招募调节性免疫细胞(Treg, MDSC)、分泌免疫抑制因子(TGF-β, IL-10, VEGF)、还是营造物理屏障(致密基质)?不同肿瘤类型、不同个体、不同治疗阶段的核心逃逸机制有何差异?如何精准识别并逆转这些机制?
治疗抵抗的动态演化机制:无论是化疗、靶向治疗还是免疫治疗,耐药性几乎是无法回避的终极挑战。抵抗是源于预先存在的耐药克隆的选择性扩增,还是在治疗压力下新产生的适应性演化(如药物靶点突变、旁路信号激活、表型转换、药物外排泵上调)?TME 如何在其中提供保护性生态位?肿瘤细胞如何通过改变代谢(如利用自噬、改变能量来源)来应对治疗压力?理解抵抗的动态发生过程对于设计克服耐药的联合疗法至关重要。
肿瘤起始、转移定植与休眠的奥秘:正常细胞如何积累关键驱动事件跨越恶性转化的临界点?早期事件如何影响后续演进?转移这一「致命步骤」如何发生?循环肿瘤细胞(CTCs)如何存活、穿越脉管、在远处器官定植并适应新的微环境形成转移灶?肿瘤细胞为何能在转移后长期处于休眠状态?哪些因素(如免疫监视、微环境信号)可唤醒休眠细胞导致迟发转移?揭示这些过程的机制是预防和阻断转移的核心。
这些核心问题相互交织,构成了肿瘤研究的宏伟蓝图。传统基于群体细胞(Bulk)的研究方法,往往掩盖了关键细胞亚群或稀有细胞的行为,也无法解析空间位置信息带来的关键调控作用。因此,亟需更高分辨率、更全面维度的研究工具来破译这些生命之谜。单细胞组学与空间组学技术的崛起,正为我们提供前所未有的「显微镜」和「空间定位仪」,引领肿瘤研究进入一个全新的精密解析时代。
单细胞测序技术:解构肿瘤异质性与微环境的革命性透镜
单细胞测序技术的突破性在于它将分析的精度从组织/细胞群体的「平均化」水平,提升到单个细胞的「个体化」层面。这种分辨率的跃迁,为揭示肿瘤的复杂性和异质性提供了强大的武器库,在肿瘤研究中开辟了广阔的应用方向:
绘制肿瘤细胞图谱与分子分型精细化
单细胞测序技术,尤其是单细胞 RNA 测序, scRNA-seq,能够无偏倚地鉴定肿瘤组织中存在的所有细胞类型和状态,揭示前所未有的细胞多样性。它可以:
- 识别新的、稀有的或功能特化的肿瘤细胞亚群(如高度转移潜能的细胞、持久性细胞、耐药前体细胞)。
- 超越传统的病理分型或 Bulk 组学分型,建立基于单细胞分子特征的、更精细、更具生物学意义和临床预测价值的肿瘤分子分型。
- 揭示肿瘤细胞在分化轨迹、细胞周期、应激反应、代谢状态等方面的连续过渡状态(拟时序分析),理解肿瘤细胞可塑性的动态过程。
- 解析肿瘤内不同克隆的转录组特征、进化关系(系统发育树构建)及其对微环境信号的响应差异。
深度解析肿瘤免疫微环境
单细胞测序是剖析 TME 中细胞组成和功能状态的黄金标准,它可以详细解析 TME 中的免疫细胞和基质细胞,包括:
- 精确量化各种免疫细胞亚型(如不同功能状态的 T 细胞、B 细胞亚群、巨噬细胞极化状态、树突状细胞亚群、髓系来源抑制细胞、NK 细胞等)的比例和丰度变化。
- 揭示免疫细胞的激活、耗竭、功能障碍或抑制状态的关键分子特征(如检查点分子表达谱、细胞毒因子、衰竭标志物)。
- 通过 T 细胞受体(TCR)和 B 细胞受体(BCR)测序(scTCR-seq / scBCR-seq),追踪肿瘤浸润淋巴细胞(TILs)的克隆扩增、多样性、克隆型共享及迁移规律,评估抗原驱动的免疫应答强度和广度,识别潜在的新生抗原反应性克隆。
- 理解免疫细胞与其他 TME 组分(如肿瘤细胞、CAFs)之间的潜在相互作用(配体-受体对分析)。
- 鉴定具有不同促瘤功能(如介导免疫抑制、驱动纤维化、促进血管生成、支持肿瘤干细胞)的基质细胞亚群。
- 解析这些亚群的空间来源(如来自正常成纤维细胞的转化、内皮间质转化、脂肪细胞或周细胞)及其独特的分子特征和信号通路。
- 揭示基质细胞在介导治疗抵抗(如形成物理或生化屏障)中的关键作用。
整合多组学解析细胞状态与调控网络
单细胞多组学技术(如 CITE-seq, REAP-seq: 同时测转录组和表面蛋白;scATAC-seq: 染色质可及性;scDNA-seq: 基因组变异;scMulti-omics: 同时多种组学)可在一个细胞中关联多个层面的信息。
- 将基因表达与关键表面蛋白表达关联,更精确地定义细胞身份和状态。
- 揭示驱动细胞状态转变和异质性的表观遗传调控基础(如超级增强子、关键转录因子结合位点可及性变化)。
- 在单细胞水平关联基因突变与转录/表观表型,理解基因型-表型关系。
- 构建更精准的细胞类型特异的基因调控网络。
追踪肿瘤进化与克隆动态
通过单细胞测序解析点突变、CNV 等,可以在单细胞分辨率下:
- 重建肿瘤的系统发育树,解析克隆起源和进化路径(分支进化 中性进化)。
- 追踪驱动克隆在肿瘤发生、发展、转移和复发过程中的动态演变和选择。
- 揭示治疗压力下耐药克隆的起源(预先存在 新生)、选择扩增和分子适应机制。
发现诊断、预后标志物与治疗新靶点
单细胞测序产生的海量单细胞数据是生物标志物和新靶点发现的源泉。
- 识别与特定肿瘤亚型、转移倾向、治疗反应(敏感/抵抗)或不良预后显著相关的稀有细胞群或特异性分子特征(基因、通路、细胞状态)。
- 发现肿瘤细胞或 TME 细胞(如特定 CAF 亚群、Treg 亚群)表面或分泌的新型治疗靶点。
- 为个体化治疗(如基于 TIL 克隆状态选择免疫治疗、针对特定 CAF 亚群)提供理论依据。
单细胞测序技术极大地深化了我们对肿瘤复杂性、异质性和微环境互作的理解。然而,由于前处理的组织解离等步骤,它不可避免地「丢失」了细胞在原位组织中的空间位置信息,而这对于理解细胞间通讯、功能区域化(如免疫排斥区、侵袭前沿)和 TME 的三维组织结构至关重要。空间组学技术的兴起,正是为了弥补这一关键维度的缺失。
空间组学技术:描绘肿瘤微环境功能图谱与细胞社会学的定位导航
肿瘤的发生发展绝非孤立细胞的随机行为,而是高度依赖其所在的空间位置和与邻近细胞及结构的相互作用。空间组学(Spatial Omics)技术革命性地保留了组织切片中分子的空间位置信息,使我们能够在原位「看到」基因表达、蛋白丰度甚至代谢活动的空间分布图谱,从而将单细胞维度的分子特征精确地锚定在组织结构的背景中,为理解肿瘤的「细胞社会学」提供了不可替代的工具。其在肿瘤研究中的应用方向正迅速拓展:
解析肿瘤组织结构的空间异质性
空间转录组学(如 10x Visium, Stereo-seq)和空间蛋白组/多组学(如多重免疫荧光 mIHC/IF, CODEX,LCM-MS,Stereo-cite 等)能够:
- 可视化肿瘤核心区、侵袭前沿、肿瘤-正常组织交界区、坏死区域、血管周围区域等不同功能区域的独特分子特征(基因表达程序、信号通路活性、蛋白表达谱)。
- 揭示肿瘤内不同分子亚型(由单细胞数据定义)的空间分布模式(混合分布/区域化隔离)及其与组织学特征(如分级、分期)的关联。
- 识别具有特殊空间定位和功能的肿瘤细胞亚群(如位于侵袭前沿具有高迁移能力的细胞)。
绘制肿瘤免疫微环境的空间图谱
理解免疫细胞的空间组织是破解免疫治疗响应的关键,空间组学检测能够:
- 精确描绘不同免疫细胞亚型(如 CD8+ T 细胞、Tregs、巨噬细胞、B 细胞)在肿瘤组织中的空间分布、丰度及相互邻近关系(邻域分析)。
- 识别关键的免疫微环境结构:
三级淋巴结构(TLS):空间技术可清晰定位 TLS(B 细胞滤泡、T 细胞区、生发中心、高内皮微静脉),评估其成熟度、位置(瘤内 vs. 瘤周)及其与预后的关系。分析 TLS 内部及周边免疫细胞的空间相互作用。
免疫排斥(Excluded)模式:可视化免疫细胞(特别是 CD8+ T 细胞)被限制在肿瘤实质边缘或间质中的现象。
免疫浸润(Inflamed)模式:显示免疫细胞(尤其是活化的 CD8+ T 细胞)深入浸润到肿瘤实质内部。
- 研究免疫检查点分子(如 PD-1, PD-L1, CTLA-4)及其配体在肿瘤细胞、免疫细胞、基质细胞上的空间共定位模式。例如,肿瘤细胞 PD-L1 是否与邻近的 PD-1+ T 细胞直接接触?这种空间邻近性是预测免疫治疗反应的重要指标。
- 分析免疫细胞与肿瘤细胞、基质细胞(如 CAFs)之间潜在配体-受体相互作用的空间共定位,推测原位信号传递。
揭示基质细胞的空间功能分区及其互作网络
CAFs、内皮细胞等基质细胞的功能高度依赖其空间位置,空间组学检测能够:
- 鉴定定位于肿瘤核心、侵袭前沿、血管周围或纤维化包膜等不同区域的 CAF 亚群,揭示其位置特异的分子特征和功能(如介导侵袭、免疫抑制、血管生成、代谢支持)。
- 可视化肿瘤血管网络的结构异常(如渗漏性、灌注不足)及其空间分布,分析不同血管区域(如尖端细胞、茎细胞区域)的分子特征和周围细胞组成(如周细胞覆盖、免疫细胞粘附)。
- 描绘细胞外基质(ECM)成分(如胶原纤维类型、交联程度、硬度相关蛋白)的空间重塑,及其对肿瘤细胞迁移、侵袭和免疫细胞浸润的物理化学屏障作用。
研究细胞间通讯的空间规则
空间邻近性是细胞间直接通讯(如膜蛋白介导的接触、突触)和旁分泌信号传递的基础,基于空间组学检测能够:
- 通过分析空间上邻近的细胞对的分子特征(配体-受体表达),推断潜在的原位细胞间相互作用网络(细胞邻域分析、利基分析)。
- 研究关键信号通路(如 WNT, NOTCH, TGF-β)的激活状态在空间上的传递模式(如从侵袭前沿到核心)。
- 揭示肿瘤细胞如何在其特定空间位置(如靠近血管或特定 CAF 亚群)获得生存、增殖或耐药优势。
整合单细胞与空间数据构建高分辨率空间图谱
结合 scRNA-seq/scMulti-omics 数据(提供高分辨率细胞类型和状态定义)与空间技术数据(提供位置信息),通过计算反卷积或直接空间分子图谱(如高分辨率原位检测 Xenium 等):
- 将单细胞定义的精细细胞类型/状态映射回组织空间位置,构建「细胞类型/状态空间图谱」。
- 在空间背景下验证单细胞数据推断的细胞间相互作用。
- 发现具有特定空间定位模式的稀有细胞亚群(如空间定义的侵袭性肿瘤细胞亚群、特定免疫细胞互作对)。
推动空间病理学与精准诊疗
- 在保留组织形态学的基础上,增加分子表达的空间维度信息,极大丰富病理诊断的内涵(「Spatial Pathology」),发现新的空间组织结构相关的诊断和预后标志物。
- 识别预测治疗反应(尤其是免疫治疗)的空间生物标志物(如 CD8+ T 细胞与肿瘤细胞的邻近度、PD-1/PD-L1 共定位评分、TLS 的存在与位置)。
- 发现具有空间特异性的新治疗靶点(如特定区域 CAF 分泌的因子、空间受限的免疫抑制信号)。
- 指导手术边界确定(识别分子定义的侵袭前沿)和放疗靶区勾画(识别免疫激活或抑制区域)。
空间组学技术正在彻底改变我们对肿瘤作为一个空间组织化生态系统的理解。它揭示了位置决定命运、邻居影响行为的肿瘤「社会法则」,为理解肿瘤发生、免疫逃逸、转移和治疗抵抗提供了全新的空间维度视角。与单细胞技术的融合,将使我们能够构建前所未有的、包含完整细胞类型、分子状态和空间位置信息的高分辨率「肿瘤生态地图」。
单细胞与空间组学联用,揭开肿瘤研究中的迷雾
单细胞与空间组学技术的协同应用,如同为我们装备了洞察肿瘤微观宇宙的「超级显微镜」和「空间定位仪」。
本手册旨在以研究方向为线索,通过 10 篇经典案例,梳理这两大前沿技术在肿瘤研究各核心领域的应用策略。我们期望它能成为肿瘤研究工作者、临床医生和技术开发人员的重要参考,助力大家更深入地解析肿瘤的复杂性,发现新的生物学机制、诊断标志物和治疗靶点,最终推动肿瘤精准医学的发展,惠及广大患者。让我们携手,利用这些强大的工具,共同揭开肿瘤的重重迷雾。
参考文献:
- Gulati, G.S., D’silva, J.P., Liu, Y. et al. Profiling cell identity and tissue architecture with single-cell and spatial transcriptomics. Nat Rev Mol Cell Biol 26, 11–31 (2025).
- Chen, J., Larsson, L., Swarbrick, A. et al. Spatial landscapes of cancers: insights and opportunities. Nat Rev Clin Oncol 21, 660–674 (2024).
- Walsh, L.A., Quail, D.F. Decoding the tumor microenvironment with spatial technologies. Nat Immunol 24, 1982–1993 (2023).
- Ren X, Zhang L, Zhang Y, Li Z, Siemers N, Zhang Z. Insights Gained from Single-Cell Analysis of Immune Cells in the Tumor Microenvironment.Annu Rev Immunol. 2021;39:583-609. doi:10.1146/annurev-immunol-110519-071134
- Rozenblatt-Rosen O, Regev A, Oberdoerffer P, et al. The Human Tumor Atlas Network: Charting Tumor Transitions across Space and Time at Single-Cell Resolution.Cell. 2020;181(2):236-249. doi:10.1016/j.cell.2020.03.053
- Lim B, Lin Y, Navin N. Advancing Cancer Research and Medicine with Single-Cell Genomics. Cancer Cell. 2020 Apr 13;37(4):456-470. doi: 10.1016/j.ccell.2020.03.008. PMID: 32289270; PMCID: PMC7899145.
- Baslan T, Hicks J. Unravelling biology and shifting paradigms in cancer with single-cell sequencing.Nat Rev Cancer. 2017;17(9):557-569. doi:10.1038/nrc.2017.58
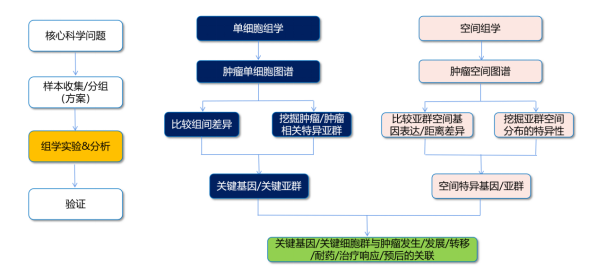
常用研究框架&数据分析思路总结