ChIP 实验面面观,博士师兄吐血总结推荐
CST中国(Cell Signaling Tech
编首语:这篇文章详细分析了 ChIP 操作的关键步骤和操作细节,让您的染色质免疫沉淀(ChIP)实验变轻松。
小板凳备好、小零食备好、阿猫阿狗管好……对面有一大波干货袭来,请摆好姿势准备迎接!
华丽丽的分割线……
一、ChIP 实验的基本原理和主要应用?
真核生物的基因组 DNA 以染色质(Chromatin)的形式存在。因此,研究蛋白质与 DNA 在染色质环境中的相互作用是阐明真核生物基因表达机制的基本途径,而染色质免疫共沉淀(Chromatinimmunoprecipitation,ChIP)技术是目前公认的研究此相互作用的最佳选择,是真核生物基因表达机制研究中不可或缺的核心技术之一。
它的基本原理是在活细胞状态下,固定蛋白质-DNA(染色质)复合物,并将其切断为一定长度范围内的染色质小片段,然后通过免疫学方法(抗体亲和)沉淀此复合体,特异性地富集目的蛋白结合的 DNA 片段,通过对目的片段的纯化与后期检测,从而获得蛋白质与 DNA 相互作用的信息。
ChIP 不仅可以检测体内多种转录因子与 DNA 的动态作用,还可用来研究组蛋白的各种共价修饰(如磷酸化、乙酰化、甲基化、泛素化等)与不同状态下基因表达的关系。而且,ChIP 与多种高通量检测方法的结合,扩大了其应用范围和测定效率:ChIP 与高通量测序技术相结合建立的 ChIP-seq 方法已广泛用于特定转录因子靶基因的高通量分析和鉴定;ChIP 与体内足迹法相结合,用于寻找转录因子的体内结合位点;RNA-ChIP 用于研究 RNA 与蛋白的相互作用以及 RNA 在基因表达调控中的作用。由此可见,随着 ChIP 的进一步完善,它必将会在基因表达调控研究中发挥越来越重要的作用。
二、ChIP 实验的一般流程是怎样的?
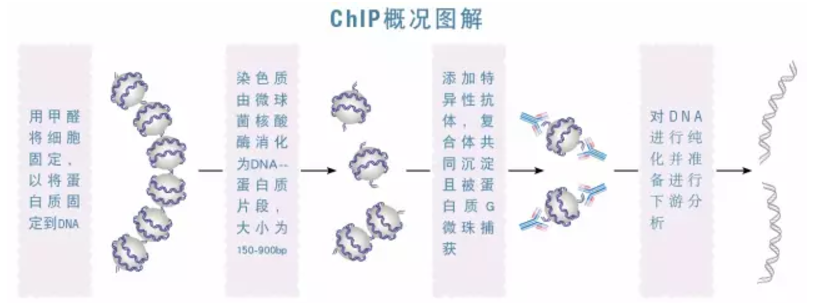
ChIP 的一般流程是:甲醛固定细胞---收集细胞,将染色质复合物中 DNA 切割---加入目的蛋白的抗体,与靶蛋白-DNA 复合物相互结合---加入 Protein A 或 Protein G 微珠,结合抗体-靶蛋白-DNA 复合物并沉淀---对沉淀下来的复合物进行清洗,除去一些非特异性结合---洗脱,得到富集的靶蛋白-DNA 复合物---解交联,纯化富集的 DNA 片段---PCR 或测序分析。
随着 ChIP 技术的不断优化,在对甲醛固定的染色质复合物中 DNA 进行切割时,传统超声随机打断 DNA 的方法逐渐被酶解法所替代。利用微球菌核酸酶的切割特性(在染色质核小体之间的 DNA 更容易被切开),可以快速温和地将染色质 DNA 切断,且最大限度地保持了细胞原有染色质结构以及目的蛋白与 DNA 的结合状态,使 ChIP 的准确性和结果可信度大大提高。
CST 推出的 SimpleChIP® 酶解法试剂盒,含有标准 ChIP 流程所需的整套试剂、ChIP 级蛋白质 G 微珠、阳性对照抗体(Histone H3(D2B12) XP® Rabbit mAb (ChIP Formulated) #4620)、阴性对照抗体、DNA 富集柱、ChIP 引物等,方法简便易行,性价比非常高,与 CST 多种高质量的 ChIP 级抗体配合使用,将使您的 ChIP 实验质量提升到一个新的水平。具体操作步骤,请参考 CST 官方网站获取:http://www.cellsignal.com/common/content/content.jsp?id=resources-protocols。
三、ChIP 实验关键步骤和需要注意的细节
1、细胞培养及固定
进行一次 ChIP 实验所需的最低细胞数为 105 左右,CST 标准操作方法所需的细胞数为 4 X 106,一般会选择 10 cm 直径的培养皿进行细胞培养(80~90% 汇合率)。为了帮助细胞计数,可以再多培养一皿细胞,进行每一皿细胞数目的估测。
甲醛是常用的细胞固定剂,可造成蛋白质与 DNA 之间的共价交联,并且这种交联是可逆的。务必使用在有效期内、正确避光保存的分析级或分子生物学级别的甲醛(Formaldehyde,甲醛饱和水溶液,浓度 37%),用培养基稀释到 1%(即稀释 37 倍)。固定条件通常为室温固定 10~15 分钟,时间不宜过长,避免固定过度。甲醛的交联反应通过加入甘氨酸溶液终止。
2、细胞核提取和染色质酶切
a)首先,细胞核提取和染色质切割所用的多种缓冲液均需要补加蛋白酶抑制剂(Protease Inhibitor Cocktail,PIC,试剂盒中已提供),且现配现加,新鲜使用。另外,细胞核提取缓冲液中还需要加入 DTT,以保证后续染色质酶切的效率。
b)整个细胞核和染色质的提取过程需要在冰上操作。
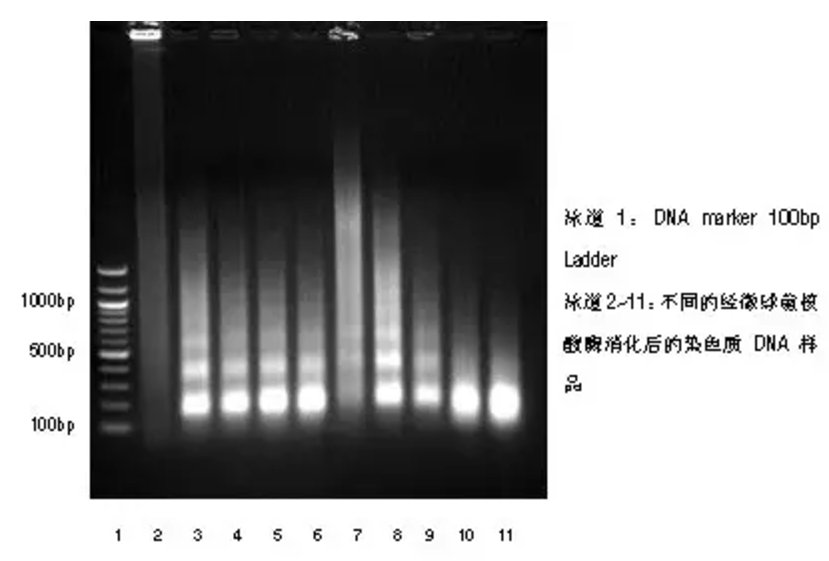
c)染色质酶切后,一般需要对酶切的效率进行判断,方法如下:首先分出 50 ul 的酶切后染色质,将染色质解交联,然后利用纯化柱将解交联后的 DNA 纯化。取 10 μL 纯化后的 DNA 样品,用 1% 琼脂糖凝胶电泳测定核酸片段大小(使用 100 bp DNA ladder 作为 Marker),消化后的 DNA 的理想长度约 150~900 bp(1 至 5 个核小体 DNA 的长度,如下图 4~6 泳道所示),消化过度将只能得到一条带(200bp 左右,如下图 10,11 泳道所示),未充分消化的染色质 DNA 呈弥散状,或在高分子量区域和加样孔内仍然有 DNA 残留(如下图 2,3,7,8,9 泳道所示)。
d)为了保证酶切的最佳效率,通常需要进行滴定法确定微球菌核酸酶与染色质量的最佳比例,具体方法请参考 CST 官方网站的 ChIP 详细操作步骤。
e)合适的染色质 DNA 浓度,对于后续的免疫沉淀和 DNA 检测是非常关键的,因此我们推荐将酶切后的染色质 DNA,取出部分经解交联和纯化后,进行 DNA 浓度测定。具体方法:纯化的 DNA 进行 50 倍稀释,然后测定 OD260 吸收值。DNA 浓度=OD260 吸收值×2500,理想的浓度应在 100~200 μg/ml。(详情请查看 CST ChIP 操作步骤)
3、染色质免疫沉淀
a)所有染色质免疫沉淀使用的缓冲液和洗涤液都需要补加蛋白酶抑制剂(PIC);
b)所有染色质免疫沉淀步骤都必须低温(冰上或 4°C)操作;
c)进行一次标准的染色质免疫沉淀,每一个实验组(如同一药物处理的不同时间点的样品)需要分别设置目的蛋白管、input 样品管、阴性对照管和阳性对照管(最好设置一个)。
目的蛋白管:进行一次标准的染色质免疫沉淀,需要 5~10ug 酶切后的交联染色质(此样品量指染色质中 DNA 的含量,按照上述 2.e 部分的描述进行 DNA 浓度测定后确定),用 ChIP 缓冲液稀释后(通常稀释 4 倍,稀释后的 ChIP 体系不得小于 500ul)进行。首先,从 ChIP 缓冲液稀释后的目的蛋白管中取 2% 体积的染色质作为 input 管样本,不进行免疫沉淀反应,暂时保存在-20 度(等待后续其他样品免疫沉淀步骤完成后,一起进行解交联和 DNA 纯化)。然后,在剩余的目的样品管染色质中加入对应的 ChIP 级抗体,加入的抗体量一般在 1~5 μg,不宜加入过多的 ChIP 抗体,对于 CST 抗体,一般加入 5~10 μl,具体用量请参见产品说明书。
Input 管:每个实验组分出的 Input 样品作为组内参照 DNA,要与经过不同 ChIP 抗体免疫沉淀后捕获的对应 DNA 样本一起完成解交联和 DNA 纯化后,通过测定(如定量 PCR)进行比较,计算 ChIP 沉淀的效率。
阴性对照管:一般可以取 ChIP 缓冲液稀释后的目的蛋白管相同含量的染色质作为阴性对照管样本,然后加入正常同型 IgG 抗体(例如,目的蛋白管所用的 ChIP 抗体如果为兔抗,此时就需要用正常兔 IgG,加入正常兔 IgG 抗体量与目的蛋白的 ChIP 级抗体量相同)。
阳性对照管:一般可以取 ChIP 缓冲液稀释后的目的蛋白管相同含量的染色质作为阳性对照管样本,然后加入与目的蛋白的 ChIP 抗体量相同的 H3 组蛋白抗体(CST 推荐的阳性对照抗体)或者 RNA polymerase II 抗体,进行后续沉淀反应。阳性对照管选择的抗体,常常在整个染色质结合位点丰富,用于检测实验操作是否正确,对于初次进行 ChIP 实验的操作者推荐使用。
上述各样品管加入抗体后,用封口膜封紧管盖,在 4°C 的转子上(可置于翻转摇床上,转速 100~150)孵育 4 小时以上或过夜。
注意:ChIP 抗体的特异性对于 ChIP 结果至关重要,决定了 ChIP 数据的质量和可信度。而 CST 生产的 ChIP 抗体多数是兔单抗,相比传统多克隆抗体(pAb) 和鼠单抗,特异性会大大提高,同时也保持了最高的亲和力,保证了 ChIP 数据的高质量和 ChIP DNA 富集的高效性。相关内部检测数据,请参看 http://www.cst-c.com.cn/resources.html 和相关文章《你看到的组蛋白修饰是不是真的?》。
然后加入 30μl 的 ChIP 级蛋白 G 微珠(强烈推荐使用 CST 的#9006 磁性微珠,便于操作),在 4°C 的转子上孵育 2 小时。注意:琼脂糖微珠要选择 ChIP 专用的、使用 BSA 和鲑精 DNA 封闭后的微珠,CST 提供的产品为 ChIP-Grade Protein G Agarose Beads (blocked with BSA and sonicated salmon sperm DNA) #9007。微珠一般保存在一定的缓冲液中,由于静置时会沉在底部,在吸取前一定要充分摇匀。微珠一般直径较小,建议使用 200μl 的枪头,并将前端 0.5 cm 剪去并用酒精灯烧圆,防止在吸取时刮伤微珠表面。
4、漂洗沉淀后的染色质
充分漂洗对于降低非特异性吸附在微珠上的染色质是非常重要的,因此建议利用低盐和高盐洗涤液多次洗涤。每次洗涤后要将管内的上清尽量吸干,对于磁性微珠就很容易做到。但是琼脂糖微珠由于是通过离心沉于管底,在吸取上清时要格外注意,防止吸走微珠而造成损失。
5、ChIP DNA 的洗脱和纯化
对于 ChIP DNA 的洗脱和纯化,严格按照 CST ChIP 操作步骤进行即可。但注意,在洗脱步骤完成后吸取洗脱上清时要格外注意,防止吸走微珠而造成样品污染。同时要注意每个样品管吸取等量的上清,防止造成样品间误差。
6、利用定量 PCR 对各组 ChIP DNA 进行检测和富集效率分析
ChIP 引物的设计
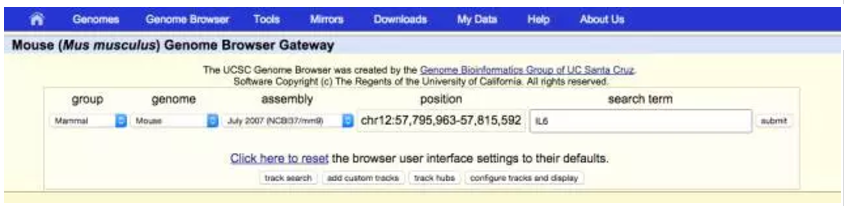
首先要选定某一个基因的启动子区域,可以利用 UCSC genome Browser http://genome.ucsc.edu/cgi-bin/hgGateway 进行分析。在 genome 框中选择种属,如小鼠,然后在对话框内,输入基因名称(如 IL6),如下图所示,
点击 submit,网页跳转后如下图所示,
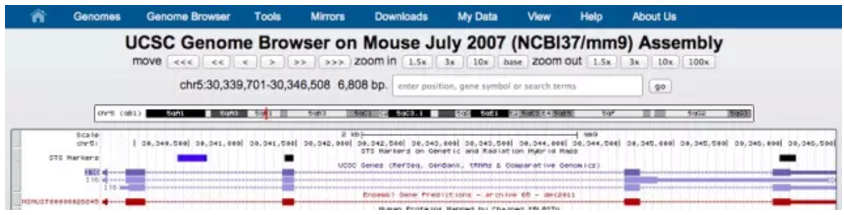
然后,点击 View,弹出下拉菜单后,点击 DNA,
网页跳转后如下图所示,
然后在 Sequence Retrieval Region Options 选项下,Add 后填写入要额外延长的 DNA 长度,(对于位于正链的基因,即从 5』向 3』转录的基因,转录方向箭头向右,向第一个数字框内输入 2000;对于位于负链的基因,即从 3』 向 5』转录的基因,转录方向箭头向左,向第二个数字框内输入 2000;作为一般基因的启动子区域),然后点击网页最下方的 extended case/color options 按钮,网页跳转后如下图所示,
勾选 Mouse mRNA 选项后,点击 submit 按钮按钮,网页跳转后如下图所示,
然后选择划线序列前所有的 2000bp 序列作为启动子区域,利用引物设计软件(如 DNAman、PrimerPremier、NCBI primer design tools 等),进行引物设计。引物要求:Tm 值在 60 度左右,GC 含量 50% 左右,扩增片段 100~200bp,并利用 NCBI 的 BLAST 软件对上下游引物在基因组进行比对检验,初步确定无明显的非特异性匹配。然后,利用定量 PCR 仪,以稀释后的 ChIP input 作为模板,进行预扩增,通过观察熔解曲线确定无明显的非特异性扩增产物,特异的扩增曲线应该是只有一个 PCR 峰。另外,需要将事先分出的 ChIP inputDNA 经过解交联和纯化后获得的精制 DNA 进行梯度稀释(不稀释,1:5,1:25,1:125),再分别扩增,获得不同的 Ct 值,以绘制标准曲线确定引物扩增效率。扩增效率如果较高,此标准曲线应该呈线性。
对于验证某个转录因子在某基因启动子区域的结合,常常需要首先确定转录因子的潜在结合位点,可以通过查阅文献、或用其他转录因子预测的软件(如 TFSEARCH、TRANSFAC、ECR browser 等) 进行预测,然后确保设计的引物的扩增产物能够横跨这个预测的潜在结合位点。而对于组蛋白修饰(如 Tri-Methyl-H3-K9) 的 ChIP 实验,对引物的扩增范围要求则相对宽松,只要扩增产物能够位于启动子区域或基因转录区域即可。
为了方便大家使用,CST 提供很多已经经过验证的检测多个基因的 ChIP primer,请前往 CST 官方网站直接输入 ChIP primer 进行搜索或联系 CST 技术支持(4006-473-287) 获取信息。
对于 ChIP DNA 的定量分析和计算,请参照 CST ChIP 详细操作步骤进行。
需要以解交联和纯化后 2% 的 Input DNA(不稀释)、目的蛋白抗体 IP 后的 DNA、IgG 抗体 IP 后的 DNA 分别作为模板(各取未稀释的 DNA 2ul),分别加入所要检测的目的基因对应的 ChIP 引物,进行定量 PCR 反应,并获得各自的 Ct 值。按照下面公式计算 ChIP 富集效率:
Percent Input = 2% x 2(C[T]2%Input Sample– C[T]IP Sample)而 IgG 抗体 IP 后的 DNA 作为模板进行定量 PCR 反应获得的 Ct 值,作为阴性对照来衡量目的蛋白组的抗体富集是否达到显著差异,是否可以作为可信的结果。通常,当抗体对特定基因位点(例如转录因子结合到目标启动子) 的富集相对于用相同抗体对非特定基因位点(例如相同转录因子结合到非目标启动子) 的富集要高至少 4 倍,相对于用 Normal RabbitIgG #2729 进行的特定基因位点的富集要高至少 5 到 10 倍时,我们定义 ChIP 结果为阳性。阳性 ChIP 实验可低至总 input 染色质的 0.5%(例如转录因子和辅因子),高达总 input 染色质的 40~50%(例如乙酰化和甲基化的组蛋白)。使用 Normal RabbitIgG #2729,我们的磁珠和琼脂糖微珠的背景水平通常在染色质总 input 量的 0.05~0.1%









