万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 提供商:
武汉康测科技有限公司
- 服务名称:
超低起始量Nano UMI MeRIP-seq
超微量MeRIP-seq(Methylated RNA immunoprecipitation with next generation sequencing,甲基化RNA免疫共沉淀结合高通量测序技术)是在转录组层面研究细胞内mRNA以及LncRNA的甲基化,即m6A(腺嘌呤N6位甲基化)定位的技术。
其基本原理直接针对total RNA进行m6A抗体富集,利用残留的rRNA增加建库RNA起始量,并使用UMI 超低起始量建库方式建库,之后剔除rRNA,二次扩增获得最终的文库。经过测序后,测序数据可以在整个转录组层面揭示RNA甲基化—这种转录后RNA表观修饰的定位,整体状况等信息。
二、项目流程
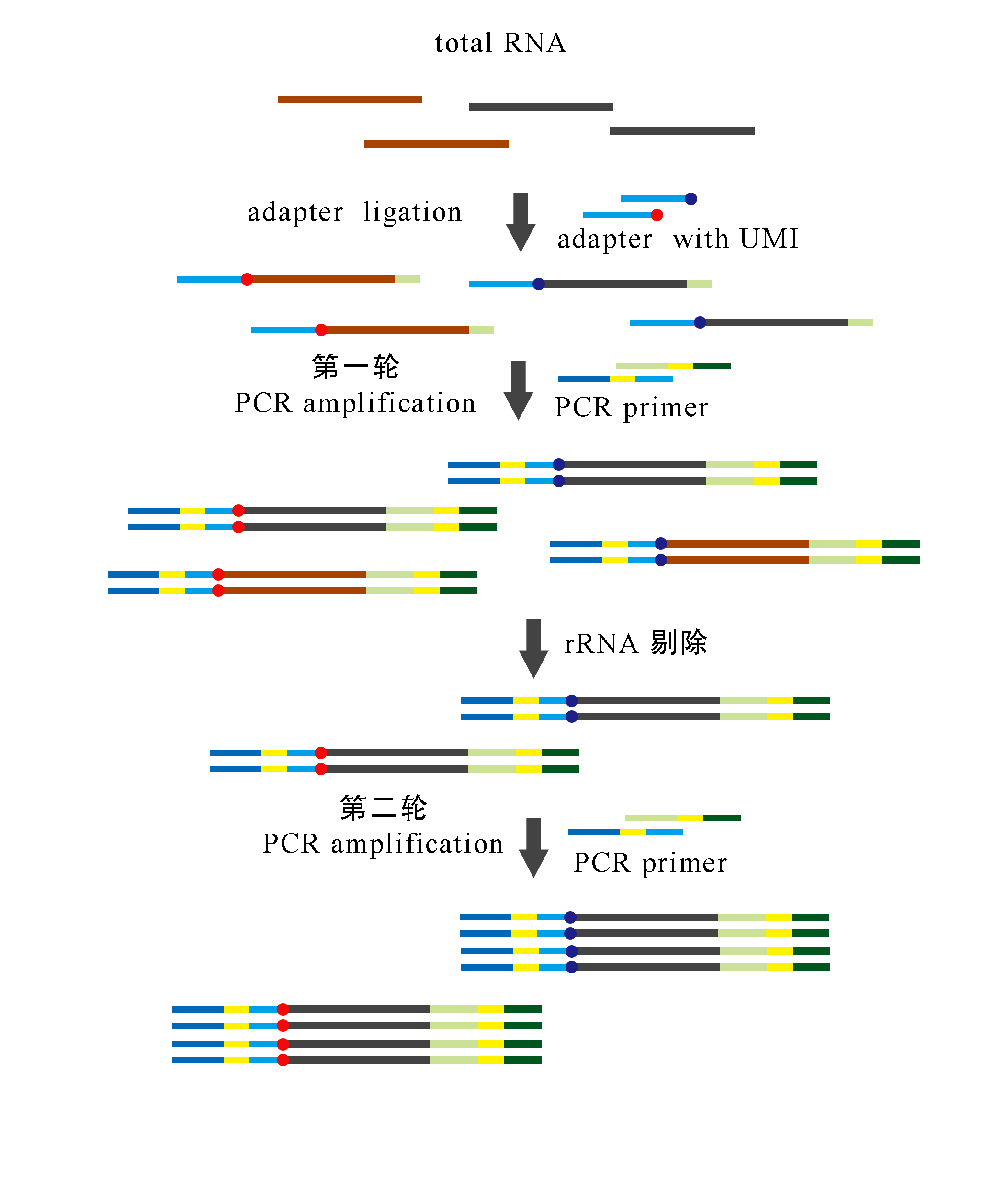
上图:文库构建流程
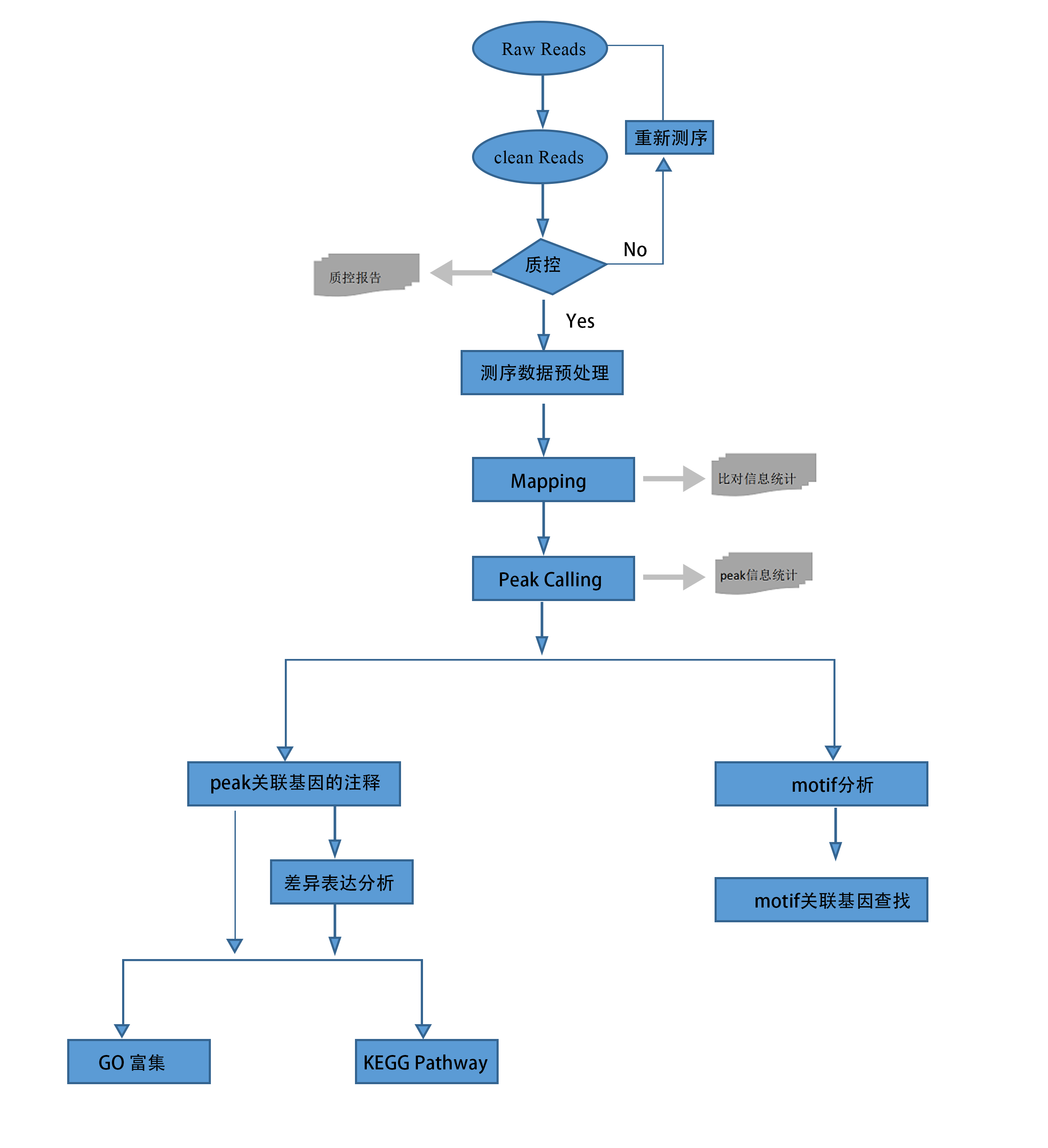
上图:实验分析流程
三、康测科技技术优势总结
1.超低建库起始量,样品准备不再难:建库起始量降低一个数量级,只需要1ug或106个细胞即可建库,样品准备更轻松;使用Novaseq测序,推荐数据量10G,完整项目周期为50个工作日。
2.数字标签还原真实,避免失真结果。
3.设置Input对照,去除背景干扰。
4.丰富项目积累,无惧困难挑战:康测科技已完成上百个IP(ChIP、RIP、CLIP、MeRIP)项目,积累了动植物、微生物近50个物种的IP经验,涉及各种类型的蛋白,如组蛋白、转录因子、解旋酶、剪接因子等,不惧复杂组织挑战。
四、部分结果展示
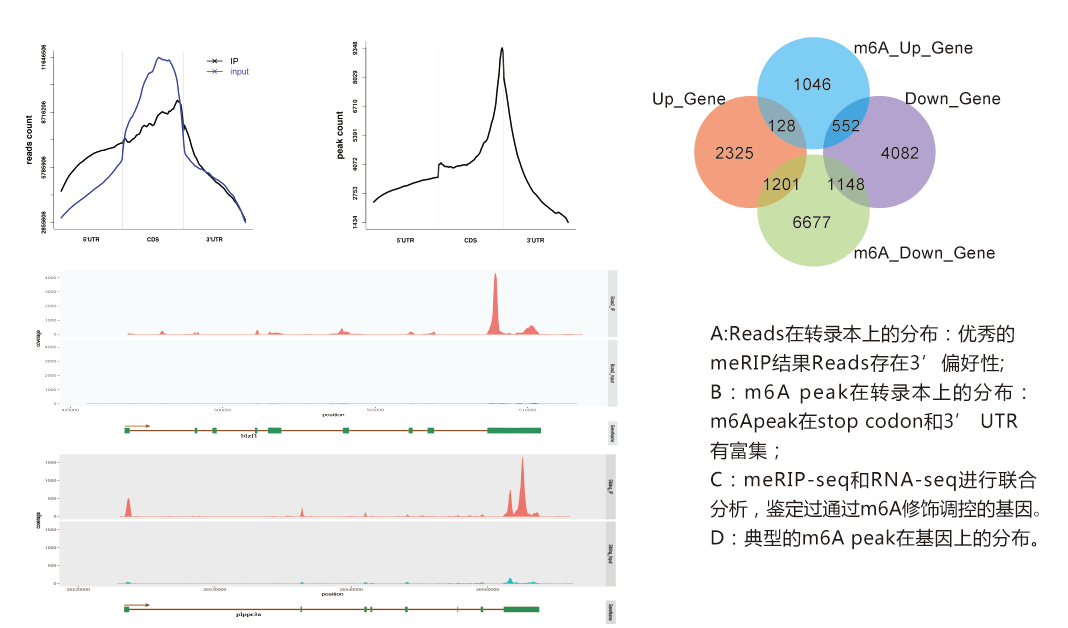
五、案例解读
RNA甲基化m6A修饰调控造血干/祖细胞分化
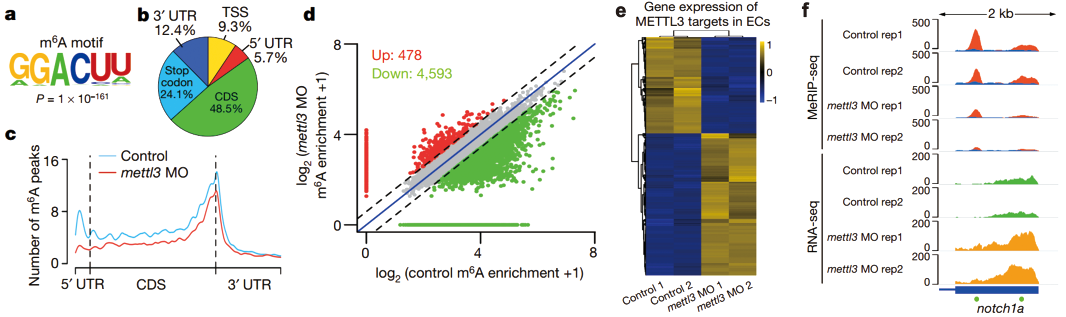
脊椎动物造血干/祖细胞(HSPC)来源于生血内皮,通过内皮-造血转换(EHT)在主动脉血管底部产生。敲除RNA甲基转移酶Mettl3造成胚胎造血功能发育受损,EHT被抑制,内皮细胞不能分化为HSPC。Mettl3敲除后,全基因组的m6A水平均下降(MeRIP-seq结果显示,m6A修饰水平下调基因多达4593个,上调仅478个,m6A修饰水平下调的基因可能为Mettl3的调控靶标,GO富集到RNA处理、胚胎发育相关term(图a-d)。
正常和敲除Mettl3的胚胎的内皮细胞进行RNA-seq,检测到Mettl3敲除胚胎中2393个基因表达上调。关联分析RNA-seq和MeRIP-seq结果,在Mettl3潜在靶标中,680个基因表达显著上调,其中Notch1a是上调最显著的基因之一,同时m6A修饰显著下调(图f),很可能是受m6A调控的HSPC分化关键基因,m6A修饰水平下调,导致m6A介导的mRNA降解途径被抑制、Notch1a表达上调,最终造成造血发育受损。
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验流程如下: 基于 TCR/BCR 检测的 V(D)J 区域靠近 5' 端,故免疫组库测序所用磁珠标签序列由也单细胞最末端的 polydT 序列改为 TSO 序列,构成 read1+barcode+UMI+TSO 的标签组成,用于单细胞的捕获识别和后续 5' 端 RNA 的抓取捕获。 如图, 凝胶珠上的序列中,barcode 序列用于标记细胞,UMI 序列用于标记转录本,而 TSO (template switch oligo) 用于 5’ 端捕获。 当磁珠与细胞结合并在油包水环境中反转
1+barcode+UMI+TSO的标签组成,用于单细胞的捕获识别和后续5'端RNA的抓取捕获。 如图, 凝胶珠上的序列中,barcode序列用于标记细胞,UMI序列用于标记转录本,而TSO (template switch oligo)用于5’端捕获。 当磁珠与细胞结合并在油包水环境中反转成cDNA后,我们对获得的cDNA序列进行扩增,对转录组文库构建方式同单细胞测序实验,免疫组富集文库则需额外设计2次引物binding到C区,对TCR/BCR序列进行富集,后期对富集cDNA随机打断,获得不同
:Buffer、体系、孵育时间、spike-in DNA 量以及建库制备和纯化过程。 图 6:不同细胞量 mESCs 的 uliCUT&RUN 数据结果[4] 将不同细胞量 mESC 的 uliCUT&RUN 数据中的 CTCF 和 H3K4me3 测序结果与已有文献发表的 ChIP-seq 数据进行 read 密度可视化比较,发现 10 个细胞起始量的测序结果可以展现出类似 50 万细胞量的结合位点富集热图。其中 CTCF 的结合区更为狭窄集中,而 H3K4me3 则占据相对宽的区段。 ✦
 技术资料
技术资料暂无技术资料 索取技术资料