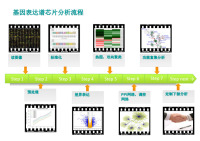
表达谱(芯片数据)基础分析
- 询价
- 上海
- 2025年07月14日
相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 服务名称:
生物信息学分析服务
- 提供商:
上海源兹生物科技有限公司
- 规格:
询价
如需其他分析详见其他产品,下图示例
数据和方法
- 表达谱数据来源
- 原始数据预处理
三、差异表达基因筛选
将基因表达矩阵分为case组和control组,筛选差异表达基因。使用limma[]提供的非配对T检验的方法计算基因表达差异的显著性P值,采用BH矫正P值。对于每个显著差异表达基因,要求P值小于0.05,|logFC|>0.5。使用R软件包pheatmap[]来绘制差异表达基因热图。
三、差异表达基因富集通路分析
使用常用的富集分析工具DAVID[](Version6.8, https://david-d.ncifcrf.gov/)分别分析上调和下调差异表达基因参与的Gene Ontology[]功能富集、KEGG通路[]功能富集。参数富集基因个数count>=2,超几何检验显著性阈值Pvalue<0.05(视为显著富集结果)。四、差异表达基因的PPI网络
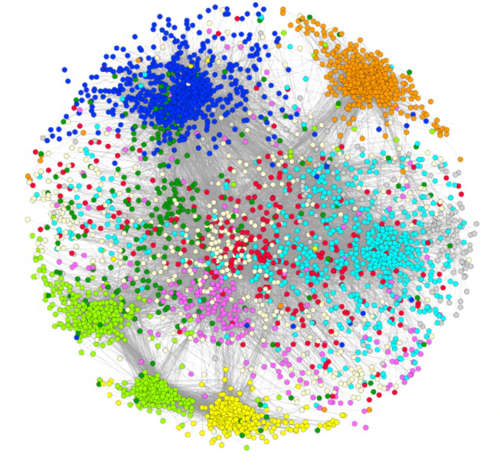
结合STRING((Version:10.0, http://www.string-db.org/))[]数据库预测分析差异表达基因编码蛋白之间是否存在互作关系。输入基因集为差异表达基因,物种为Saccharomyces cerevisiae。参数PPI score设为0.4(表示Medium confidence),要求发生互作的蛋白质节点均为差异表达基因。构建差异表达基因的PPI网络。构建网络软件为Cytoscape(vesrion:3.2.0)[]。利用网络拓扑性质指标Degree Centrality来分析节点在网络中的得分。节点得分越高表示在网络中的位置越重要,越有可能是关键节点。
我们对可能的关键节点,即degreeTOP10的gene在case组和control组的表达水平做条形图。
五、子网络模块
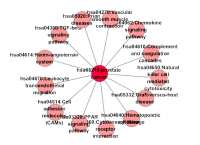
单个的基因往往不会独自发挥作用,它们可能通过与别的对象互作,共同作用下才能发挥调节作用。而同在一个模块中的基因产物蛋白质往往具有相同的或者相似的功能,它们作为一个模块在行使着同一个生物学作用。使用cytoscape的插件MCODE[]方法分析网络中最显著聚类的模块。六、miRNA和疾病靶基因调控网络分析
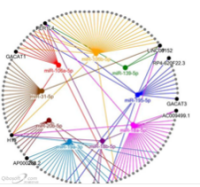
随着RNA干扰机制(RNAi)的发现,非编码RNA在基因表达和调控方面的功能受到了很大的关注,而miRNA因其与生物体的多项调控功能有着密切联系而成为关注重点。miRNA主要通过与其靶基因相互作用进而调控基因的表达。我们通过webgestal[]( http://www.webgestalt.org/option.php), 采用Overrepresentation Enrichment Analysis(ORA)富集方法,分别对上调和下调的差异表达基因进行miRNA-target富集预测,分别获得TOP10的结果,整理得到全部的miRNA-targets关系对,并使用cytoscape软件构建miRNA-target调控网络。
分析结果
- 差异表达分析基本信息统计
- 差异表达基因功能富集结果
表1 上调差异表达基因显著富集的 KEGG Pathway和GO结果
| ID | Name | Count | PValue | |
| KEGG_PATHWAY | sce00230 | Purine metabolism | 6 | 1.65E-02 |
| sce01100 | Metabolic pathways | 18 | 2.29E-02 | |
| sce00330 | Arginine and proline metabolism | 3 | 4.30E-02 | |
| GO_BP | GO:0000096 | sulfur amino acid metabolic process | 3 | 3.47E-03 |
| GO:0000165 | MAPK cascade | 3 | 1.77E-02 | |
| GO:0090630 | activation of GTPase activity | 3 | 2.10E-02 | |
| GO:0071555 | cell wall organization | 6 | 3.31E-02 | |
| GO:0007165 | signal transduction | 5 | 4.39E-02 | |
| GO_CC | GO:0031225 | anchored component of membrane | 6 | 5.68E-03 |
| GO:0005887 | integral component of plasma membrane | 8 | 5.97E-03 | |
| GO:0005886 | plasma membrane | 17 | 1.03E-02 | |
| GO:0000324 | fungal-type vacuole | 12 | 1.15E-02 | |
| GO:0016020 | membrane | 42 | 2.00E-02 |
| Count | <10 | 10—20 | 20—30 | >30 |
| PValue | 0.05-0.01 | 0.01-0.001 | 0.001-1.E-09 | <1.E-09 |
表2 下调差异表达基因显著富集的 KEGG Pathway和GO结果(TOP5)
| ID | Name | Count | PValue | |
| GO_BP | GO:0015891 | siderophore transport | 7 | 2.53E-10 |
| GO:0055085 | transmembrane transport | 20 | 1.26E-09 | |
| GO:0055072 | iron ion homeostasis | 9 | 5.85E-09 | |
| GO:0006811 | ion transport | 14 | 1.54E-08 | |
| GO:0006810 | transport | 31 | 1.02E-06 | |
| GO_CC | GO:0071944 | cell periphery | 30 | 2.89E-18 |
| GO:0005576 | extracellular region | 16 | 3.45E-11 | |
| GO:0005886 | plasma membrane | 28 | 4.07E-10 | |
| GO:0009277 | fungal-type cell wall | 14 | 7.57E-09 | |
| GO:0005618 | cell wall | 11 | 3.95E-08 | |
| GO_MF | GO:0008559 | xenobiotic-transporting ATPase activity | 4 | 3.15E-04 |
| GO:0042626 | ATPase activity, coupled to transmembrane movement of substances | 4 | 4.60E-03 | |
| GO:0005215 | transporter activity | 6 | 6.73E-03 | |
| GO:0005355 | glucose transmembrane transporter activity | 4 | 7.89E-03 | |
| GO:0005351 | sugar:proton symporter activity | 4 | 7.89E-03 |
| Count | <10 | 10—20 | 20—30 | >30 |
| PValue | 0.05-0.01 | 0.01-0.001 | 0.001-1.E-09 | <1.E-09 |
- 差异表达基因PPI网络
表3 PPI网络degree值TOP10差异表达基因列表
| Node | SLT2 | DDR2 | HSP26 | HXT5 | TMA10 | HXK1 | FMP16 | BDH2 | ECM4 | GAP1 |
| Description | up | down | down | down | down | down | down | down | up | up |
| Degree | 26 | 25 | 24 | 23 | 23 | 22 | 21 | 21 | 21 | 20 |
- 网络聚类模块
表4 模块显著富集的GO-BP结果
| ID | Name | Count | PValue | |
| Module a | GO:0034605 | cellular response to heat | 3 | 5.43E-04 |
| GO:0046323 | glucose import | 2 | 3.72E-02 | |
| Module b | GO:0055072 | iron ion homeostasis | 8 | 3.84E-16 |
| GO:0006811 | ion transport | 9 | 3.49E-14 | |
| GO:0015891 | siderophore transport | 6 | 7.10E-14 | |
| GO:0006879 | cellular iron ion homeostasis | 5 | 1.93E-07 | |
| GO:0044718 | siderophore transmembrane transport | 3 | 1.81E-05 | |
| GO:0015677 | copper ion import | 3 | 9.91E-05 | |
| GO:0006810 | transport | 7 | 2.21E-04 | |
| GO:0006826 | iron ion transport | 2 | 1.57E-02 | |
| GO:0055085 | transmembrane transport | 3 | 4.38E-02 |
- miRNA-target调控网络
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验pianpianqiwu812 求助各位老师,近期我做了6张miRNA芯片,实验组3张,对照组3张,公司给我的数据只有相对值,没有P值。原因是芯片数量少,不需要做统计学分析。这种说法是否对?还有就是在计算差异倍数时是用实验组相对值分别除以对照组的相对值,倍数在2倍以上的入选。这样计算是否对,还是倍数计算也是要有相关软件的?我在这方面真是只菜鸟,不懂的很多,公司说的我感到有些可疑。:( zhujoker 这个你在做的时候就应该
刚才在网上看到的,不知道是不是有人发过了。 网址:http://www.csc.fi/oppaat/siru/ 下载地址:http://www.csc.fi/oppaat/siru/siruwww.pdf 分部分下载地址: Part I http://www.csc.fi/oppaat/siru/sirupartI.pdf PartII http://www.csc.fi/oppaat/siru/sirupartII.pdf PartIII http://www
今天给大家介绍一种简单快速的基因芯片数据分析网站GCBI 基因云平台(www.gcbi.com.cn),可以让科研人员及医生低门槛并且快速的使用生物信息学进行基因相关的研究。 分析思路 先以表达谱数据为例介绍基本的分析思路: 1. 差异基因筛选: 对实验组和对照组进行差异基因筛选,筛选出与实验因素(可以是疾病、干扰或过表达等)相关的差异表达基因。 2. 差异基因功能与通路分析: 对差异基因基于 GeneOntology 和 KEGG 数据库进行显著
 技术资料
技术资料暂无技术资料 索取技术资料