相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 询价记录
- 文献和实验
- 技术资料
- 服务名称:
全基因组重测序
- 提供商:
上海英拜生物科技有限公司
技术优势:
· 多种变异检测:单核苷酸多态行(SNP)、插入缺失(InDel)和结构变异(SV);
· 与芯片方法比较,可以检测到新的变异序列;
· 与人类全基因组从头测序相比,耗时更短、成本更低。
研究内容:
一、 数据产出统计
基因组单碱基深度分布及覆盖度分析
二、 一致性序列组装
根据与参考基因组序列的比对结果,利用贝叶斯统计模型检测出测序个体基因组中每个碱基位点最大可能性的基因型,并组装出该个体基因组的一致序列。
三、SNP检测及在基因组的分布
在全基因组一致性序列的基础上,从中提取全基因组中所有的潜在多态性位点,再根据质量值、深度、重复性等因素做进一步的过滤筛选,最终得到高可信度的SNP数据集。根据已有的基因集对检测到得变异进行注释。
四、InDel检测及在基因组的分布
使用测序个体的短序列与参考基因组进行Paired-End比对,将比对结果进行聚类分析,并结合Paired-End关系检测可信的short InDel。在检测过程中,gap的长度为1~3个碱基。对于每个InDel的检测,至少需要3个双末端序列的支持。
五、结构变异检测及在基因组中的分布
利用测序个体序列与参考基因组序列进行比对,检测全基因组水平的结构变异,目前能够检测到得结构变异类型主要有缺失、插入、复制、倒位、易位等,并根据已有的基因集对检测到得变异进行注释。
*具体包括:
1. 标准信息分析
i. 测序数据基本分析 ;
ii. 比对;
iii. 产出数据统计;
iv. 一致性序列生成;
v. SNP检测、注释及统计;
2. 动植物个体高级信息分析
i. InDel检测、注释及统计;
ii. SV检测、注释及统计;
3. 动植物群体高级信息分析
i. 连锁不平衡分析(LD);
ii. 群体进化树分析(Phylogeny tree);
iii. 群体结构分析(Structure);
iv. 群体主要成分分析(PCA);
v. 多态性分析 ;
vi. InDel检测、InDel注释及统计(群体Indel注释);
vii. SV检测、注释及统计;
4. 人个体高级信息分析
i. InDel 检测、注释及统计;
ii. SV检测、注释及统计;
iii. CNV检测、注释及统计;
iv. 根据已有数据库的信息对表型或疾病风险进行评估;
v. 血统分析;
5. 人群体高级信息分析
i. InDel 检测、注释及统计;
ii. CNV检测、注释及统计(15X/个体以上可选);
iii. 群体SNP检测;
iv. 无偏群体频谱估计(基于群体SNPs);
v. 群体InDel检测;
vi. 单体型分析:连锁不平衡,单体型块预测等 ;
vii. Demographic 分析(分析风险高):分歧年代预测,人口迁移预测等 ;
viii. Population structure and phylogenetics:群体结构分析,系统发育树,主成分分析;
ix. 选择信号分析(有风险):Tajima’D,Fst,PBS分析等;
6. 癌症研究的高级信息分析
i. 成对的样本(normal-tumor)SNP/InDel/SV/SNV/CNV检测、注释及统计 ;
ii. 氨基酸置换预测;
iii. Pathway,GO富集分析;
iv. 筛选出的突变与已有数据库比较(cosmic,dbSNP等);
v. 病毒插入序列检测 ;
vi. 重排检测及注释;
vii. 选择压力检测以识别出driver gene;
viii. Mutation target network ;
7. 复杂疾病研究的高级信息分析
i. 基于低深度大样本的关联分析(群体SNP检测,群体MAF估计,关联分析);
ii. 基于PLINK软件的高深度大样本或者合理选取小样本量的关联分析;
iii. 基于家系样本的de novo mutation检测;
8. 单基因病研究的高级信息分析
i. 性别判断;
ii. InDel识别、注释、统计(选择此项,比对软件使用BWA);
iii. SV检测、注释及统计 ;
iv. CNV检测、注释及统计 ;
v. 筛选非编码区突变并进行数据库比较;
vi. Case共有的突变筛选 ;
vii. HMM预测 ;
viii. SIFT保守性预测 ;
ix. 筛选出的基因GO、KEGG注释;
x. CNV与DGV数据库的比较;
9. 定制化信息分析
i. 可结合客户的需求,协商确定定制化信息分析服务内容。
ii. 癌症全基因组重测序高级信息分析(基于标准信息分析)
案例:应用全基因组测序和RNA测序来描绘常见的变异型免疫缺陷综合症(CVIDs)的基因图谱
背景:常见的变异型免疫缺陷综合症(CVIDs)是机体免疫应答反应中不能产生抗体的最主要原因。CVIDs变异度很高,大概5%的病人是由基因改变引起的。
目的:利用Illumina HiSeq2500和HTSeq测序平台对CVIDs进行全基因组及转录水平的研究,揭示并绘制CVIDs的信号调控图谱。
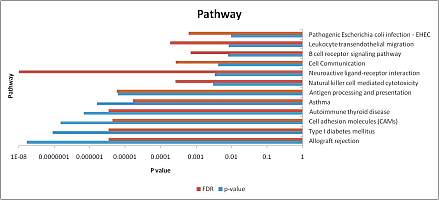
结果:通过对CVIDs基因组和转录组进行测序和综合分析发现, TNFRSF13B, TNFRSF13C, LRBA and NLRP12等基因在CVIDs存在突变,这些突变的基因与B细胞受体信号通路,非同源末端连接修复,细胞凋亡,T细胞调控及ICOS信号通路的调控有关。该研究揭示了新的CVIDs相关的信号通路。
原文索引:
Pauline A. van Schouwenburg, Emma E. Davenport (2015) Application of whole genome and RNA sequencing to investigate the genomic landscape of common variable immunodeficiency disorders. Clinical Immunology, 160 (2), 301–314

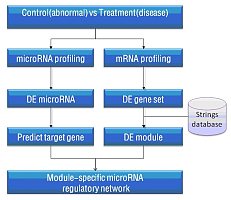
图1:CIVD病人与正常人的差异表达基因分析

图2: CIVD病人间的变异基因overlap图
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
- 作者
- 内容
- 询问日期
 文献和实验
文献和实验molrep 有哪位战友知道怎么做全基因组的基因knock-down,我听说有全基因组的shRNA文库,怎么用这个东西呢,谢谢 bigbang_0_0 做转染,然后看你们所期待的表型有什么变化即可 freecell 何不参考一下这篇文献? http://www.sciencedirect.com/science?_ob=ArticleURL&_udi=B8G3Y-4TT80P4
海边海鸥 偶的题目大致是研究某一细菌产生ESBLs,其基因组中含有耐药基因,但是表型(药敏试验仍对该类抗生素敏感)阴性。可能存在某些机制使该耐药基因未表达。打算进行该两株的全基因组测序,想请教一下主要从哪几方面考虑未表达的可能性。谢谢大家:) 海边海鸥 怎么没有建议呢? zhujoker 我来胡说2句吧,不知道细菌基因组有没有甲基化修饰,自己不做细菌,所以不能确定(但是偶刚才查一下的确好像
相关专题 DNA甲基化是基因组 DNA的一种主要表观遗传修饰形式。近年来,大量研究表明DNA甲基化修饰对于维持正常细胞功能、传递基因组遗传印记、胚胎发育以及人类肿瘤发生,起着至关重要的作用。甲基化研究成为表观遗传学的热点,相应的技术也是层出不穷,根据实验目的的不同,这些技术大体能够分成两类:全基因组(高通量)甲基化研究技术以及特异性甲基化位点(低通量)研究技术。下面将介绍几种当前最常用的一些技术全基因组或者说中高通量甲基化研究手段,供大家参考