相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
—————————————————————
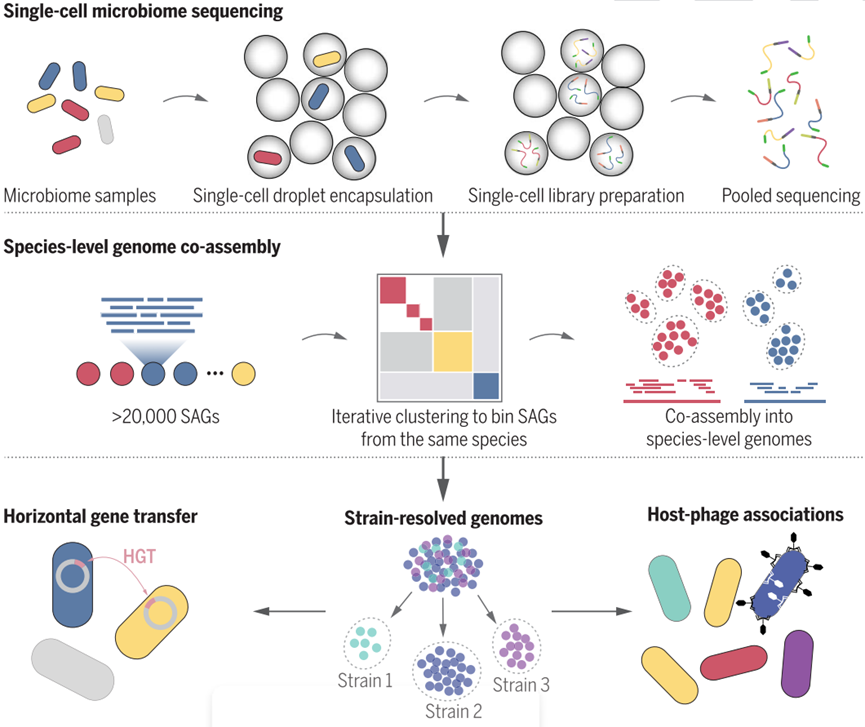
带领微生物研究进入高通量单细胞时代
Microbe-seq是一种具有菌株分辨率的高通量单细胞测序方法。Microbe-seq技术集成了多种液滴微流控操作技术和定制开发的生物信息学分析手段,可应用于其他复杂的微生物群落,例如土壤和海洋中的微生物群落等。
微生物群落栖息于许多自然生态系统中,包括海洋、土壤和动物的消化道。微生物群落的行为和生物效应不仅取决于其组成,还取决于每个微生物群落内发生的生化过程以及它们之间的相互作用;这些过程强烈地受到生活在该群落中的每个微生物的基因组的影响。至今想要阐明微生物群落中的菌株水平信息,还面临相当大的挑战,如果能从微生物群落中解析出菌株水平的高质量基因组,那么这将极大地帮助我们理解微生物行为及其对宿主的影响。
目前,宏基因组测序技术被广泛应用到探索复杂微生物群落,但宏基因组学通常无法解析菌株水平的基因组。相反,基于培养的方法和基于滴度板的单细胞测序可以产生菌株解析的基因组,但只能获得有限数量的微生物菌株,且成本偏高。
Microbe-seq技术的发明解决了以上难题,它不需要培养即可从复杂微生物群落中获取成千上万个单细胞微生物的基因组信息,并组装出高质量的菌株水平基因组,从而能够在不损失分辨率或广泛物种适用性的基础上探究微生物群落的基因组。该方法应用面广泛,可用于具有复杂微生物群落的样本,如粪便、土壤和海洋等,在微生态研究中具有极大的市场应用潜力。
【技术原理】
实验原理:
1、利用液滴微流控技术,将成千上万的微生物单独地包裹在液滴中;
2、在每个液滴中,裂解微生物并释放DNA;
3、进行全基因组扩增,将扩增出来的DNA打断并连上接头,随后在液滴中用带有特定序列的标签标记液滴内DNA;
4、将所有液滴内的DNA合并,进行建库测序。
生信分析原理:
1、通过提取并比对各单细胞的基因组标志性信息,将样品中来自同一物种的单细胞微生物识别出来,然后合并在一起来组装出物种水平的参考基因组;
2、进一步将单个微生物的基因组和参考基因组对比,识别来自于不同菌株的单细胞微生物并进行基因组组装。
【技术优势】
- 高通量检测:一次可以获得成千上万单一微生物基因组,且无需培养
- 新物种、新菌株:发现新物种、新菌株,弥补宏基因组分辨不足的短板
- 基因组组装质量好:基因组质量与分离培养相同,支持功能基因/代谢通路研究
- 挖掘军逐渐关系:分析水平基因转移,研究宿主-噬菌体关联
【应用领域】
- 高精准度解析微生物群落结构
- 发掘有潜在应用价值的微生物资源
- 深入研究种间/种内的互相关系
- 深入研究病原菌的治病分子机制
- 单细胞水平解析耐药基因分布特征
- 改善复杂微生物组的参考基因组数据库
【技术参数】
| 产品类型 | 样本要求 | 测序策略 | 数据要求 |
|---|---|---|---|
| 微生物高通量单细胞基因组测序 |
|
Illumina 2×150 | 每个样本: 6000 cells, 150G raw |
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验吸烟越多,DNA 修复能力却可能更强?最新研究揭示为什么大多数吸烟者不会患肺癌
平稳可能源于这些人拥有更熟练的修复 DNA 损伤或解毒香烟烟雾的能力。 图片来源:Nature Genetics 为了识别体细胞突变并准确评估其在正常组织中的频率,该团队在 2017 年开发了一种改进的单个细胞基因组测序方法,即 SCMDA。正如 Nature Methods 杂志所报道的那样,相比于传统单细胞全基因组测序方法,这种方法能够精确定量单细胞全基因组分析中的突变,同时避免了长期的克隆扩增和额外突变的引入。 图片来源:Nature Methods 主要研究内容 研究整体
革新技术!谢晓亮院士 PNAS 重磅推出迄今为止最精准的单细胞 SNV 检测方法
背景介绍在过去的十年里,单细胞测序技术的蓬勃发展深刻地扩展了我们对于基础生物学的理解。目前,单细胞转录组测序在一次实验中就可以检测数万个细胞,在解析细胞异质性和鉴定新型细胞亚群方面具有独特的优势。 虽然说单细胞基因组测序亦可为细胞间异质性和基因组不稳定性等问题提供了新的视角,对于生物学和医学同样非常很重要,不过,当前的单细胞基因组测序主要依赖全基因组扩增的方法,而这种方法的普及目前仍被拷贝数变异检测的低精度、低保真度以及测序成本所阻碍。 单细胞中单核苷酸变异 (SNVs) 的测定一直存在假阳
GUT:国家癌症中心崔巍联合多组学技术团队发表血清代谢组肠癌早筛新机制
:四个研究队列(发现队列、建模队列、验证队列、血清和粪便匹配队列),每个队列中均有不同数量的正常人群(蓝色)、腺瘤(红色)和 CRC 患者(绿色)样本。 2、非靶代谢组分析:血清和粪便样本中提取的代谢物,进行 LC-MS 非靶代谢分析 3、靶向代谢物分析:血清中游离胆汁酸和脱氧胆汁酸 4、宏基因组测序和分类:提取粪便样本 DNA,使用鸟枪法进行宏基因组测序,并进行肠道微生物组的分类和功能分析。 5、空间代谢组分析:9 对人类结直肠组织样本(包括晚期腺瘤或 CRC 以及癌旁组织)进行 AFADESI









