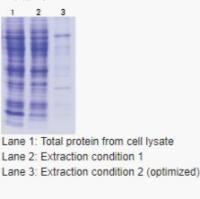
3 个回答
Miracle星
有帮助
不可以的
简单的说:1.要遵循引物设计的基本原则,,重组表达的引物相对容易设计一点(因为位置固定);
2.目的产物是:蛋白1+linker+蛋白2,则需插入载体的目的片段为“酶切位点1+蛋白1+linker+蛋白2+酶切位点2”
3.那么想获得这个目的基因片段,则需要设计4个引物,分两步进行。
1)引物:P1——> 5'- 内切酶1+蛋白1上游 -3' (正向序列)
P2——> 5'- linker+蛋白1下游 -3' (反向互补序列)
P3——> 5'- linker+蛋白2上游 -3' (正向序列)
P4——> 5'- 内切酶2+蛋白2下游 -3' (反向互补序列)
2)首先以 蛋白1的模板+P1、P2 ——> 获得 蛋白1+linker 片段;
蛋白2的模板+P3、P4 ——> 获得 linker +蛋白2 片段;
3)第二部回收两种片段的PCR产物后,混合作为模板,以P1、P4做引物,PCR即可得到目的片段
这就是传说中的重叠延伸 PCR(splicing by overlap extension-PCR,SOE-PCR
bamboopiggy
有帮助
不能,linker序列和酶切位点是不一样的,酶切位点没法转弯
汤姆卜丽波
有帮助
不可以的,应该说最好不要,没有Linker的功能区域直接融合可能会导致许多不良的结果,包括融合蛋白的错误折叠、蛋白产量低或生物活性受损等
相关产品推荐
相关问答
提问
扫一扫

实验小助手

扫码领资料
反馈
TOP
打开小程序