生信小课堂 | 一文了解ORA、GSEA、ssGSEA !
上海吉凯基因医学科技股份有限公司
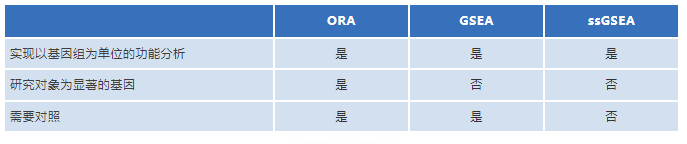
在某一项研究中,我们可以通过测序或质谱获得实验组和对照组序列信息,进而获得基因表达量。但生物功能的执行往往涉及多个基因或蛋白的相互作用,实验者通常更希望在分子途径或其他功能相关的基因分组的层面上提出问题,仅对基因功能进行描述远无法满足人们的需求。在此需求的基础上,ORA、GSEA、ssGSEA应运而生,但这三者在应用范围(Table1)原理等方面有所不同,接下来,小编会对三者的原理和结果进行一个简单介绍。
Table1. ORA、GSEA、ssGSEA比较
过代表分析
过代表分析(Over-Representation Analysis, ORA)是在一组显著表达的基因或代谢物中,推断出在该数据集中受干扰的生物途径或过程,以及在该途径或过程中起作用的基因或代谢物。这里的生物途径或过程可以是多种,例如Gene Ontology(GO) 描述的功能(biological function),KEGG或Reactome描述的通路 (pathway),即非来自数据本身的任何基因分组,具体取决于使用者感兴趣的方向。此外,ORA应用范围很广,在转录组学、代谢组学等数据集中均可使用。
ORA原理
ORA最常见的方法是在显著表达的基因中,检验某些由基因或代谢物组成的生物途径或过程是否被过代表 (over-represented)。该过程通过使用统计上的超几何分布实现。
ORA通过g:Profile的实现
g:Profile是一个可执行包括ORA功能在内的网页( Fig.1),可选择感兴趣的数据集,自定义临界值等,并可对结果进行可视化(Fig.2)。其输出是一个交互式的曼哈顿图,表明ORA富集结果。X轴代表不同的基因集,Y轴表示调整后的富集度p值,每一个圆圈代表一个基因集,如果将鼠标悬停在圆圈上,将会显示该基因集的名称及对应的p值,浅色的圆圈代表不显著。点击一个圆圈,就会把这个圆圈固定下来,并在图的下面创建一个结果表(Fig.3),然后在表格中显示详细信息,如数据源、术语的id和名称以及相应的p值。
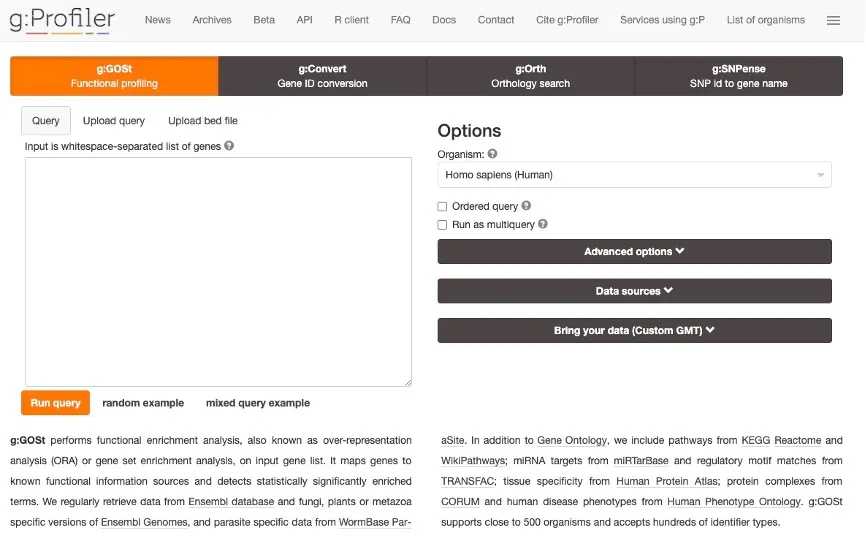
Fig.1 g:Profile网页版
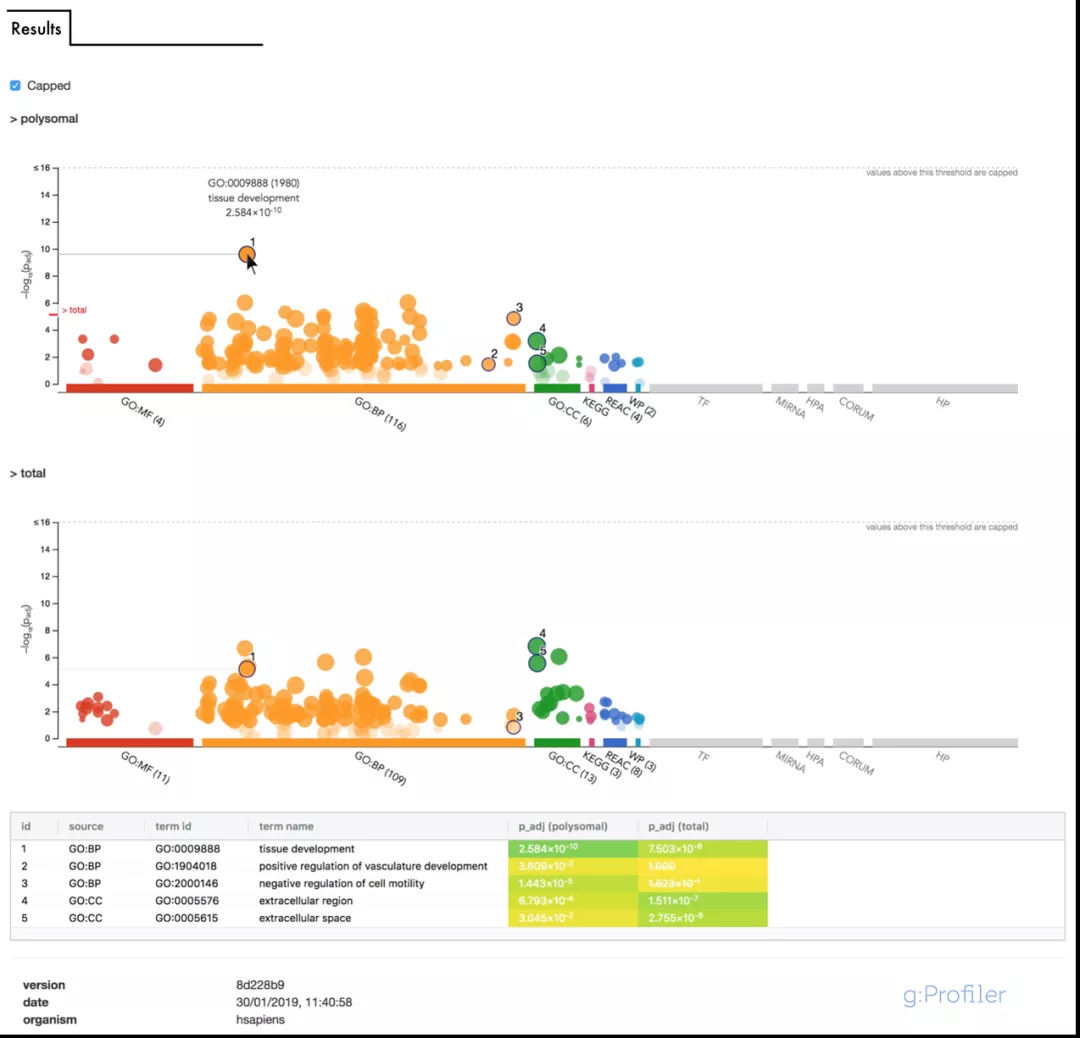
Fig.2 g:Profile分析结果
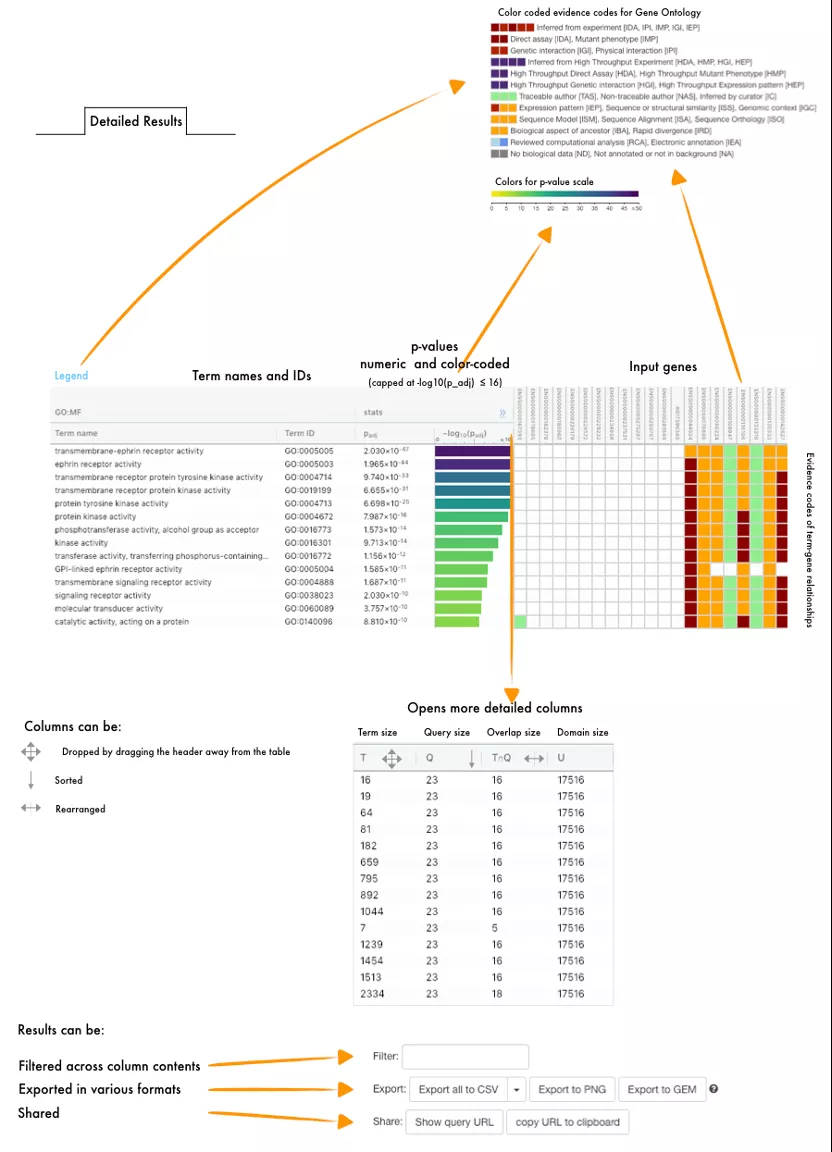
Fig.3 g:Profile详细结果
基因组富集分析
尽管ORA可以用来解释生物过程,但其计算过程仍有一定缺陷,如许多基因表达是相关的,假设基因间独立是不可能的;ORA需要在显著表达的基因或代谢物中进行过代表分析,变化小但作用大的基因可能被遗漏等。为了克服这些问题,Subramanian等人开发出了基因组富集分析(Gene Set Enrichment Analysis, GSEA)。
GSEA原理
Subramanian等人开发出名为GSEA-P的package实现GSEA。其原理为先利用表达数据计算基因在AB组的差异,然后按差异排序,如Fig. 3A,该基因排序顶端比较体现A组特征,底端比较体现B组特征。然后检验感兴趣的基因集(如编码代谢途径中的产物的基因)是随机分布在排列好的基因上,还是更集中在某一端,与表型区别相关的基因集倾向于分布在一端。如在Fig. 3A中基因集S大部分集中在基因列表顶部,则该基因集在A组富集。
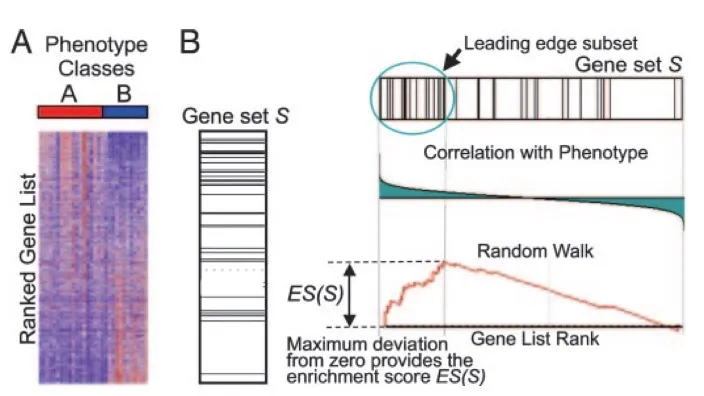
Fig.4 GSEA原理
MsigDB数据库
MsigDB是Subramanian等人为了GSEA创建的数据库,从位置、功能、代谢途径、靶标结合等多种角度出发,构建出一系列具有功能的数据集。这些基因集被整理至http://www.gsea-msigdb.org/gsea/msigdb/index.jsp,该网站提供检索,下载等多种功能。对基因集名称、描述、分类等均有详细描述。
Fig.5 MsigDB数据库
GSEA rank plot结果解读
GSEA rank plot是GSEA结果中最重要的图,针对每一个感兴趣的基因集,有一张这样的GSEA rank plot。分成三部分,顶部是Enrichment Score(ES score)的折线图,横轴是排序后的基因,纵轴是对应的Running ES,折线图有一个峰值,该峰值为该基因集的ES score,绝对值越高,富集程度越高。
中间部分是基因位置图,黑线代表感兴趣的基因集中的基因处于当前所有基因排序后的位置,红蓝相间的热图是表达丰度排列,颜色越深差异越大。
底部的图显示了随着排序基因下移,排名指标的值,即所有基因的排序依据,默认使用signal-to-noises的比值。
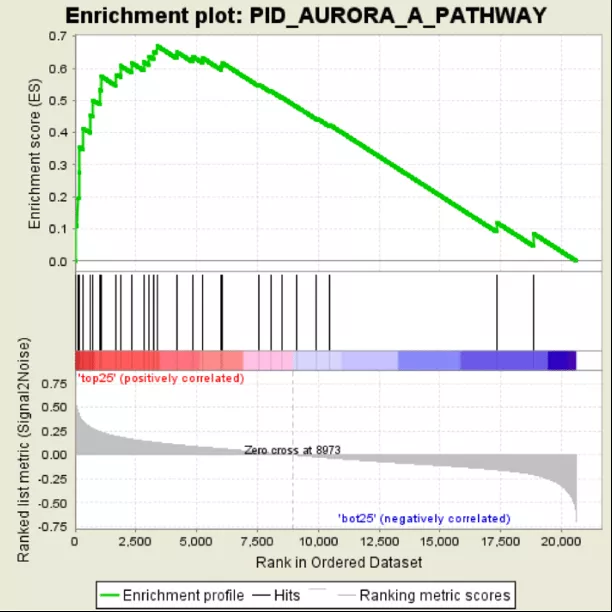
Fig.6 GSEA rank plot
ssGSEA
单样本GSEA(Single-sample GSEA, ssGSEA)是GSEA的延伸,针对单样本无法做GSEA而提出的一种实现方法,每个ssGSEA富集分数代表了特定基因组中的基因在一个样本中协调性地上调或下调的程度。ssGSEA的原理是,对感兴趣的基因组中的基因进行打分,汇总得到的值即为该基因组的ES分值,然后通过假设测验判定该基因集是否富集。
以上即为ORA、GSEA、ssGSEA三种方法的联系与差异,通过本篇文章,希望能帮助大家区分三个易混淆的概念。












