经典遗传学实验
相关专题 基因芯片技术的诞生为生物技术工作人员打开了一道科研的便利之门,曾被评为1998年年度十大科技进展之一。本文对基因芯片的实验原理、技术基础、分类、用途、操作主要环节等内容做详细的介绍。 1.基本原理和技术基础 基因芯片以DNA杂交为基本原理,基于A和T、G和C的互补关系。它是在探针的基础上研制出的。所谓探针是一段人工合成或筛选出的已 ...
相关专题 蛋白酶K,是一种切割活性较广的丝氨酸蛋白酶。它切割脂族氨基酸和芳香族氨基酸的羧基端肽键。此酶经纯化去除了RNA酶和DNA酶活性。由于蛋白酶K在尿素和SDS中稳定,还具有降解天然蛋白质的能力,因而它应用很广泛,包括制备脉冲电泳的染色体DNA,蛋白质印迹以及去除DNA和RNA制备中的核酸酶。蛋白酶K的一般工作浓度是50—100μ ...
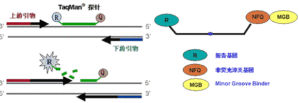
相关专题 实验室很多同学都要做Real time PCR实验,实验室的师兄师姐都会有很多宝贵意见,不过也有实验室前没有做过的,查找了下资料和大家分享下关于实时荧光Taqman 探针设计、实时荧光PCR探针的选择、引物的设计及评价。 荧光探针法是用序列特异的荧光标记探针来检测产物,探针法的出现使得定量PCR技术的特异性比常规PCR技术大大提高 ...
相关专题 慢病毒包装技术专题 一、慢病毒转染贴壁细胞实验方法 1、慢病毒转染前18-24小时,将贴壁细胞以1×10^5/孔铺到24孔板中。使细胞在慢病毒转染时的数量为2×10^5/孔左右。 2、第二天,用含有6 μg/ml polybrene的2ml新鲜培养基替换原培养基,加入适量病毒悬液。37℃孵育。 3、(对polybrene毒性敏 ...
相关专题 慢病毒包装技术专题 慢病毒包装简要步骤: 以六孔板中的1孔为例,每个样品需要1×106个293T细胞。 1、取1.5ml灭菌EP管,加入1.5μg包装混合质粒和0.5μg表达质粒以及250μl的无血清培养基。轻柔混匀,室温孵育5min。 2、取1.5ml灭菌EP管,取9μl 脂质体2000l溶于250μ ...
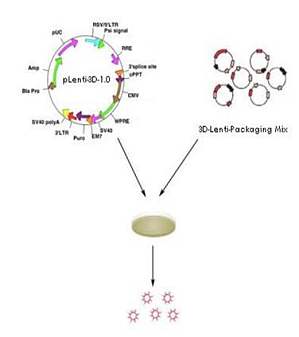
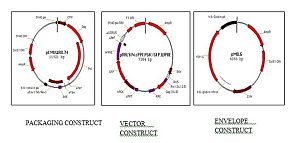
相关专题 慢病毒包装技术专题 现今常用的制备慢病毒载体的方法为使用3或者4质粒系统转染293T细胞。此外,也有使用其它几类慢病毒包装细胞系制备慢病毒载体。 瞬时转染制备慢病毒: 细胞:慢病毒包装常用人胚肾细胞(HEK human embryonic kidney)293T,其含有SV40病毒的大T抗原蛋白编码基因,转染效率极高。但其贴壁性不好 ...
相关专题 慢病毒包装技术专题 慢病毒(Lentiviruses)属于逆转录病毒科。慢病毒核蛋白质前整合复合物具有噬核特性,病毒基因组运输至细胞核,从而使慢病毒可以感染和在非有丝分裂细胞中复制。这一特性使慢病毒成为基因治疗的转移载体。 HIV (Human immunodeficiency virus) 、EIAV (Equine infect ...
相关专题 慢病毒包装技术专题 RNA干扰靶序列设计的方法最初由Elbashir et.al 2001发表。之后由于发现了RISC与siRNA结合的偏好,设计干扰效果优秀的siRNA的成功率大大增加。我们主要采用Pei and Tuschl(N2N8N8N2)发表的设计原则进行设计。对初步筛选出的所有符合条件的序列进行siRNA正义和反义链自由 ...
相关专题 慢病毒包装技术专题 «介绍一下贵公司的慢病毒载体,有何特点? 本公司的慢病毒载体是在第三代慢病毒载体系统的基础上优化而成的,经过优化,我们的载体滴度高,安全性好,转染高效。 «慢病毒如何储存? 客户在收到慢病毒液后,如果在很短的时间内就开展实验的话,可以将病毒置于4℃保存;如果长期保存可将病毒装入冻存管中置于-80℃冰箱(注意:病毒 ...
相关专题 一、慢病毒转染贴壁细胞实验方法 1、慢病毒转染前18-24小时,将贴壁细胞以1×10^5/孔铺到24孔板中。使细胞在慢病毒转染时的数量为2×10^5/孔左右。 2、第二天,用含有6 μg/ml polybrene的2ml新鲜培养基替换原培养基,加入适量病毒悬液。37℃孵育。 3、(对polybrene毒性敏感的细胞选作此步骤 ...
相关专题 本方法的核心部分是医科院基础所的生化脂蛋白组的吴刚老师所创,在其基础上进行了一些改动,主要是DNA回收上采取了更简单的方法。(本方法最适用于双酶切制作片断并进行克隆的情况。对于分步酶切制作片断,也可以使用本方法,但需要加倍起始酶切DNA的量。) 一、片断平移的克隆(也适用于多片断连接) 简介:将用作载体的质粒A(制作大片断)酶切3 ...
相关专题 FISH的特点:操作烦琐,步骤简单。说它操作烦琐,是因为它的操作步骤烦多,每一步又需要相当长的时间,如果自己标探针的话每批标本大约三天时间(买试剂盒只需一天,但价格昂贵),你说是不是很烦琐?说它步骤简单也是有道理的。FISH其实并没什么神秘之处,只要操作过程仔细,严格按照操作步骤操作,很容易做成功,比PCR要简单得多,所以说它是步 ...
相关专题 1 甲基化敏感性限制性内切酶(methylation-sensitive restriction Endonuclease,MS-RE)-PCR/Southern法 这种方法利用甲基化敏感性限制性内切酶对甲基化区的不切割的特性,将DNA消化为不同大小的片段后再进行分析。常使用的甲基化敏感的限制性内切酶有HpaⅡ-MspⅠ(识别序列 ...
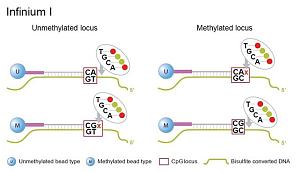
相关专题 DNA甲基化是基因组DNA的一种主要表观遗传修饰形式。近年来,大量研究表明DNA甲基化修饰对于维持正常细胞功能、传递基因组遗传印记、胚胎发育以及人类肿瘤发生,起着至关重要的作用。甲基化研究成为表观遗传学的热点,相应的技术也是层出不穷,根据实验目的的不同,这些技术大体能够分成两类:全基因组(高通量)甲基化研究技术以及特异性甲基化位点 ...
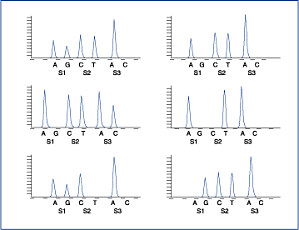
相关专题 近年来涌现出不少DNA甲基化的检测技术,少说也有十几种。大致可以分为两类:特异位点的甲基化检测和全基因组的甲基化分析,后者也称为甲基化图谱分析(methylation profiling)。下面大家介绍一些常用的方法。 特异位点的甲基化检测 甲基化特异性PCR(MS-PCR) 这种方法经济实用,无需特殊仪器,因此是目前应用最为广泛 ...
相关专题 G418 :白色粉末,融点138~144℃,溶于水、甲醇。 CAS No.:108321-42-2 分子式 :C20H40N4O10 •2H2SO4 分子量 :692.71 比旋:+104o~+121o 水分:≤10% 吸光值:≤0.015 (280nm,1mg/ml), ≤0.1 (570nm, 10 ...
相关专题 1.G418的配制: 方案一: 300mgG418加入3ml PBS溶液,浓度为100mg/ml,完全溶解后,0.22um 过滤,-20℃保存。 方案二:(1)配制1M HEPES:23.8g HEPES(缓冲能力更强)粉末溶于100ml ddH2O,10N NaOH(加量较多)调节pH至7.3,过滤除菌(较难过滤),4℃保存,终 ...
相关专题 Q1:G418怎么配制? A1:我觉得不能用水配,因为这样PH会变化很大,至少要用PBS。我是配在HEPES溶液中的,具体方法如下:1g包装的G418瓶子中,加入10ml HEPES溶液,浓度为100 mg/ml完全溶解后,0.22 um过滤,-20度保存。HEPES缓冲液配方如下:90 ml 水中,0.8 g NaCl 0.03 ...
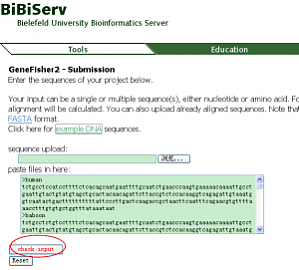
相关专题 一步步教你设计简并引物 GeneFisher提供一个在线的简并引物设计工具,基础是同源基因。目前的版本是2.0,加入了AJAX技术。主页:http://bibiserv.techfak.uni-bielefeld.de/genefisher2/ GeneFisher2 - Submission 点击 Genefisher2 Subm ...
相关专题 最近想用“简并引物”设计点实验做做,上网找了点东东,还是发现了不少东西。特总结如下:主要包括简并引物设计常用的几个方面及简并因为设计时的注意事项。 简并引物是指代表编码单个氨基酸所有不同碱基可能性的不同序列的混合物。密码子具有简并性,单以氨基酸顺序推测编码的DNA序列是不精确的,但可以设计成对简并引物,扩增所有编码已知顺序的核酸序 ...