相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 询价记录
- 文献和实验
- 技术资料
- 提供商:
表观生物
- 服务名称:
CUT&RUN测序
- 规格:
10G*2(IP+input)
CUT & RUN(cleavage under targets and release using nuclease),即核酸酶靶向切割和释放技术,是一项用于在细胞天然染色质环境下检测蛋白-DNA复合体的新方法,与传统的ChIP-Seq研究方法相比,该技术无需甲醛进行交联剂免疫沉淀等操作,因此具有快速、所需低细胞量,背景信号低、重复性好等优点,可用于单细胞水平研究。CUT & RUN对于表观遗传、基因调控等领域的研究具有革命性意义。
技术原理
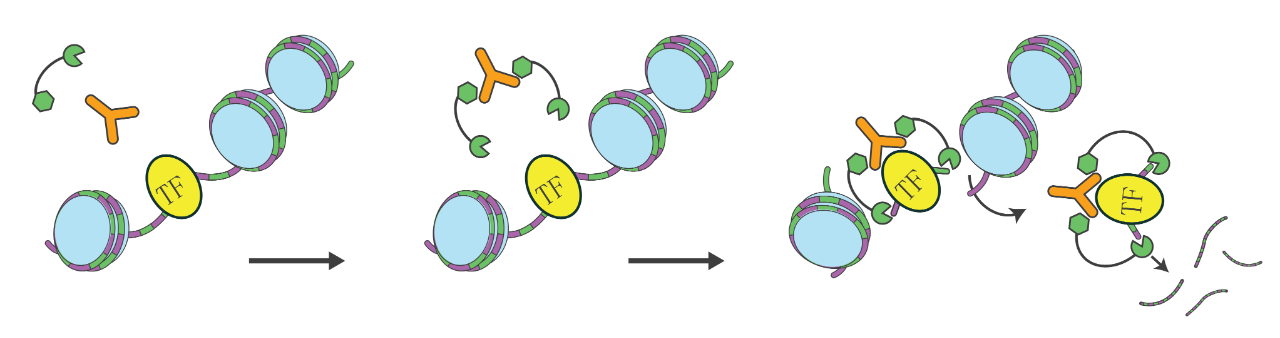
技术应用
技术特点
分析内容
送样要求
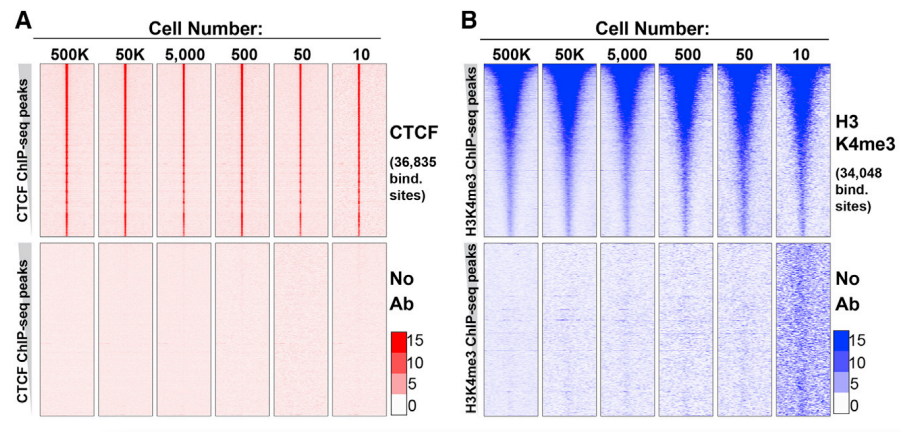
表观生物实测数据
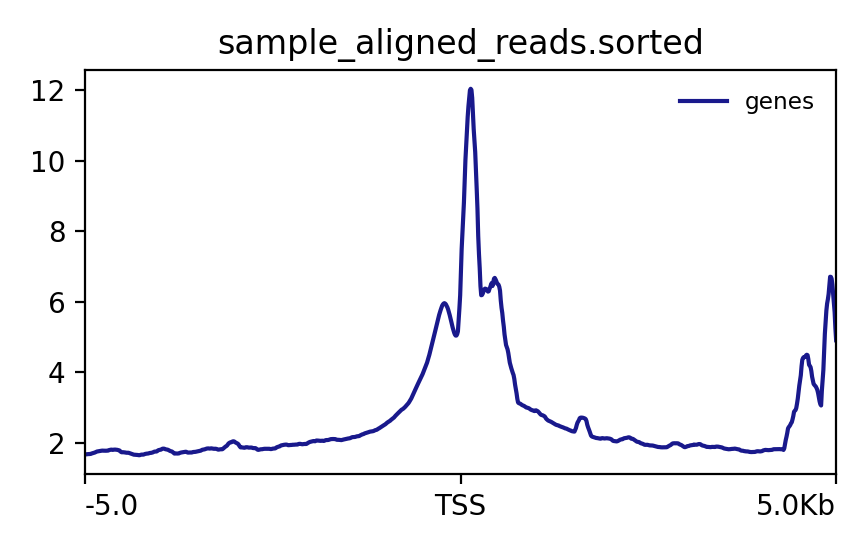
*注释:reads分布在TSS附近
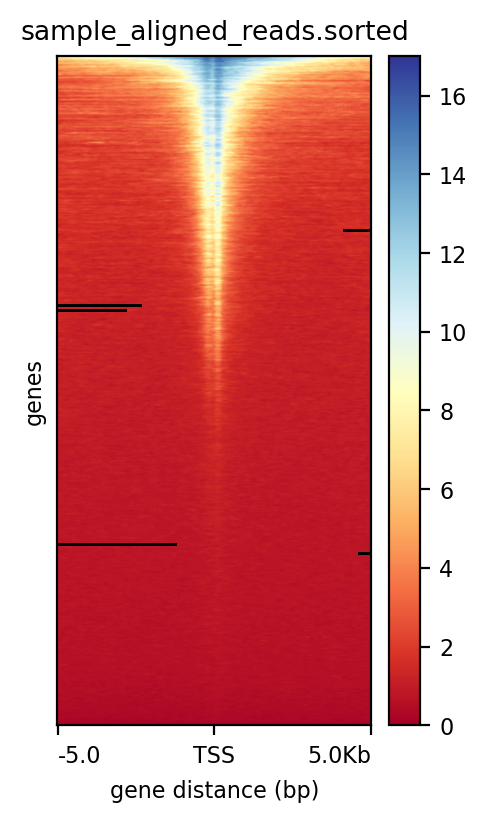
*注释:由热图可见,TSS附近reads富集程度
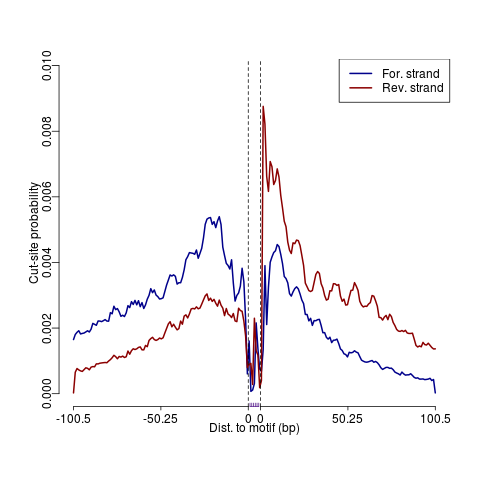
*注释:motif的footprinting可查看motif在各个位置上的切割频率,与峰调用相比,该方法可实现结合位置的更高分辨率映射。切割位点源自通过募集至抗体结合位点的pA-MN融合物染色质切割后产生的单个DNA片段的两端。由于染色质相关的蛋白结合,切割频率较低的区域倾向于受到保护,而没有结合的侧翼区域则显示较高的切割频率,特征性双峰,中心位置反映出motif位置范围
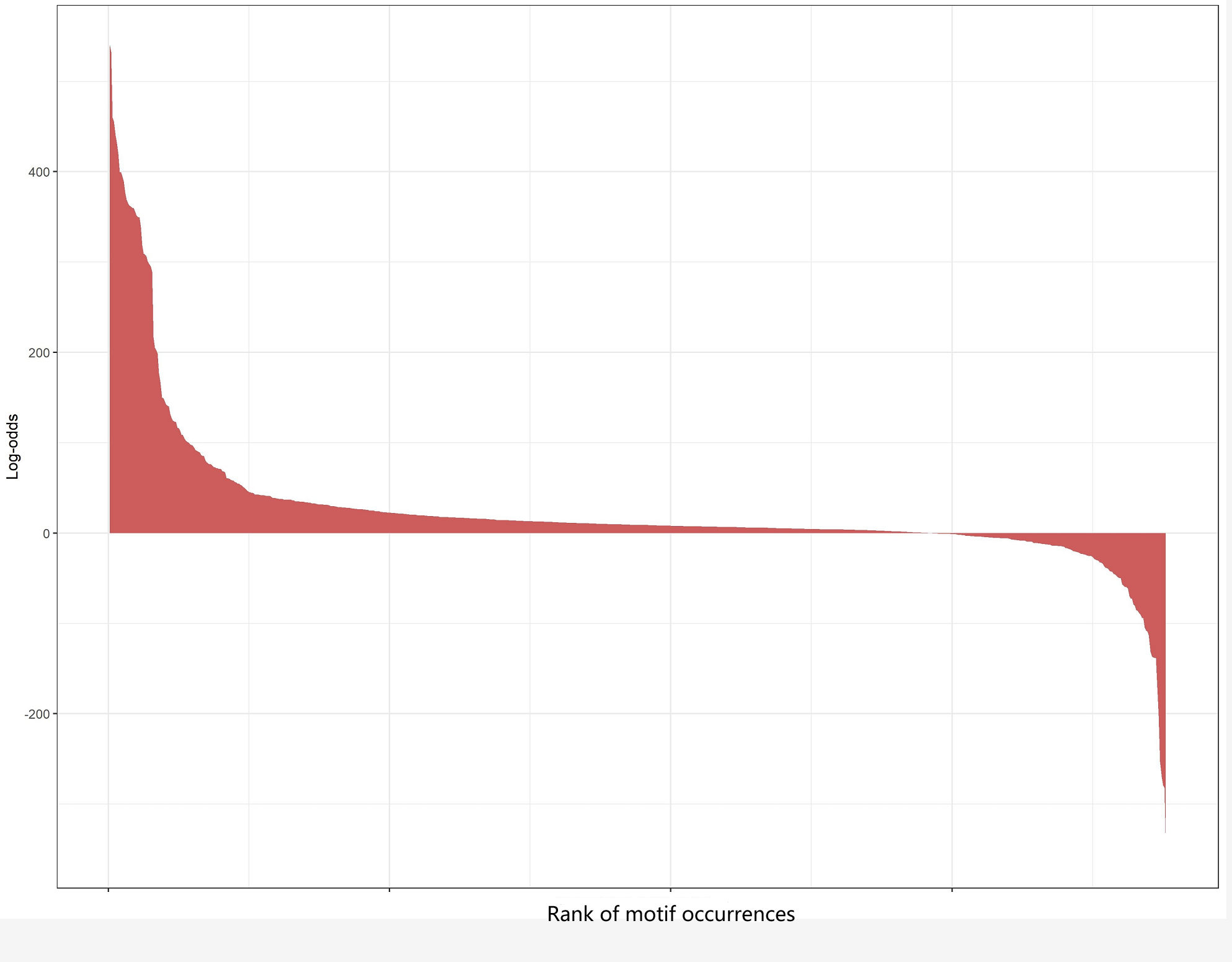
图4:log-odds图
*注释:由log-odds值对数量化了footprinting图中的切割位点,通过log-odds值得分对切割点进行排名,log-odds>5的得分位点可视为直接结合位点。
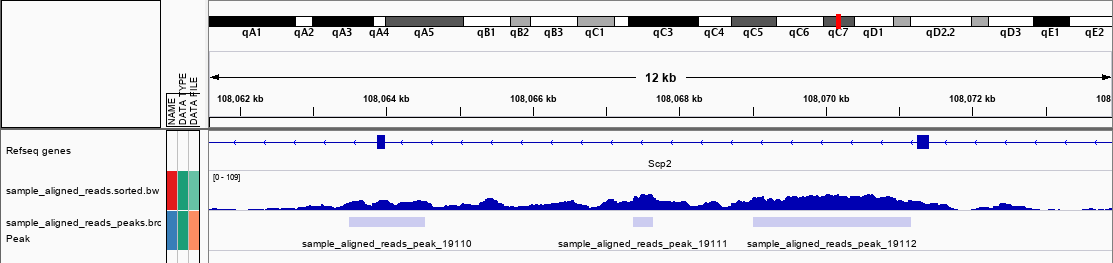
图5:IGV视图
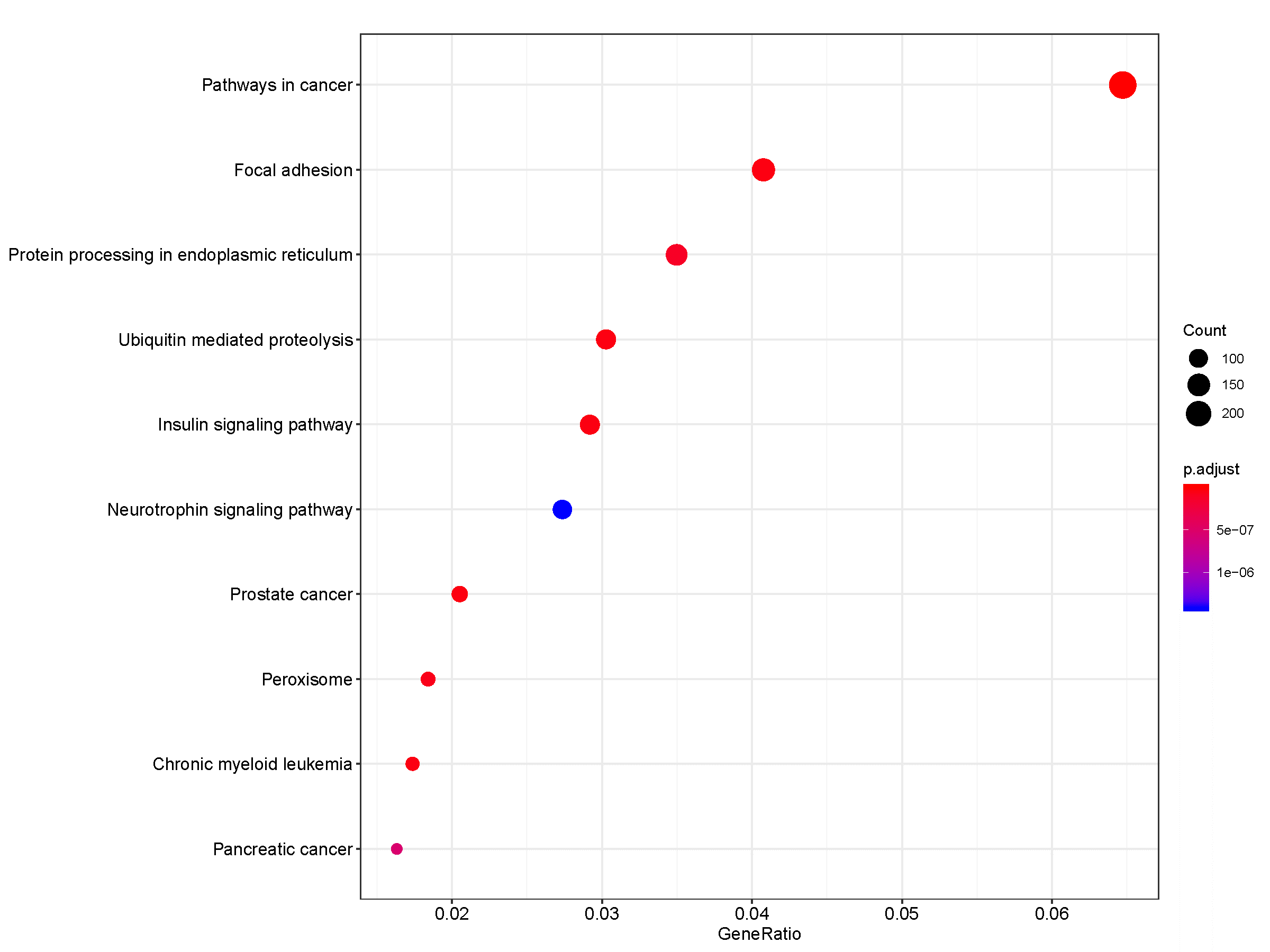
图6:KEGG富集
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
- 作者
- 内容
- 询问日期
 文献和实验
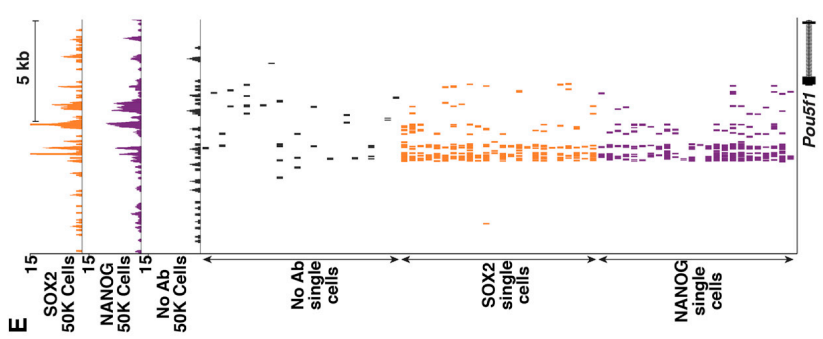
文献和实验[1] Sarah J, Ana B, Kurtis N, etal. Profiling of Pluripotency Factors in Single Cells and Early Embryos. Cell,2019, May 16; 177: 1-11.
。 2019 年 4 月 Thomas G Fazzio 教授等发表在 Cell 期刊的 uliCUT&RUN 技术,将蛋白质-DNA 互作研究升级到单细胞水平。近几个月来,又相继有文章报道了 CUT&Tag 等技术,进一步优化了蛋白质-DNA 互作研究方法,本文将对系列方法一并总结介绍。 1.蛋白质-DNA 互作新技术发展历程 1997 年 Orlando等研发出ChIP[1],主要用于研究转录因子与 DNA 结合位点的序列信息。 2009 年 Dominic Schmidt
endosperm using CUT&RUN (PMID:30719569) 的前端处理。 2. CUT&Tag 技术公布的 Protocol 适用于活细胞,对细胞状态有要求吗?那么经过 PFA 交联过的细胞可以使用吗? CUT&Tag 在活细胞上确实成功率比较高,在准备细胞时需要保证细胞处于高细胞活性状态,若细胞活性不佳,胞内蛋白质与核酸相互作用的状态会发生改变,对于死亡的细胞,目的蛋白有可能从染色质上脱落,进而影响实验数据。在准备细胞样本时使用台盼蓝进行细胞活性的检测
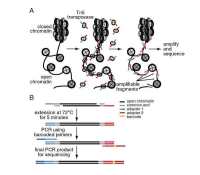
x PCR reaction buffer, 1.6 µl 4dNTP mix (250 pmol/µl), 2 µl T12 MN primer, 2 µl arbitrary primer (decamer), 0.2 µl Taq DNA polymerase (5 U/µl) use the PCR conditions as under section III. run PCR product on a 1.5 % agarose gel
 技术资料
技术资料