相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 询价记录
- 文献和实验
- 技术资料
- 保存条件:
-20度
- 保质期:
有效期1年
- 供应商:
翌圣生物科技(上海)股份有限公司
- 英文名:
Hieff Mut™ Multi Site-Directed Mutagenesis Kit
- 库存:
大量
- 规格:
10T
产品描述
Hieff Mut™ Multi Site-Directed Mutagenesis Kit 是基于Hieff Clone® 快速克隆技术的定点突变系统。使用本试剂盒,可一次性向目标质粒上三至五个不连续位点(相距超过50bp)同时引入定点突变。该试剂盒由两个模块组成:Hieff® II Pfu DNA Polymerase扩增模块和Hieff Clone® 快速克隆模块。Hieff® II Pfu DNA Polymerase超高的保真度显著降低了扩增过程中引入新突变的可能性,其卓越的长片段扩增能力,广泛适用于长度小于20 kb的任何质粒扩增。Hieff Clone®快速克隆系统利用高效的同源重组反应替代传统的退火成环反应。因此使用Hieff Mut™ Multi Site-Directed Mutagenesis Kit进行DNA定点突变时,引物设计更加灵活,且扩增反应以指数方式进行,极大减少了模板使用量,有利于原始甲基化模板的彻底降解。
Hieff Mut™ Multi Site-Directed Mutagenesis Kit中的重组酶Exnase MultiS经过优化,专门针对多碱基进行定点突变。此外,如扩增产物特异,其DpnI消化产物可不进行DNA纯化而直接用于重组反应。高度优化的反应缓冲液、快捷的操作流程以及极高的成功率,使得Hieff Mut™ Multi Site-Directed Mutagenesis Kit成为DNA多点突变试剂盒。
应用范围
DNA定点突变
注:如使用本品对质粒进行定点突变,请使用甲基化酶无缺陷的宿主菌 (例如Top10、DH5α、JM109)扩增原始质粒!
产品组成
|
编号 |
组分 |
产品编号/规格 |
|
11004ES10 (10 T) | ||
|
11004-A |
2×Hieff® II PCR buffer |
1.25 mL |
|
11004-B |
dNTP Mix (10 mM each) |
50 μL |
|
11004-C |
Hieff® II Pfu DNA Polymerase (1 U/μL) |
50 μL |
|
11004-D |
Dpn I (10 U/μL) |
50 μL |
|
11004-E |
2× Hieff Clone® MultiS Enzyme Premix |
100 μL |
运输与保存方法
冰袋(wet ice)运输。产品-20℃保存。有效期1年。
不连续多碱基(相距超过50bp)定点突变实验流程(以不连续三碱基为例)
实验流程概览(图一)
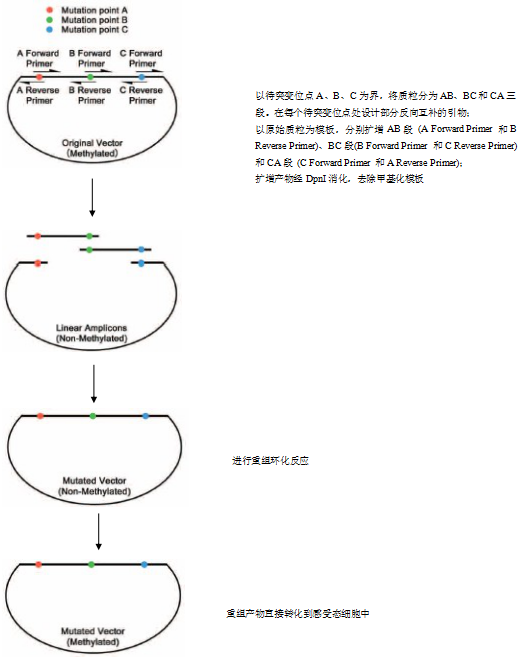
图一:使用Hieff Mut™ Multi Site-Directed Mutagenesis Kit进行不连续三碱基定点突变实验流程
1. 引物设计
向质粒三个不连续位点引入定点突变,只需设计三对引物将质粒分段扩增即可。引物设计基本原则为:在每个待突变位点处设计部分反向互补的扩增引物对。每对正反向引物5’端包含15-21 bp反向互补区域(GC含量40%-60%为佳),各引物待突变位点至引物3’端区域Tm值高于60℃为佳。所需引入突变可以包含在互补区域内 (需要两条引物上均引入点突变),也可以包含在任一条引物的非互补区域 (只需在一条引物上引入点突变),请勿将突变位点置于引物末端。以向pUC18引入三碱基突变为例,引物设计具体方案如图二所示。
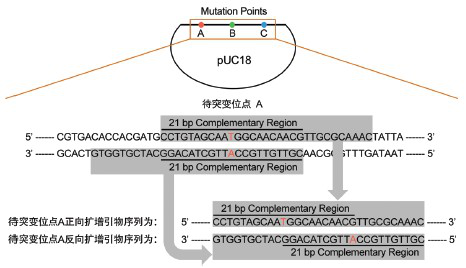
图二:向质粒引入不连续多碱基定点突变引物设计示意图
注:待突变位点B、C处的正反向扩增引物设计方式与位点A 处的引物设计方式一致。计算引物Tm值时,应计算待突变位点至引物3’端这一区域内的碱基,待突变位点至引物5’端区域内的碱基不应参与计算。
2. 目标质粒分段扩增
以待突变位点A、B、C为界,将质粒分为AB、BC段和CA段。使用Hieff® II Pfu DNA Polymerase对各段分别进行扩增。AB段扩增引物对为:待突变位点A正向扩增引物和待突变位点B反向扩增引物;BC段扩增引物对为:待突变位点B正向扩增引物和待突变位点C反向扩增引物;CA段扩增引物对为:待突变位点C正向扩增引物和待突变位点A反向扩增引物。
2.1 配制PCR反应体系
反应各组分解冻后请充分摇匀,使用完毕后及时放回-20℃。2×Hieff® II PCR buffer请勿长时间敞口放置。
为了增加扩增特异性,反应体系配制过程请于冰水浴中进行。为了防止Hieff® II Pfu DNA Polymerase的校对活性降解引物,请将聚合酶最后加入反应体系中。
推荐反应体系如下:
|
ddH2O |
Up to 50 μL |
|
2×Hieff® II PCR buffer |
25 μL |
|
dNTP Mix (10 mM each) a |
1 μL |
|
模板DNA b |
Optional |
|
引物1 (10 μM) |
2 μL |
|
引物2 (10 μM) |
2 μL |
|
Hieff® II Pfu DNA Polymerase (1 U/μL) c |
1 μL |
a. 请勿使用dUTP和带有尿嘧啶的引物或模板。
b 在各片段能正常扩增的前提下,应尽量减少质粒模板使用量,推荐≤1 ng。待扩增质粒长期放置、反复冻融会导致模板质粒断裂、开环或降解,因此推荐使用新鲜制备的质粒作为模板。
c. 推荐的酶的终浓度为1 U/50 μl反应。可将Hieff® II Pfu DNA Polymerase在0.5-2 U/50 μl之间进行优化,但不要超过2 U/50 μl,尤其当扩增子长度大于5 kb时。
2.2 PCR扩增反应
推荐PCR反应条件:
|
循环步骤 |
温度 |
时间 |
循环数 |
|
预变性 |
95℃ |
30 sec |
1 |
|
变性a 退火b 延伸c |
95℃ 60℃~72℃ 72℃ |
15 sec 15 sec 30-60 sec/kb |
30 d |
|
彻底延伸 |
72℃ |
5 min |
1 |
a. 对于大多数质粒,变性温度使用95℃即可。
b. Hieff® II Pfu DNA Polymerase能够促进模板和引物高效退火。一般来说,退火温度设置为引物Tm值即可。如果需要,可以建立一个温度梯度反应去寻找引物模板结合的最适温度。退火时间太长可能导致扩增产物在胶上呈现弥散状。
c. 对于大多数扩增反应,延伸过程可在72℃进行。长的延伸时间有助于提高扩增产量。
d. 为了防止扩增过程中引入非目标突变,强烈建议扩增循环数≤35。如果扩增效率良好的话,推荐扩增循环数应≤30。
反应结束后取少量扩增产物进行琼脂糖电泳检测。如目标质粒正确扩增,则可进行下步实验。
3. 扩增产物DpnI消化,去除甲基化模板质粒
因上述扩增产物中包含原始模板质粒,为防止其在转化后形成假阳性转化子,必须在进行重组环化之前进行DpnI消化。
推荐反应体系如下:
|
DpnI |
1 μL |
|
扩增产物 |
40~50 μL |
将上述反应体系置于37℃恒温反应1~2小时。如产物扩增特异,产物条带单一,DpnI消化产物无需纯化,可直接用于后续重组反应。如扩增不特异,DpnI消化结束后应胶回收纯化目标扩增产物。
4. 重组反应
4.1 配制重组反应体系
质粒AB、BC和CA段扩增产物末端分别包含相对应的完全一致的一段序列,因此在Exnase MultiS催化下三扩增产物末端可以发生同源重组,完成扩增产物环化过程。于冰水浴中,将下列组分依次加到无菌的1.5 ml Eppendorf管或PCR管的管底。如果不慎将液体粘在管壁,请务必通过短暂离心使其沉入管底。体系配制完成后,用移液器上下轻轻吹打几次混匀各组分,避免产生气泡 (请勿剧烈震荡或者涡旋混匀)。
|
ddH2O |
Up to 20 μL |
|
AB段DpnI消化产物 |
x ng |
|
BC段DpnI消化产物 |
x ng |
|
CA段DpnI消化产物 |
x ng |
|
2× Hieff Clone® MultiS Enzyme Premix |
10 μL |
Exnase MultiS多点突变重组反应体系最适DNA使用量为每片段0.03 pmol。该摩尔数对应的DNA质量可由以下公式粗略计算获得:
DpnI消化产物最适使用量 = [0.02 × 目标质粒碱基对数] ng (0.03 pmol)
例如,AB段长度为1 kb,BC段长度为2 kb,CA段长度为5 kb。则DpnI消化产物最适使用量为:AB段0.02 × 1000 = 20 ng;BC段0.02 × 2000 = 40 ng;CA段0.02 ×5000 = 100 ng。
注:DNA量太多或者太少都将降低环化效率。常用的吸光度测量法极易受DNA纯度、DNA稀释液pH等因素影响,测定值和DNA实际浓度往往偏差较大。因此,请务必通过琼脂糖电泳预先确认DNA浓度,尽量严格按照推荐量配制反应体系。当DpnI消化产物最适使用量计算值不足10 ng或者超过200 ng时,加入10ng或200 ng即可。DpnI消化产物不纯化直接用于重组反应时,使用量不应超过反应总体积的1/5,即4μl。
4.2 重组反应
1)将上述体系置于50℃反应 20-50 min。
2)待反应完成后,立即将反应管置于冰水浴中冷却5 min。
3)之后,反应产物可直接进行转化;也可储存于-20℃,待需要时解冻转化。
5. 反应产物转化、涂板、克隆鉴定
1)取10 μl冷却反应液,加入到100 μl感受态细胞中,轻弹管壁数下混匀,在冰上放置30 min。
2)42℃热激45~90秒,冰水浴孵育2 min。
3)加入900 μl SOC或LB培养基,37℃孵育10min充分复苏。
4)37℃摇菌45 min。
5)取100 μl菌液均匀涂布在含有适当抗生素的平板上。将平板倒置,于37℃过夜培养。
注:我们推荐您使用转化效率>108 cfu/μg的感受态细胞。如果感受态转化效率<108 cfu/μg (例如用CaCl2法新鲜制备的感受态转化效率通常在106-107 cfu/μg之间),请将培养菌液在5,000 rpm离心3 min收集菌体,用100 μl LB培养基重悬后全部涂板。
注意事项
1)引物设计时5’端反向互补区尽量选择无重复序列,且GC含量比较均匀的区域。当这一区域内GC含量在40%~60%范围之内时,重组环化效率将达到最大。如这部分区域GC含量高于70%或者低于30%,重组环化效率会受到较大影响。
2)当两突变位点相距小于50bp时,应将其视为一个突变位点,将两突变引入同一条/对引物进行实验。
3)DpnI只能识别甲基化DNA,请务必使用从甲基化酶无缺陷的宿主菌中扩增的质粒作为PCR模板。
4)质粒PCR扩增结果需高度特异,杂带较多会影响实验结果。
5)重组反应体系中应尽量避免金属络合剂(如EDTA)的带入。因此,建议将DNA纯化产物溶解在pH8.0的ddH2O中保存(常规胶回收试剂盒中的洗脱液可用pH8.0的ddH2O替代),请勿使用TE进行DNA保存。
6)冷却后的反应产物应在1h内进行转化,且转化前保持在冰水浴中。如需储存,于-20℃冻存。尽量避免室温较长时间放置或4℃长期储存。
7)反应产物的转化体积不应超过感受态细胞体积的1/10,否则会降低转化效率。另外,当转化的DNA浓度太高时,会抑制转化反应。将重组反应产物稀释5倍后取1/5进行转化。
8)为了您的安全和健康,请穿实验服并戴一次性手套操作。
9)本产品仅作科研用途!
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
- 作者
- 内容
- 询问日期
 文献和实验
文献和实验[1] Luo P, Xu Z, Li G, et al. HMGB1 represses the anti-cancer activity of sunitinib by governing TP53 autophagic degradation via its nucleus-to-cytoplasm transport. Autophagy. 2018;14(12):2155-2170. doi:10.1080/15548627.2018.1501134(IF:11.100)
[2] Wu R, Chen Y, Liu Y, et al. m6A methylation promotes white-to-beige fat transition by facilitating Hif1a translation. EMBO Rep. 2021;22(11):e52348. doi:10.15252/embr.202052348(IF:8.807)
[3] Ren X, Zhao H, Shi L, et al. Phosphorylation of Yun is required for stem cell proliferation and tumorigenesis. Cell Prolif. 2022;55(5):e13230. doi:10.1111/cpr.13230(IF:6.831)
[4] Wang M, Liu H, Zhang X, et al. Lack of MOF Decreases Susceptibility to Hypoxia and Promotes Multidrug Resistance in Hepatocellular Carcinoma via HIF-1α. Front Cell Dev Biol. 2021;9:718707. Published 2021 Sep 1. doi:10.3389/fcell.2021.718707(IF:6.684)
[5] Mo C, Li W, Jia K, Liu W, Yi M. Proper Balance of Small GTPase rab10 Is Critical for PGC Migration in Zebrafish. Int J Mol Sci. 2021;22(21):11962. Published 2021 Nov 4. doi:10.3390/ijms222111962(IF:5.924)
[6] Yao Y, Qu D, Jing X, et al. Molecular Mechanisms of Epileptic Encephalopathy Caused by KCNMA1 Loss-of-Function Mutations. Front Pharmacol. 2022;12:775328. Published 2022 Jan 13. doi:10.3389/fphar.2021.775328(IF:5.811)
[7] Wang Q, Ren D, Bi Y, et al. Association and functional study between ADIPOQ and metabolic syndrome in elderly Chinese Han population. Aging (Albany NY). 2020;12(24):25819-25827. doi:10.18632/aging.104203(IF:4.831)
[8] Zhang S, Zhang Z, Shen Y, et al. SCN9A Epileptic Encephalopathy Mutations Display a Gain-of-function Phenotype and Distinct Sensitivity to Oxcarbazepine. Neurosci Bull. 2020;36(1):11-24. doi:10.1007/s12264-019-00413-5(IF:4.246)
[9] Jin X, Yu W, Ye P. MiR-125b enhances doxorubicin-induced cardiotoxicity by suppressing the nucleus-cytoplasmic translocation of YAP via targeting STARD13. Environ Toxicol. 2022;37(4):730-740. doi:10.1002/tox.23438(IF:4.119)
[10] Jin Y, Liu W, Xiang Y, et al. Maternal miR-202-5p is required for zebrafish primordial germ cell migration by protecting small GTPase Cdc42. J Mol Cell Biol. 2020;12(7):530-542. doi:10.1093/jmcb/mjz103(IF:4.000)
[11] Deng L, Gong K, Wang G. Hsa_circ_0008344 Promotes Glioma Tumor Progression and Angiogenesis Presumably by Regulating miR-638/SZRD1 Pathway. Neurotox Res. 2022;40(3):825-836. doi:10.1007/s12640-022-00504-8(IF:3.911)
[12] Liu W, Xiang Y, Zhang W, Jia P, Yi M, Jia K. Expression pattern, antiviral role and regulation analysis of interferon-stimulated gene 15 in black seabream, Acanthopagrus schlegelii. Fish Shellfish Immunol. 2018;82:60-67. doi:10.1016/j.fsi.2018.07.041(IF:3.185)
[13] Yang M, Zhu L, Zhu L, Xu D, Yuan Y. Role of a Rare Variant in APC Gene Promoter 1B Region in Classic Familial Adenomatous Polyposis. Digestion. 2021;102(4):527-533. doi:10.1159/000509234(IF:2.692)
[14] Shen L, Ji C, Lin J, Yang H. Regulation of circADAMTS6-miR-324-5p-PIK3R3 ceRNA pathway may be a novel mechanism of IL-1β-induced osteoarthritic chondrocytes. J Bone Miner Metab. 2022;40(3):389-401. doi:10.1007/s00774-021-01308-0(IF:2.626)
Site-Directed Mutagenesis of Antibody-Variable Regions
oligonucleotide-directed mutants. Several polymerase chain reaction-based mutagenesis techniques have been described (3 –5 ). However, available thermostable polymerases are error-prone, leading to undesired sequence changes. For example, Taq polymerase
合成的突变质粒不被切开从而除去原来的模版质粒。虽然这个试剂盒比上一个麻烦,但实际上是引入了两个定点突变。只不过一个用于酶切除掉模版的反应。 有的时候研究可能需要多个位点的定点突变,比如改造酶的活性或者动力学特性,研究蛋白之间的相互作用位点等,单点突变不能满足实验的需要,重复进行单点突变也非常浪费时间。因而Stratagene公司又推出了QuikChange Multi Site-Directed Mutagenesis kit。最多一次实验可以引入5个定点突变。这个试剂盒的原理
作用位点等,单点突变不能满足实验的需要,重复进行单点突变也非常浪费时间。因而Stratagene公司又推出了QuikChange Multi Site-Directed Mutagenesis kit。最多一次实验可以引入5个定点突变。这个试剂盒的原理和Clontech的相似,就是准备多个带突变的引物(同方向,对同一单链模版),退火后全部突变引物(不超过5个)都结合在同一环状单链模版,PfuTurbo聚合酶延伸,碰到下一个引物就停止,各片断经连接成环,和单链模版组成杂和环,DpnI消化双链模版,也消化杂和环