万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
真核有参转录组测序是基于已知参考基因组或转录组的物种,通过高通量测序技术全面分析基因表达水平、可变剪切、融合基因等分子特征的技术。
一、实验流程
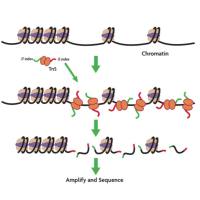
1. 样本准备与RNA提取
- 样本类型:组织、细胞、血液等(需避免降解)。
- RNA提取:使用试剂盒(如TRIzol)提取总RNA,检测纯度(OD260/280 ≥1.8)、浓度及完整性(RIN值 ≥7,Agilent Bioanalyzer)。
2. 文库构建与测序
- mRNA富集:用oligo(dT)磁珠捕获真核生物polyA尾mRNA(适用于完整RNA)。
- 链特异性建库:片段化后合成cDNA,添加测序接头(如Illumina TruSeq)。
- 测序平台:Illumina NovaSeq/HiSeq(主流),PE150双端测序,推荐数据量6-12 Gb/样本。
3. 质控环节
- RNA质检(Qubit、Bioanalyzer)、文库质检(Qubit、qPCR)、测序数据质控(FastQC评估)。
二、分析内容
1. 基础分析
- 数据质控:FastQC检查原始数据,Trimmomatic过滤低质量reads。
- 序列比对:Hisat2/STAR将reads比对到参考基因组。
- 基因定量:StringTie/featureCounts计算基因/转录本表达量(FPKM/TPM)。
2. 核心分析
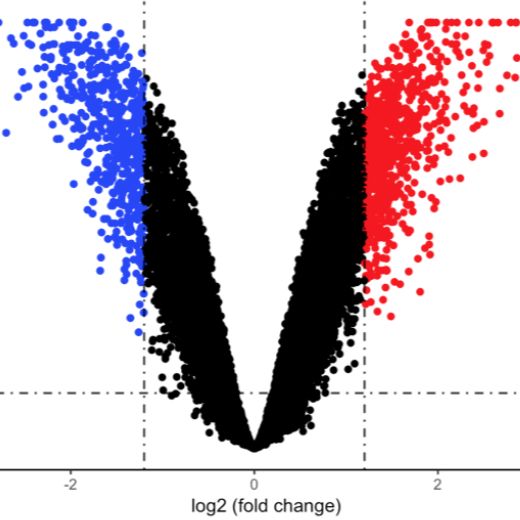
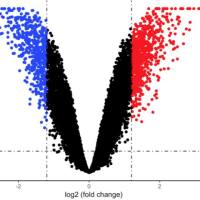
- 差异表达分析:DESeq2/edgeR筛选差异基因(阈值:|log2FC|>1,p<0.05)。
- 功能注释与富集:GO、KEGG、Reactome等数据库注释,ClusterProfiler进行富集分析。
- 可变剪切分析:rMATS/MISO检测差异可变剪切事件。
3. 高级分析
- 融合基因检测:STAR-Fusion/Arriba分析基因融合事件。
- SNP/INDEL分析:GATK检测RNA水平的突变。
- 共表达网络:WGCNA挖掘基因模块与表型的关联。
4. 数据交付
- 原始数据(fastq)、表达矩阵、差异基因列表、可视化图表(火山图、热图、通路图等)。
三、应用方向
1. 基因表达调控
- 比较不同条件(疾病vs正常、处理vs对照)的差异表达基因,揭示调控机制。
2. 疾病机制研究
- 癌症、神经退行性疾病等关键基因筛选(如肿瘤标志物发现)。
3. 发育与分化研究
- 追踪胚胎发育、干细胞分化的动态表达变化。
4. 药物研发
- 药物靶点筛选、毒理评估(如药物处理后的基因表达扰动)。
5. 分子标记开发
- 基于差异基因开发诊断或预后生物标志物(如血液肿瘤分型)。
6. 环境响应
- 研究生物对胁迫(高温、重金属)的转录响应。
7. 进化与比较基因组学
- 跨物种比较基因表达差异,解析进化机制。
四、技术优势
- 高灵敏度:可检测低丰度转录本。
- 多维度分析:整合表达量、突变、可变剪切等多层信息。
- 个性化定制:根据需求扩展分析(如LncRNA、circRNA研究)。
适用物种:人类、小鼠、拟南芥等基因组注释完善的物种。
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验[1] Stark R, Grzelak M, Hadfield J. RNA sequencing: the teenage years. Nat Rev Genet. 2019 Nov;20(11):631-656. doi: 10.1038/s41576-019-0150-2. Epub 2019 Jul 24. PMID: 31341269.
[2] Polioudakis D, de la Torre-Ubieta L, Langerman J, Elkins AG, Shi X, Stein JL, Vuong CK, Nichterwitz S, Gevorgian M, Opland CK, Lu D, Connell W, Ruzzo EK, Lowe JK, Hadzic T, Hinz FI, Sabri S, Lowry WE, Gerstein MB, Plath K, Geschwind DH. A Single-Cell Transcriptomic Atlas of Human Neocortical Development during Mid-gestation. Neuron. 2019 Sep 4;103(5):785-801.e8. doi: 10.1016/j.neuron.2019.06.011. Epub 2019 Jul 11. PMID: 31303374; PMCID: PMC6831089.
[3] Huala E, Dickerman AW, Garcia-Hernandez M, Weems D, Reiser L, LaFond F, Hanley D, Kiphart D, Zhuang M, Huang W, Mueller LA, Bhattacharyya D, Bhaya D, Sobral BW, Beavis W, Meinke DW, Town CD, Somerville C, Rhee SY. The Arabidopsis Information Resource (TAIR): a comprehensive database and web-based information retrieval, analysis, and visualization system for a model plant. Nucleic Acids Res. 2001 Jan 1;29(1):102-5. doi: 10.1093/nar/29.1.102. PMID: 11125061; PMCID: PMC29827.
[4] Wang C, Hao L, Sun X, Yang Y, Yin Q, Li M. Response mechanism of psychrotolerant Bacillus cereus D2 towards Ni (II) toxicity and involvement of amino acids in Ni (II) toxicity reduction. J Hazard Mater. 2022 May 15;430:128363. doi: 10.1016/j.jhazmat.2022.128363. Epub 2022 Jan 29. PMID: 35183050.
[5] Zhu Y, Jin L, Shi R, Li J, Wang Y, Zhang L, Liang CZ, Narayana VK, De Souza DP, Thorne RF, Zhang LR, Zhang XD, Wu M. The long noncoding RNA glycoLINC assembles a lower glycolytic metabolon to promote glycolysis. Mol Cell. 2022 Feb 3;82(3):542-554.e6. doi: 10.1016/j.molcel.2021.11.017. Epub 2022 Jan 25. PMID: 35081364.
 技术资料
技术资料暂无技术资料 索取技术资料