万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 技术资料
- 库存:
9999
- 规格:
50 rxns
产品货号:F1501S
储存条件:-20℃
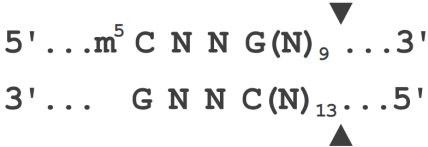
SgeI 可识别与切割含有 5-mC 位点的 DNA 靶标序列,一条链或双链甲基化均可被识别。
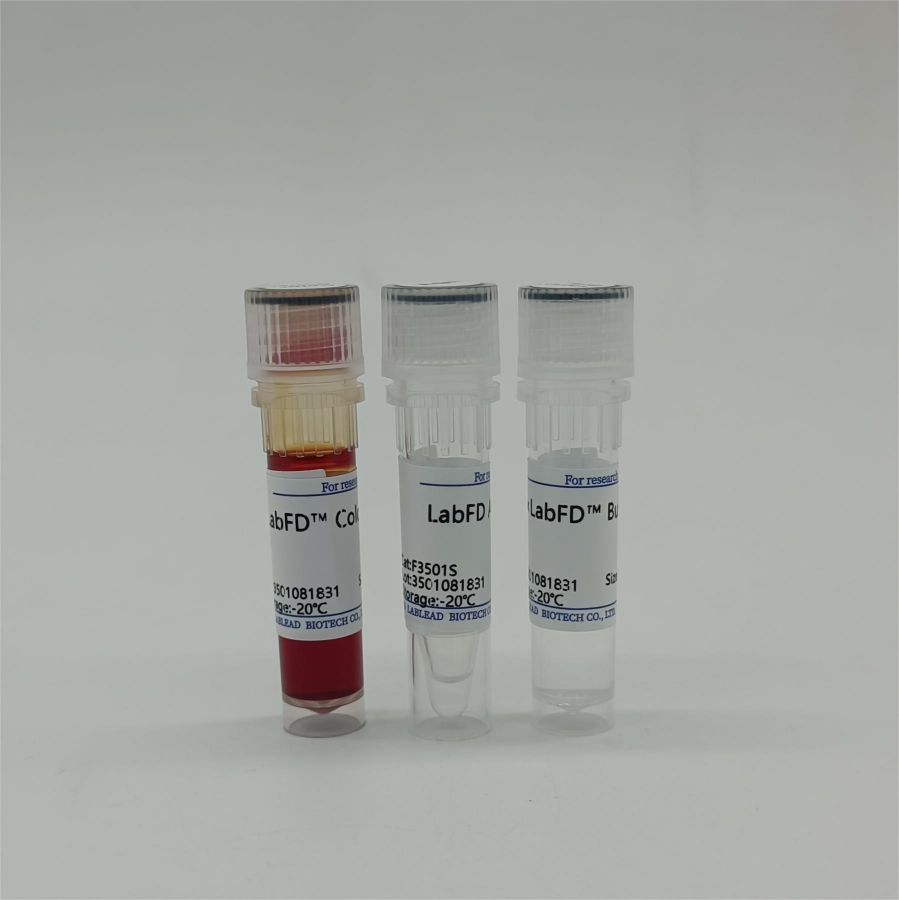
产品组成
|
组分 |
规格 |
|
LabFD™ Sgel |
50ul |
|
10×LabFD™ Buffer |
1ml |
|
10×LabFD™ Color Buffer |
1ml |
产品简介
LabFD™快速内切酶是一系列经过基因工程重组、能够在 5~15 分钟内精确完成 DNA 切割的高保真限制性内切酶,适用于质粒DNA、PCR 产物或基因组DNA 等的快速酶切。LabFD™快速内切酶具有如下特点:5~15 分钟内即可完成酶切;共用一种酶切Buffer,大大简化酶切反应体系;良好的酶活冗余度, 轻松应对底物过量或困难模板酶切。此外,LABLEAD去磷酸化、连接试剂在 LabFD™酶切 Buffer 中具有100%活性,支持一管化反应,提升“酶切-修饰-连接”的体验。
酶活定义
在 1×SgeI Buffer 条件下,1μg pUC19-SgeI DNA (Dcm+) 在50μl 反应体系中37℃孵育1 h,不断增加酶量,直至酶切产物DNA带型不随着酶量的增加而发生变化,此时酶量定义为1 U。
质量控制
核酸内切酶残留检测
将 3U Sgel 与超螺旋质粒 DNA 在 37℃温育 4 h,通过DNA电泳检测质粒无变化。
核酸外切酶残留检测
将 5U Sgel 与双链 DNA 底物在 37℃温育 1 h,通过 DNA 电泳检测双链DNA 底物无变化。
注意事项
1. 底物至少需要 2 个 SgeI 识别序列才能有效酶切。
2. 甲基化 DNA 完全酶切取决于 SgeI 的识别位点的数量,另外由于识别位点酶切产生的DNA产物会促进SgeI
的非特异性酶切,因此建议酶切时优化SgeI酶量用于酶切反应。
使用方法
1. DNA 酶切流程
① 在冰上按如下建议的加样顺序配制反应体系:
|
质粒 DNA |
PCR 产物 |
基因组 DNA | |
|
ddH2O |
15ul |
16ul |
30ul |
|
10×LabFD™ Buffer 或 10×LabFD™ Color Buffer |
2ul |
3ula |
5ul |
|
底物 DNA |
2ul(up to 1ug) |
10ul(~0.2ug) |
10ul(5ug) |
|
LabFD™ SgeI |
1ul |
1ul |
5ul |
|
Total |
20ul |
30ul |
50ul |
注:反应体系可以按比例放大或缩小。反应时间不建议超过 1 h。
② 轻柔吸打或轻弹管壁以混匀(切勿涡旋),然后瞬时离心以收集挂壁液滴;
③ 37℃温育1 h;
④ 80℃温育 20 min即可使酶失活,停止反应(可选)。
2.双酶切或多酶切
每种快速内切酶的用量为 1ul,并根据需要适当扩大反应体系;
所有快速内切酶的体积总和不得超过总反应体系的 1/10;
如果所用的几种快速内切酶的最适反应温度不同,应先以最适温度低的酶开始酶切,再添加最适温度较高的酶,在其最适反应温度下进行酶切反应。
3.适用于质粒的扩大反应体系
注:如果总反应体系大于 20ul,应适当增加温育时间,尽量使用水浴、金属浴或沙浴。
|
DNA |
1ug |
2ug |
3ug |
4ug |
5ug |
|
LabFDTM Sgel |
1ul |
2ul |
3ul |
4ul |
5ul |
|
10×LabFD™ Buffer 或 10×LabFD™ Color Buffer |
2ul |
2ul |
3ul |
4ul |
5ul |
|
Total |
20ul |
20ul |
30ul |
40ul |
50ul |
甲基化修饰影响
|
Dam |
Dcm |
CpG |
EcoKI |
EcoBI |
|
无影响 |
总是切割被 Dcm甲基转移酶甲基化的DNA |
切割与 CpG 甲基化序列重叠的靶点 |
无影响 |
无影响 |
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 技术资料
技术资料暂无技术资料 索取技术资料