上海烈冰生物医药科技有限公司品牌商
11 年
手机商铺
- NaN
- 0
- 0
- 0
- 0
推荐产品
技术资料/正文
【IF=41.444】RNA-seq助力海军军医大学/上海九院团队构建更安全的基因编辑系统!
256 人阅读发布时间:2022-12-07 15:46
生命体是个错综复杂的网络,任何一类RNA分子都不是独立存在的,越来越多的研究团队发现长RNA可以通过“miRNA 海绵”机制结合和吸附部分互补的miRNA,从而间接调控miRNA靶基因的表达。
CRISPR-Cas系统是原核生物一种抵御外来入侵核酸的免疫系统,已被广泛用于遗传育种、新药开发、疾病治疗、动物模型构建、转录调控以及分子诊断等各领域的基因组编辑中,被誉为在后基因组时代开创了遗传疾病精准治疗的新时代。
烈冰生物合作伙伴——海军军医大学王越教授团队、上海交通大学医学院附属第九人民医院秦兴军/王业飞合作团队与美国南加州大学应其龙团队合作在《Molecular Cancer》杂志(IF=41.444)上发表Cas9 RNA新机制解析和新应用开发的研究成果。

基于基因编辑技术和RNA-seq,团队发现了外源性长链核酸物质Cas9 RNA可通过miRNA海绵机制影响宿主细胞基因表达,并开发了RNA序列优化策略,以提高该基因编辑系统的安全性。烈冰生物提供了研究中的RNA测序和部分生物信息数据分析支持。
- Cas9 RNA通过 miRNA 海绵机制影响宿主细胞基因表达

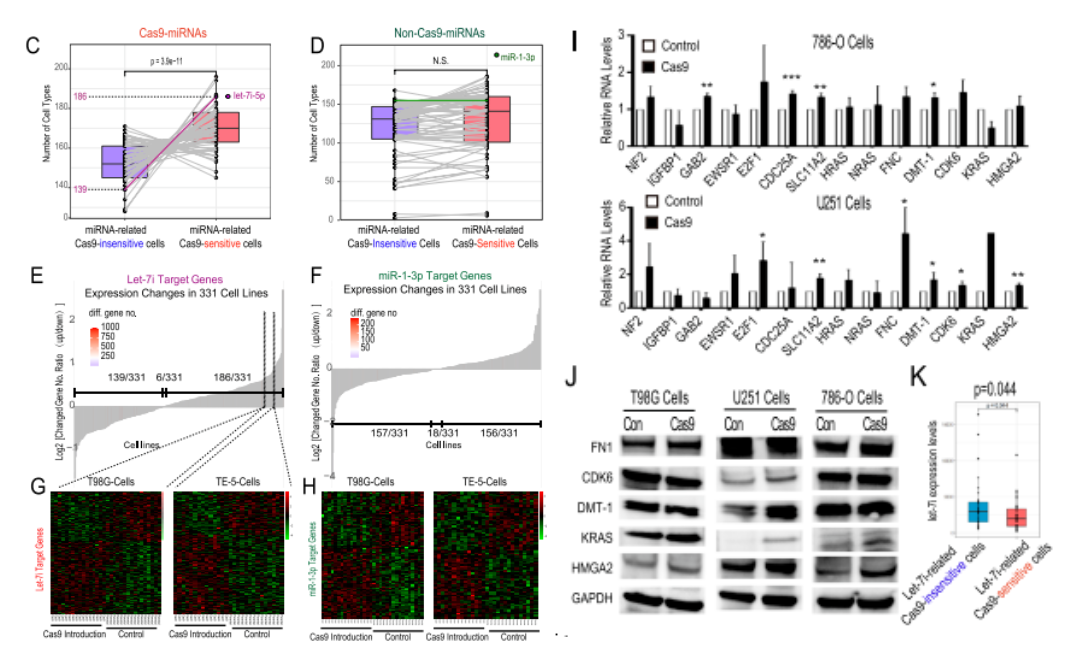
在这331个细胞系中,研究者发现大多数“Cas9-miRNAs”的靶基因在很多细胞中存在Cas9 RNA导入后上调宿主细胞中相应miRNA的靶基因表达的趋势;而“non-Cas9-miRNAs”的靶基因就不存在这种趋势,这符合Cas9 RNA通过 miRNA 海绵机制发挥作用的假设。其中Let-7家族是Cas9 RNA的主要调节 miRNA(186种细胞在引入Cas9后,let-7i的相关靶基因普遍上调表达),此外qPCR和Western Blot实验也证实,在更多类型的细胞(U251、786-O、T98G 与 MCF7)在引入 Cas9 后,一些let-7i的靶基因显著上调。因此,Cas9 RNA可能充当了 microRNA 海绵,进而影响 Cas9-miRNA靶基因。
- 那么为什么 Cas9 的 miRNA 海绵效应在不同的细胞系中差异很大呢?
基于miRNA基础表达数据库(CCLE)和331个细胞系数据的联合分析后,团队发现let-7i相关Cas9敏感细胞的let-7i靶基因表达水平较低,而let-7靶基因的高频突变都分布在“Cas9不敏感”细胞中,即靶基因的表达量和突变等可能影响了Cas9 RNA发挥miRNA海绵效应。
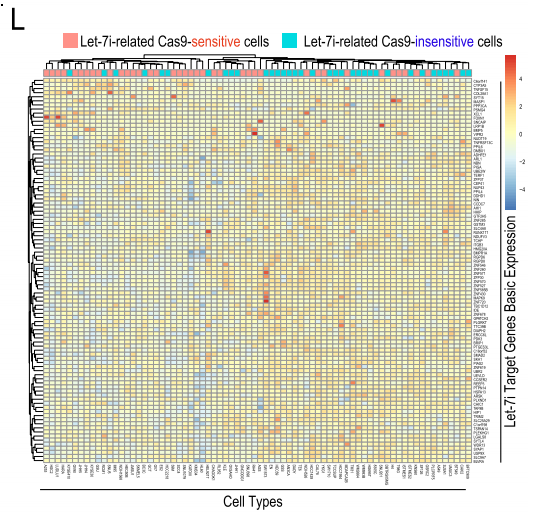
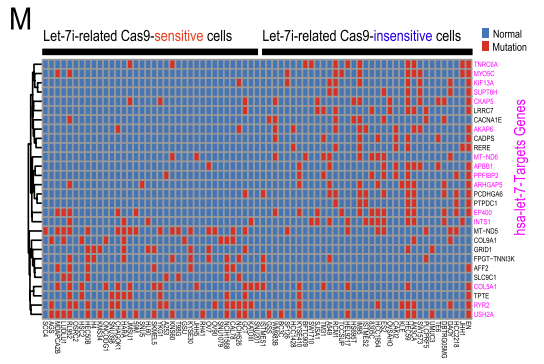
- 优化 Cas9 RNA 序列可降低其对细胞增殖和let-7靶基因的影响
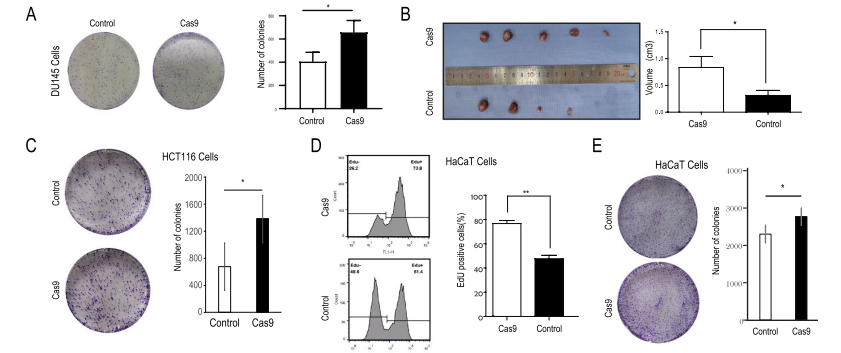
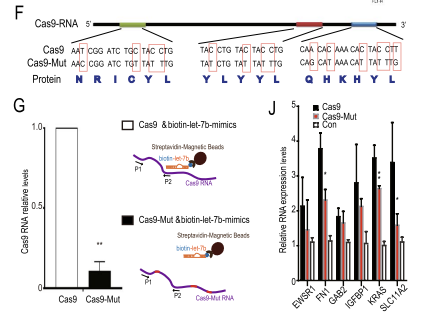
为了进一步验证miRNA海绵机制及在RNA水平上优化Cas9序列,研究人员构建了一个同义突变质粒(Cas9-Mut),发现Cas9-mut的mRNA可以被翻译成与Cas9相同的氨基酸序列。HEK293细胞的RNA-pulldown实验显示,相较于Cas9组,Cas9-mut与let-7的结合显著减少,转染 Cas9-Mut的DU145细胞的增殖能力和let-7靶基因上调能力也弱于转染Cas9的细胞,这给未来可以通过同义突变的方法进一步优化Cas9 RNA提供了可能。
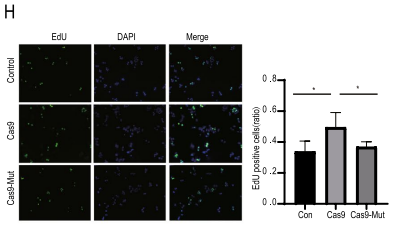
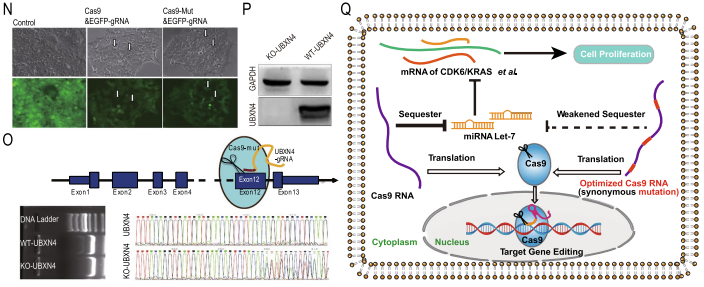
为了确定优化后的Cas9-Mut是否仍然具有基因编辑功能,团队将Cas9-Mut和设计的 EGFP-gRNA引入稳定表达EGFP的HEK293细胞后,出现了一些EGFP阴性细胞;Cas9-Mu质粒和靶向UBXN4基因的gRNA转入DU145细胞也证实Cas9-Mut仍然具有基因编辑功能,这为其在实际应用中提供了支持。
海军军医大学王越、上海交通大学医学院附属第九人民医院秦兴军/王业飞,美国南加州大学的应其龙为论文的共同通讯作者,海军军医大学的蒋俊锋,曾韬,张莉和范杏飞为文章的并列第一作者。
原文链接:https://doi.org/10.1186/s12943-022-01604-x
转载请注明“烈冰生物”
烈冰生物依托NovelBrain®生物信息数据分析系统,应用细胞聚类分析、subCluster分析、差异基因表达分析、GO/Pathway分析、细胞通讯分析、QuSAGE分析等多种分析工具,帮助研究团队在生信分析层面抽丝剥茧,团队进一步结合免疫荧光等多种技术手段,建立了翔实可信的子宫内膜细胞亚群及微环境互作图谱。








