清华大学刘翔宇团队连发两篇 PNAS
2025-11-11 18:00点击次数:92
关键词:近日,清华大学药学院/北京生物结构前沿研究中心刘翔宇实验室在《PNAS》杂志先后发表了两项研究成果,提出了一种名为「Click Fusion」的研究策略。他们利用近年来兴起的机器学习驱动的蛋白质设计工具,深入探索了蛋白质从头设计在 GPCR 融合蛋白设计中的应用潜力,为解析非激活态 GPCR 结构以及筛选 GPCR 新配体提供了具有广阔应用前景的新工具。


一、G 蛋白偶联受体(GPCRs)是什么?
G 蛋白偶联受体(GPCRs)作为一类具有七次跨膜结构的膜蛋白,是生理调控的核心元件。它们识别多样的胞外信号,比如激素、神经递质、光等,并将信号向胞内传递,介导广泛的细胞应答。正因其在疾病发生与发展中的重要作用,GPCRs 被视作药物研发的关键靶点。这一重要性在临床用药中得到印证:据统计,约 34% 的 FDA 批准药物以 GPCR 为靶点;在这些药物中,拮抗剂占比过半,激动剂占比超过四成。
二、为什么非激活态 GPCR 结构解析很困难?
结构生物学是揭示分子药理机制与指导药物设计的关键手段。然而,在 GPCR 研究中,解析其非激活态的冷冻电镜结构仍是一大技术瓶颈。这与结合 G 蛋白的激活态 GPCR 结构研究中形成鲜明对比:在冷冻电镜下,这种 90-150 kDa 的 GPCR-G 蛋白复合物因其具有 G 蛋白这一可溶性「信号源」,为颗粒对齐提供了稳定特征,因而更易于解析。相反,拮抗剂结合的 GPCR(尤其分子量仅 37-50 kDa 的 A 类受体)结构解析则困难得多,它们因缺乏额外的可溶结构域,如同一个难以捕捉的微小目标,给结构解析带来了巨大挑战。
应对这一挑战的关键在于引入不被去垢剂胶束包裹的结构域作为颗粒对齐的特征,例如引入纳米抗体或融合蛋白。自 2007 年 Brian Kobilka 团队利用融合 T4L 蛋白获得第一个人源 GPCR 结构以来,融合蛋白已成为 GPCR 结构研究的重要工具。随后,Aashish Manglik 团队与程亦凡团队验证了 BRIL/PGS 等融合蛋白在冷冻电镜结构解析中的可行性。他们同时指出,在目标受体与融合蛋白之间形成刚性连接,从而使受体与融合蛋白的相对位置保持一致,是帮助结构解析的关键。进一步地,陶余勇团队、胡红丽团队以及徐俊等人开发了多种融合蛋白策略,在非激活态 GPCR 的冷冻电镜结构解析中验证了效果。然而,这些方法依赖于对蛋白融合位点的生化筛选,工作量较大。
三、GPCR 配体筛选的难点是什么?
GPCR 配体筛选面临的主要挑战之一源于该受体家族的高度动态性。由于 GPCR 在非活性和活性状态之间存在构象动态平衡,其结构灵活性使得直接筛选并富集针对某一特定构象的配体变得尤为复杂。目前一些基于结合实验的筛选技术,如 DNA 编码化合物库、酵母表面展示库和噬菌体展示库,已经在探索 GPCR 配体的化学空间及实现构象选择方面展现出潜力。但是这些方法成功的关键在于获得构象稳定的高纯度受体。而获得固定构象,尤其是激活态构象的受体并不容易。目前常用的制备激活态受体的策略包括引入完全激动剂、G 蛋白或其模拟物来进行构象稳定。这些方法虽有一定效果,却存在明显局限:完全激动剂会占据正构配体结合口袋,从而阻碍其他正构激动剂的筛选;同时,现有策略制备的受体-G 蛋白复合物往往稳定性不足,在实验过程中易发生解离,影响筛选的准确性与效率。因此开发具有稳定构象(尤其是激活态构象)的工程化受体,以破解相关研究瓶颈,已成为 GPCR 药物筛选领域的迫切需求。
四、如何设计融合蛋白?
研究团队将 AI 驱动的蛋白质设计技术应用于融合蛋白设计,研发出一种名为「Click Fusion」(点击融合)的新策略。该命名具有双重含义:一方面,蛋白质工程设计过程高度依赖计算机设计,只需轻点鼠标即可完成,无需进行大量实验筛选;另一方面,融合蛋白能够像匹配的卡扣一样「咔嗒」一声精准结合,实现无缝转移。基于这一策略,研究团队成功设计出分别将 GPCR 稳定在激活态与非激活态的融合蛋白。其设计思路源于对 GPCR 激活过程中关键构象变化的把握,即调控第六跨膜螺旋 TM6 的摆动。通过将 TM6 稳定在「内收」或「外摆」的位置,可相应地将受体稳定于非激活态或激活态(图 1 A)。
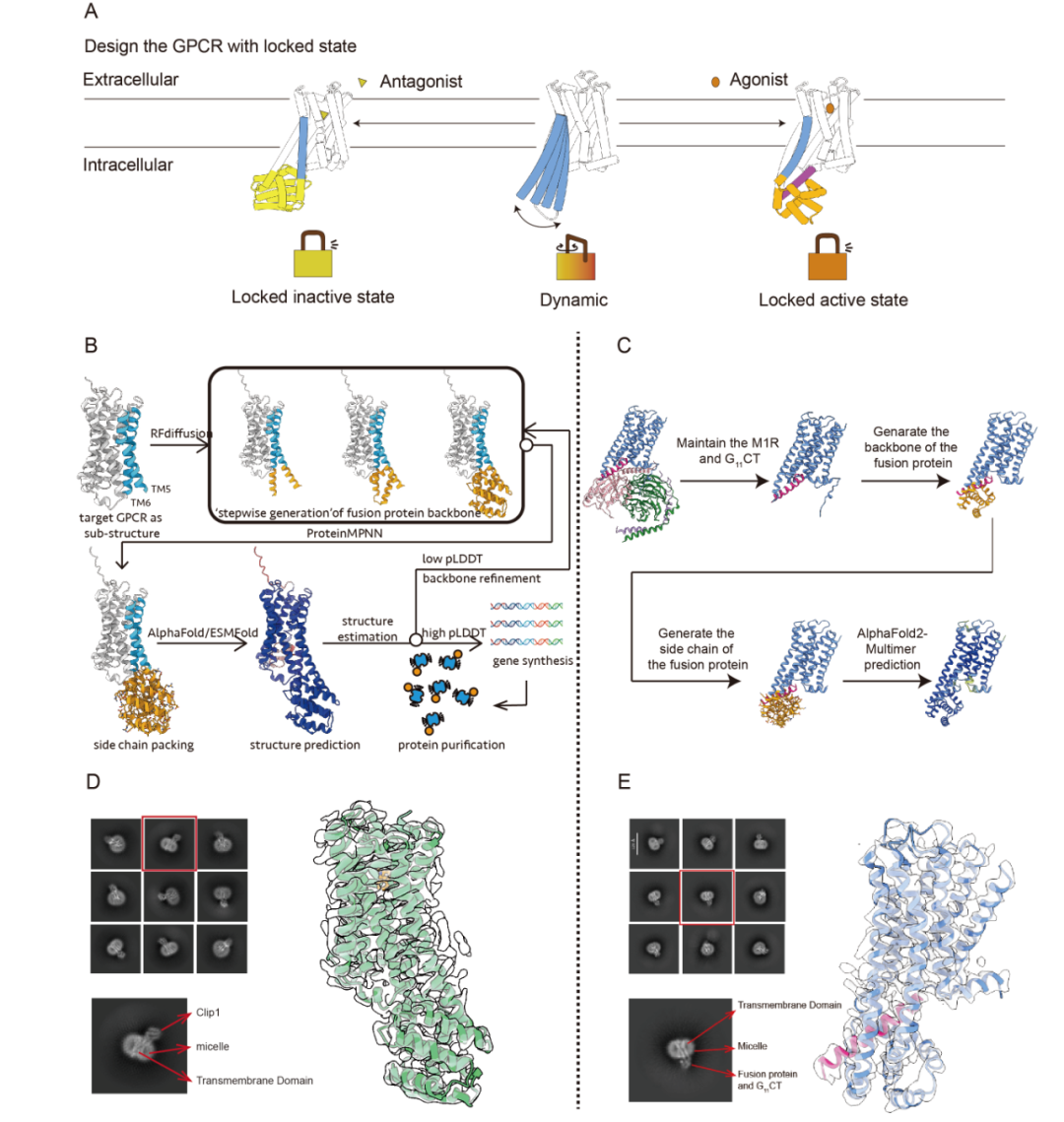
图 1:融合蛋白设计(A)设计融合蛋白稳定 GPCR 的非激活态和激活态构象(B)GPCR 稳定在非激活态的 M1R-Clip 设计流程(C)GPCR 稳定在激活态的 M1R-G11CT 设计流程(D)M1R-Clip1 的冷冻电镜结构(E)M1R-G11CT 的冷冻电镜结构
1、将 GPCR 稳定在非激活态的融合蛋白设计
设计流程如图所示,分为以下几个关键步骤:1. 融合位点选择:基于 GPCR 结构模型(来自 AlphaFold2 预测或 GPCRdb 数据库),删除细胞内环 3(ICL3),并确定 TM5 和 TM6 的连接残基作为融合位点;2. 骨架生成:使用 RFdiffusion 方法分三步生成融合蛋白骨架,包括延伸 TM5 和 TM6 的螺旋段、在延伸区域插入稳定的中间螺旋作为结构支撑,以及生成完整的 Clip 蛋白骨架(长度约 100–150 个氨基酸);3. 序列优化与结构预测:使用 ProteinMPNN 进行侧链生成和序列优化,采用 AlphaFold2 或 ESMFold 对融合构建体进行结构预测,并以 pLDDT 分数评估结构可信度和连接区域的刚性,对低评分区域再次利用 RFdiffusion 优化骨架结构;4. 实验验证准备:选择高 pLDDT 分数的设计方案,合成编码 DNA,在细胞中表达、纯化以获得 GPCR-Clip 蛋白,进行后续生化与结构验证(图 1 B)。
2、将 GPCR 稳定在激活态的融合蛋白设计
设计原理基于激活态 GPCR 的结构基础。G 蛋白的 α5 螺旋被保留下来用于锁定 TM6 结构域向外移动的构象。前期研究表明,仅 α5 螺旋即可稳定 β2-肾上腺素受体的活性状态。基于这些研究基础,稳定激活态受体的融合蛋白设计流程与非激活态融合蛋白的设计流程基本一致,但进行了独特的改进。其流程分为以下几个关键步骤:1. 输入结构准备:基于 GPCR-G 蛋白复合物结构,保留完整的受体和 G 蛋白的 α5 螺旋;2. 融合蛋白生成:使用 RFdiffusion 生成融合蛋白的骨架,使其环绕包围整个 α5 螺旋,并使用 ProteinMPNN 进行侧链生成和序列优化,同时使用 AlphaFold2-multimer 进行结构预测;3. 构造设计:在 TM5 和 TM6 之间插入融合蛋白的同时,将 α5 螺旋通过 2×GGSGG 的连接序列融合到受体的 C 末端(图 1 C)。
五、M1R-Clip1 和 M1R-G11CT 的生化验证
为验证 Click Fusion 策略的可行性,研究团队首先选择了 M1 型毒蕈碱乙酰胆碱受体(M1R)作为模式受体。研究团队分别设计了稳定在非激活态的 M1R-Clip1 蛋白和稳定在激活态的 M1R-G11CT 蛋白。M1R-Clip1 和 M1R-G11CT 都能在昆虫细胞中表达,并且其表达量相较于野生型的 M1R 有明显提高。在配体竞争结合实验中,M1R-Clip1 对拮抗剂 atropine 和激动剂 iperoxo 的结合能力与野生型 M1R 基本一致,表明其保留了原有药理特性。M1R-G11CT 对激动剂 iperoxo 的结合能力相对于野生型的 M1R 提升了近三个数量级,且形成一个拟合良好的单相曲线,表明了 M1R-G11CT 被稳定在均一的激活态。
随后,研究团队成功解析了 M1R-Clip1 与 M1R-G11CT 的冷冻电镜结构。在二维分类图像中,可清晰观察到融合蛋白的特征信号,这表明该信号在颗粒对齐过程中起到了关键作用。最终获得的三维结构不仅与 AlphaFold2 的预测模型高度一致,其整体构象和配体结合位点也与已报道的结构无明显差异。更重要的是,结构分析明确显示,M1R-Clip1 处于非激活状态,而 M1R-G11CT 则被稳定在激活状态(图 1 D、E)。
六、融合蛋白辅助非激活态 GPCR 结构解析
非激活态 GPCR 结构解析长期受困于两大痛点:缺乏有效的对齐标记与受体构象不稳定。Clip 的设计既为冷冻电镜提供了刚性对齐特征,又将受体牢牢锚定在非激活态。M1R-Clip1 结构的成功解析便是此策略可行的首个证明。
为验证本方法的普适性。研究团队选择了神经调节素 B 受体(NMBR)—— 一个缺乏非激活态结构的受体作为试金石。最终,凭借 NMBR-Clip2 构建体,团队成功解析了 NMBR 的三维结构(分辨率 3.4 Å),并揭示了 NMBR 的活性调节机制。结构清晰地显示,当内源性激动剂 NMB 深入结合口袋时,其末端的甲硫氨酸会与「开关残基」产生相互作用,触发了开关的下移,从而触发了受体的激活。而拮抗剂不会触碰到该「开关残基」,不会引起受体的激活。
七、融合蛋白辅助 GPCR 的胞外纳米抗体筛选
基于 Click Fusion 策略,研究团队开发了一种筛选 GPCR 胞外纳米抗体的新方法。该方法利用序列生成模型,构建出一组骨架相同但序列各异的融合蛋白。这些融合蛋白帮助生成一组被称为「胞内正交受体」的工具受体蛋白,其胞外区域完全一致,而胞内性质各不相同。利用它们进行交叉筛选,可避免抗体在胞内区域的非特异性富集,从而实现对胞外纳米抗体的精准筛选。以 M1R 为模型,研究者成功筛选到特异性识别激活态与非激活态受体的胞外纳米抗体,其功能实验进一步证实了它们具有别构调节活性。该胞内正交筛选法为 GPCR 功能性纳米抗体的开发提供了通用解决方案,在药物研发中具有应用潜力(图 2)。
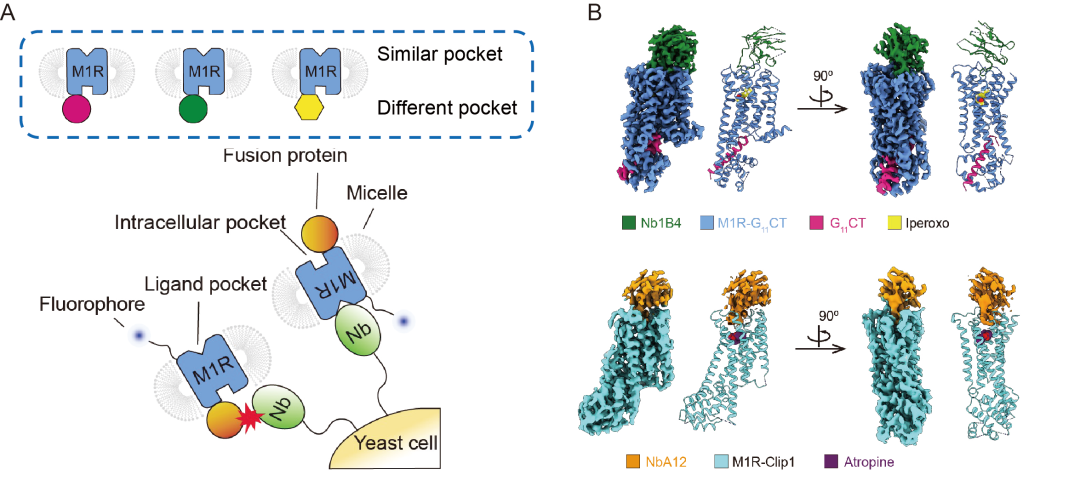
图 2:GPCR 胞外纳米抗体筛选(A)胞内正交筛选策略(B)本工作筛选得到的构象选择性纳米抗体与 M1R 受体的冷冻电镜结构
八、即插即用、快速迁移
融合蛋白的快速迁移性是 Click Fusion 策略得以广泛应用的根本。得益于 GPCR 家族高度的结构相似性,研究团队选取 5-HT2BR 为模型,通过结构比对确定融合位点,成功将 M1R-Clip1 上的融合蛋白迁移至 5-HT2BR,构建了 5-HT2BR-Clip1 蛋白。他们进一步解析了拮抗剂 Balovaptan 与 5-HT2BR 的复合物结构,揭示了其相互作用的分子机制,从而证实了 Clip 在辅助 GPCR 非激活态结构解析中的通用性。此外,团队还成功构建了 α1A-GqCT、μOR-GiCT 等一系列稳定于激活态的受体。放射性同位素结合实验表明,这些受体对各自激动剂的亲和力均显著提升。这些工作为筛选具有特定功能的 GPCR 配体提供了极具吸引力的技术平台(图 3)。
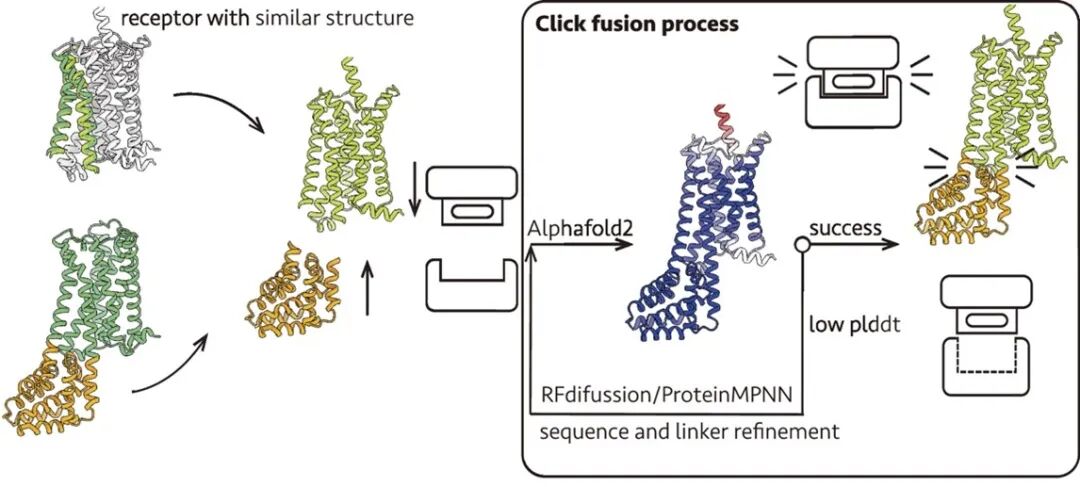
图 3:融合蛋白的快速迁移
清华大学药学院/北京生物结构前沿研究中心刘翔宇副教授为这两篇文章的(共同)通讯作者,中南大学湘雅医院妇产科张卫社教授为第一篇文章的共同通讯作者。清华大学药学院已毕业博士生高凯旋,2021 级博士生张欣,中南大学湘雅医院已毕业博士生聂佳为共同第一作者。清华大学药学院田博学副教授、医学院博士后赵佳伟(已出站)为课题提供了重要指导。本研究受到国家自然科学基金、北京生物结构前沿研究中心、清华北大生命科学联合中心、清华大学笃实计划等支持。国家蛋白质科学中心(北京)清华大学分中心冷冻电镜平台,清华大学生物医学分析中心同位素室、流式细胞仪机组为本研究提供了技术支持。
原文链接
https://www.pnas.org/doi/abs/10.1073/pnas.2422360122
https://www.pnas.org/doi/10.1073/pnas.2508879122
来源:北京生物结构前沿研究中心
如需代发文章宣传、新闻稿、招聘等,请后台回复【学术】添加小编
我们长期为科研用户提供前沿资讯、实验方法、选品推荐等服务,并且组建了 70 多个不同领域的专业交流群,覆盖神经科学、肿瘤免疫、基因编辑、外泌体、类器官等领域,定期分享实验干货、文献解读等活动。
添加实验菌企微,回复【】中的序号,即可领取对应的资料包哦~
【2401】论文写作干货资料(100 页)
【2402】国内重点实验室分子生物学实验方法汇总(60 页)
【2403】2024 最新最全影响因子(20000+ 期刊目录)
【2404】免疫学信号通路手册
【2405】PCR 实验 protocol 汇总
【2406】免疫荧光实验 protocol 合集
【2407】细胞培养手册
【2408】蛋白纯化实验手册
【2501】染色体分析方法汇总
【2502】国自然中标标书模板
【2503】WB 实验详解及常见问题解答
【2504】DeepSeek 论文写作常用口令
【2505】中国科学院期刊分区表(2025 年最新版)
【2506】期刊影响因子(2025 年最新版)
【2507】130 种实验室常用试剂配制方法(附全套资料)
【2508】常见信号通路
【2509】限制性核酸内切酶大全