PyRosetta-FoldTree
ZeroDesigner
如果你打开一个 PDB 文件,你会看到(x,y,z)来表示蛋白的 3D 坐标,当你移动蛋白时,你会修改单个原子的三个坐标,即为每个原子都有(x,y,z)三个自由度,对于大量原子计算来说,3 个自由度的计算量太过庞大。为了使计算简单,rosetta 使用分子内坐标进行替代,即使用一个原子的相邻原子之间的角度,键以及扭转,来进行定义。
内坐标此时同样也具有三个自由度:键,角度,扭转( bond, angle, and torsions)。但这三个自由度并不是同等重要的。对大多数蛋白模拟来说,在键和角度之间这里有非常小的改变,我们可以将这两个固定来进行考虑。对于蛋白来说,发生的移动大多数发生在扭转角的变化,因此,我们将每个原子的三个自由度缩减到一个自由度。除此之外,对于某些原子 (例如氢原子),他们的扭转角自由度是被周围的化学环境固定的。对这些原子来说,自由度可以从 3 到 0.
如果你在一系列的残基中改变了某个扭转角,会发生什么?
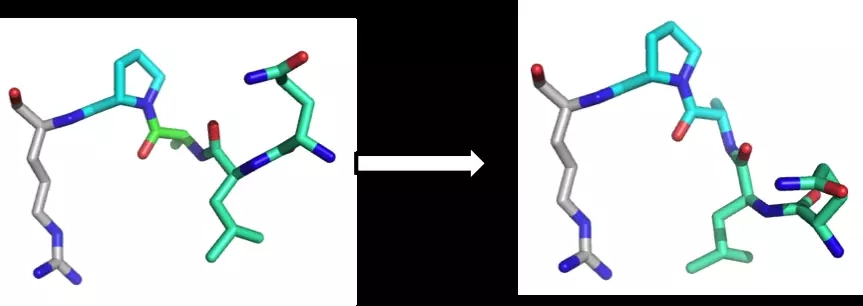
如上图所示,你改变了青色氨基酸的一个扭转角,在其右侧的所有的原子将会移动。换句话说,尽管原子相互之间是被固定的,但右侧原子仍然一起移动以适应青色氨基酸扭转角的额改变。
下面展示另外一个例子:
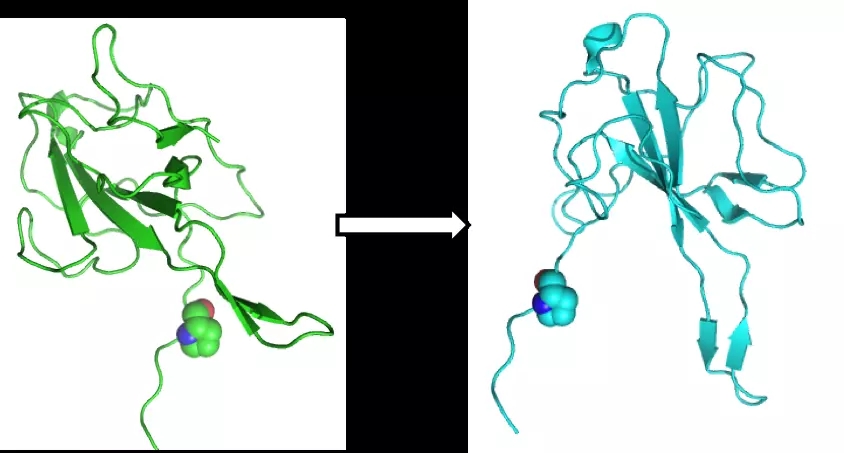
你可以看到在蛋白质的 N 端部位的扭转角的小小的改变,将会导致蛋白质其余部位的巨大变化,这就是所谓的杠杆效应(the lever arm effect)。
我们如何去避免这种变化
使用 FoldTree
在这里我们将会看到如何使用 FoldTree 去控制蛋白的部分移动,但原子之间的相互关系仍然不会变化。FoldTree 告诉 rosetta 在给定的结构中的残基之间的相互的关联性。他确定了在上游的残基(或称为父类),以及在下游的残基(或称为子类)。一些例子将会使这个概念清晰。
在本例中,您将使用冠状病毒的核壳部分的 N 端的结构。
查看此蛋白的 FoldTree
FOLD_TREE EDGE 1 133 - 1
这个读法应该是边缘是从 1 - 133,这是一个聚合边缘
你从 A 链的 1 位残基开始一直到最后的残基 133 位。即任何的上游残基(N 段残基)的移动将会导致下游残基(C 端残基)的移动。这条链是一条未被破环的连续残基(中间没有 miss),通过共价键以线性方式相连。Rosetta
就将这样的聚合成为 EDGE - 1,这个意味着在这个 EDGE 中的链接均是共价键的形式。
如果你使你的蛋白的 1 - 20 位的残基弛豫,而不改变剩余的结构的骨架,看看什么发生。
将改变后的文件与原始的文件相互对比。您可以看到,与原始结构相比,N 端部位已经大幅移动。 如果我们只对齐 N 端部分,您可以看到蛋白质的其余部分已经大幅度移动以适应小的 20 残基 N 端的变化。
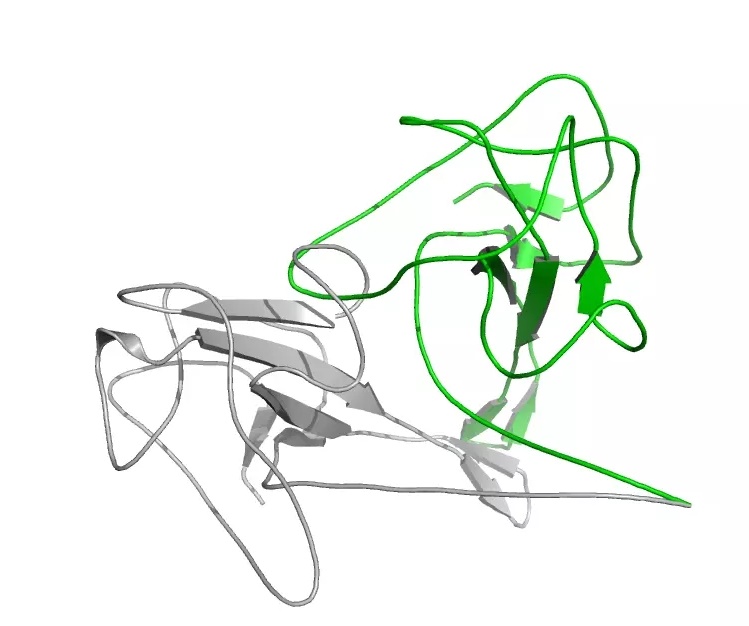
> * 原始的结构为灰色而蓝色结构为改变后的结构。
如果我们将上下游的位置互变,会发生什么。
下面是输出结构与原始蛋白。你可以看到蛋白的移动的幅度变小了。这是因为 C 端是发生改变的上游部位,所以下游的 N 端部位的移动并不会造成上游部位改变。
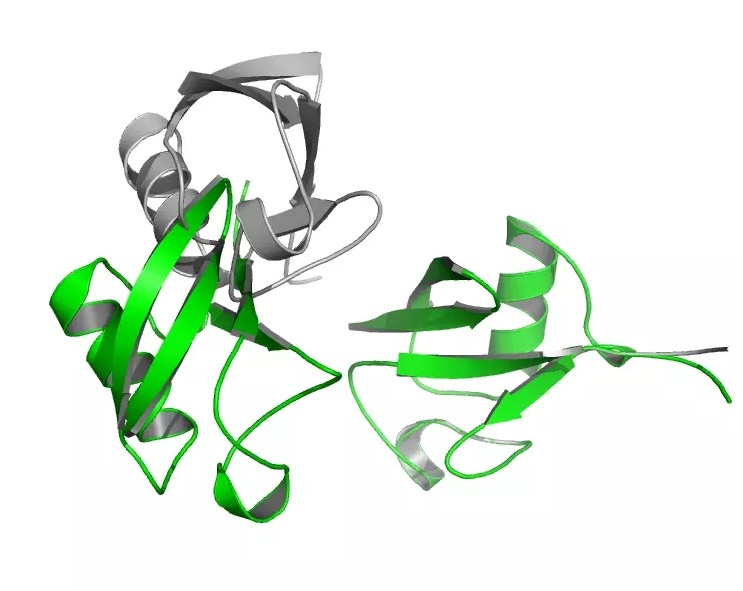
原始结构是灰色,青色的部位为改变的结构
所以,如果你有一个很灵活的 N 末端,改变 foldtree 将会是一个不错的选择。
具有多个链和跳跃的复合物
在这个例子中,我们将会运行一个虚拟的泛素二聚体。
首先看下,在弛豫之后会发生什么。使用 rosetta 的默认的 foldtree。
FOLD_TREE EDGE 1 76 - 1 EDGE 76 77 1 EDGE 77 152 - 1
蛋白边缘:1 - 76,77 - 152
Jump 边缘:76 - 77
我们有三个 EDGE,第一个,氨基酸残基从 1 - 76,共价线性链接(chainA)。第三个,氨基酸残基从 77 - 152,共价线性链接(chainB)。
注意:
第二个 EDGE,从 A 链的 76 到 B 链的 77,这并不是一个共价链接。这并不是一个共价连接,但是,因为 Rosetta 使用内坐标(扭转角),并不是一个 3D 坐标,在这个结构中的所有部位都必须相互链接。如果并没有共价连接,那么我们创造一个赝连接,这个赝连接就叫做 Jump。一个 Jump 的边缘是他的自己。在 Foldtree 中,jumps 以正整数形式显示,第一个 Jump 得到数字 1,每个 Jump 的数字都会增加。
首先,我们来对默认的 foldtree 进行弛豫。
在输出文件中,我们可以看到第二条链与他的原始位置相比较移动了很多。这是另外一个杠杆效应的例子。
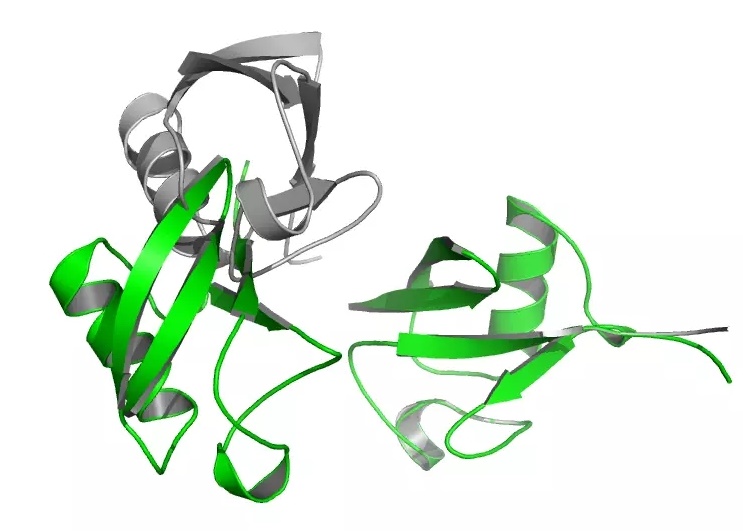
原始结构是灰色,改变的结构的为绿色。
现在我们进行一些操作。目前已知在实验中,Val71(chainA)与 Val146(ChainB)在表面的形成中是十分重要的,并且我们想要他们在弛豫中也保持紧密的连接。所以,我们可以重新确定 foldtree,使 Val71(A)以及 Val146(B)成为直系父类(最上游)。
FOLD_TREE EDGE 71 1 - 1 EDGE 71 76 - 1 EDGE 71 146 1 EDGE 146 77 - 1 EDGE 146 152 - 1
读法:
protein egde (- 1), protein edge (- 1), jump edge (1), protein edge (- 1), protein edge (- 1)
目前你可以看到这里有许多的 EDGEs,A 链由两个 EDGE 组成,第一个为 71 位的残基直到 A 链的 N 末端,第二个 EDGE 为 71 位的残基直到 A 链的 C 末端。JUMP 从 71 到 146,两个父类。B 链同样也由两个 edges 组成。