微生物组学领域新突破!师徒齐发 Science,通过不同的系统性手段揭示细菌代谢产物及其相应功能
人体中存在的细菌总量高达一千亿左右,它们能够感知周围的环境,并分泌一系列小分子做出反应。其中一些分子同时能够对其它微生物个体或宿主本身产生影响。
近年来,大量研究揭示了人体菌群稳态与身体健康或疾病发生之间的相关性,但其中的分子机制或直接产生作用的因子仍不清楚。
近期, Science 上刊登的两篇研究性文章中,研究者们分别通过不同的系统性手段揭示了细菌代谢产物以及相应的功能。
其中,来自斯坦福大学生物工程系的 Michael A. Fischbach 教授团队通过基因缺失突变的手段筛选出了对人体免疫系统存在影响的细菌代谢产物 1;

图片来源:Science
来自普林斯顿大学分子生物学系的 Mohamed S. Donia 教授团队描述了如何通过宏基因组学(metagenomics)以及生物信息学分析手段寻找此前未知的细菌代谢产物,并筛选其中具有抗生素活性的亚群 2。

图片来源:Science
据说 Donia 教授在担任普林大学助理教授之前,曾在 Fischbach 教授实验室从事博士后研究工作。
因此,本期的两篇「背靠背」文章也是师徒传承的典型范例,值得我们好好学习一下!
研究背景
DNA 测序技术的普遍化使得人类微生物组相关的数据呈爆发式增长。通过对微生物组以及代谢组学的数据进行分析,可以得到与特定微生物代谢产物相关的基因,从而便于进一步的研究。
然而,相对于人类基因组来说,微生物组学涉及更多的基因以及不同的细菌个体的相互作用,因此要想搞清楚任何一个分子的产生过程以及宿主作用靶点是及其复杂且困难的事情。
要想真正确定某个基因的功能,需要通过遗传学手段将其「敲除」并进行功能验证。
对于微生物组学研究来说,由于目前缺乏广泛的基因编辑工具,因此限制了向下进行机制研究的可能。此外,微生物组生物学机制研究的另一个重要障碍是宿主 - 菌群相互作用的复杂性。
人类遗传学的差异,个体微生物群的空间和时间动态,人与人之间以及不同环境下个体之间微生物群的变异性以及微生物群内的功能冗余都使得难以清晰地了解单个分子可能发挥的作用。
针对这两个问题,Fischbach 教授与 Donia 教授分别给出了解决方案。
主要内容
1. 「芽孢梭菌」基因编辑系统的建立与功能验证
CRISPR-Cas9 大家可能已耳熟能详。目前 CRISPR-Cas9 基因编辑工具已被用于各类遗传学研究以及动物模型的建立;进一步开发后更展现出了治疗各类遗传性疾病的潜力。
对于微生物组学的机制研究来说,CRISPR-Cas9 也是理想的基因编辑工具之一。然而,由于绝大多数肠道微生物属于厌氧性细菌,因此难以进行体外的培养,更别提进行任何遗传操作。
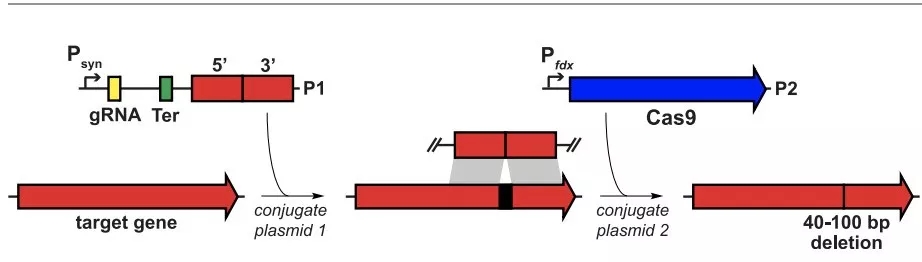
对此,Fischbach 教授等人开发了一类适用于「芽孢梭菌(C. sporogenes)」的 CRISPR-Cas9 基因编辑系统。
为了达到这一目的,作者使用粘性末端连接酶(NEB)将 Cas9 编码序列与 pMTL83153 载体组装在一起。
另外,gRNA 和修复模板与 pMTL82254 载体重组在一起。修复模板由两个 700 到 1200 bp 的序列组成,靶向 Cas9 负责切除的,约 100 bp 的芽孢杆菌基因组序列。
之后,作者将 gRNA + 修复模板重组质粒导入大肠杆菌中,并通过后者的接合反应(Bacterial Conjugation)将重组质粒转入野生型芽孢杆菌菌体之中。
一段时间之后,作者采用相同的手段再将 cas9 重组质粒转入芽孢杆菌菌体中,从而实现进行基因编辑。
图片来源:Science
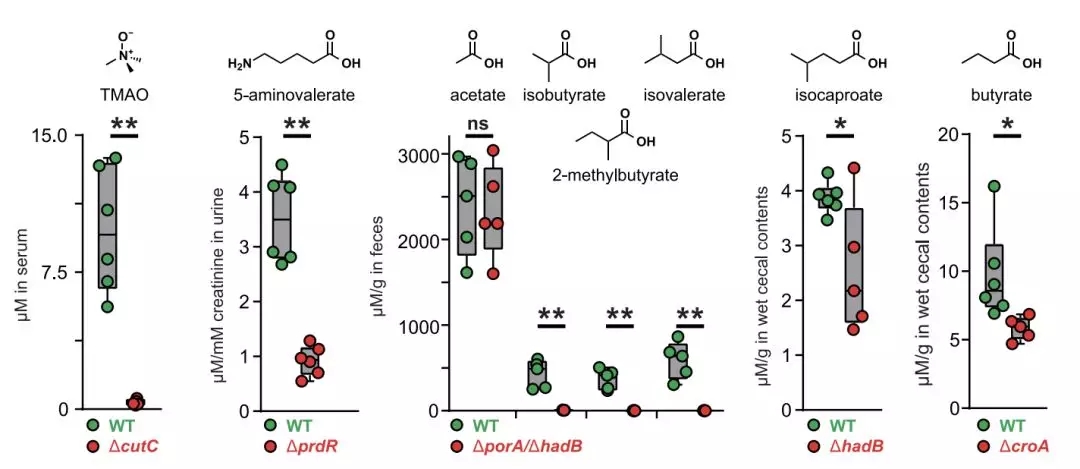
在本文中,作者选择性地敲除了与 10 个代谢产物有关的途径:三甲胺,5 - 氨基戊酸酯,色胺,丙酸吲哚,异戊酸酯,2 - 甲基丁酸酯,异丁酸酯,异丙酸酯,丙酸酯和丁酸酯。
通过液相色谱以及质谱手段,作者验证了突变体菌株中这些代谢产物的缺失,从而确认了基因编辑的效果。
进一步,作者给无菌小鼠(Germ-Free mice)小鼠肠道分别定殖了野生型芽孢杆菌或者上述突变体菌株。
结果表明,野生型细菌在小鼠肠道内能够正常分泌上述代谢产物,并且能够随时间累积,然而突变株则丧失了对应的代谢产物的分泌能力。
下图展示了在定殖了野生型菌株以及不同类型缺失突变株后,无菌小鼠血清中相应代谢产物的水平。
图片来源:Science
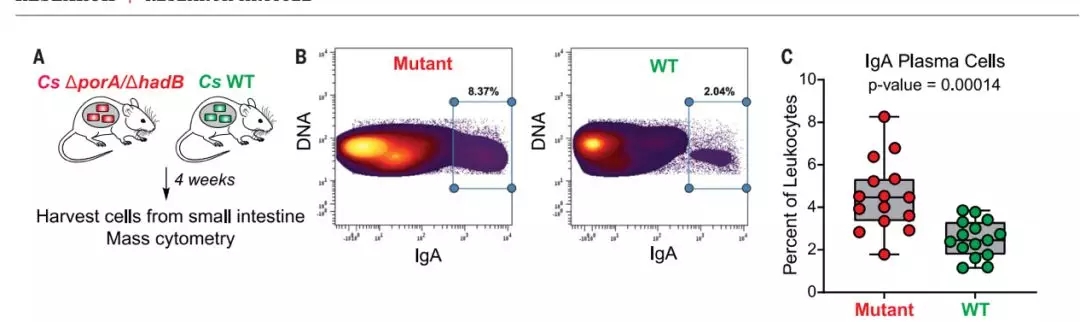
有意思的是,通过比较定殖野生型细菌以及「支链短链脂肪酸(branched SCFA)」缺陷型菌株的小鼠,作者发现:定殖缺陷型细菌的小鼠体内 IgA 的含量明显上升。
由于 IgA 是肠道抗体的主要形式,因此该结果表明小鼠肠道存在一定程度的炎症反应。
结合此前研究揭示的短链脂肪酸在调节宿主免疫反应方面的作用。作者认为支链锻炼脂肪酸的合成缺陷会导致宿主免疫系统过度活化,从而导致炎症反应的发生。
图片来源:Science
2. 宏基因组学与「生物合成基因」聚类分析
不同于 Fischbach 教授等人在微生物组基因编辑这一实验技术方面的革新, Donia 教授更偏爱对已有数据进行分析,从而寻找新的线索。
在本文中,作者等人认识到微生物基因组中存在特殊的基因簇,称为「生物合成基因簇(biosynthetic gene clusters, BGC)」,这一特征使得科学家们能够通过计算的方法预测相关基因的产物。
先前从人类微生物组中寻找 BGC 的研究主要依赖于分析单个「分离株」的参考基因组数据。
尽管这种方法揭示了很多微生物组编码的 BGC 数据,但它并不能反映尚未成功培养或分离的人类微生物组的所有生物合成特征。
对此,该研究采用了数据库中未分类的单个基因组数据,通过解析数据库中的完整基因组以识别生物合成基因的分布。
具体来讲,作者首先着眼于一个易于识别的特征: 在 II 型聚酮化合物合成酶基因簇中存在的一个必不可少的结构域,这类基因簇负责合成一类抗生素类分子,并且与脂肪酸具有共同的起源。
以这种方式,他们发现了 13 个全新的聚酮化合物生物合成基因簇。下图简单描述了 BGC 的发现流程:
首先需要设计一个适用于宏基因组 BGC 特征发现的算法工具(左图),然后利用这一工具对大规模数据库中的基因组数据进行筛选,经过定性、定量以及聚类分析,从而找到潜在的具有相似特征的 BGC。
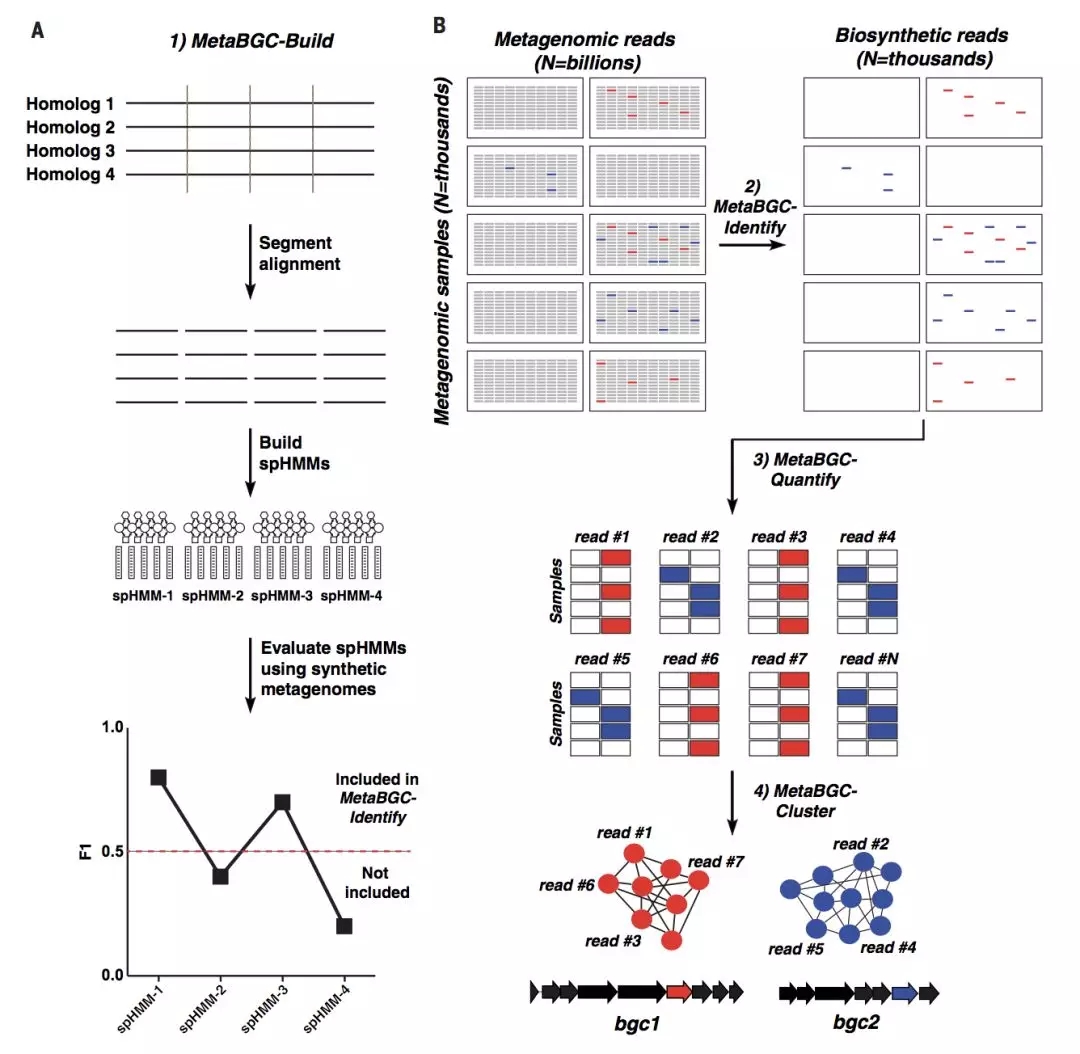
图片来源:Science
之后,作者对数千个临床样本进行筛查,发现在被检测群体中几乎所有人都带有至少一种含有这些 BGC 的细菌;此外,不同的身体部位(即皮肤,肠道,口腔或阴道)存在不同的 BGC。
下图描述了 13 个新发现的 BGC 的遗传组织结构:其中箭头的颜色与上方的指示相对应,用于指征该基因的功能;
此外,图中还显示了 BGC 的体内存在部位,临床样本来源,以及最接近的微生物物种等信息。
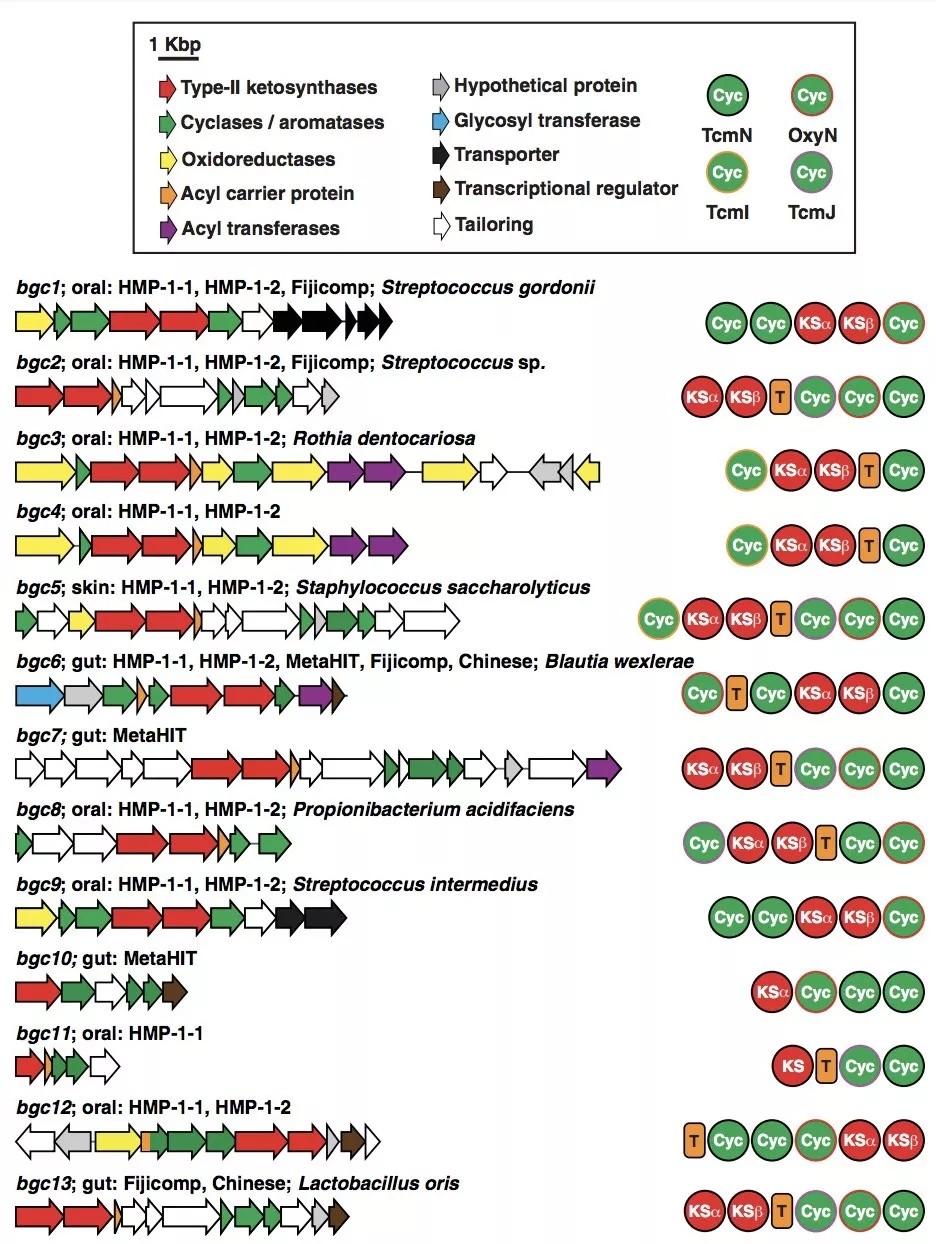
图片来源:Science
接下来,作者利用大规模的合成生物学平台,将新发现的新型 BGC 过表达,并且通过液相色谱 - 质谱手段检测其负责合成的代谢产物类型。
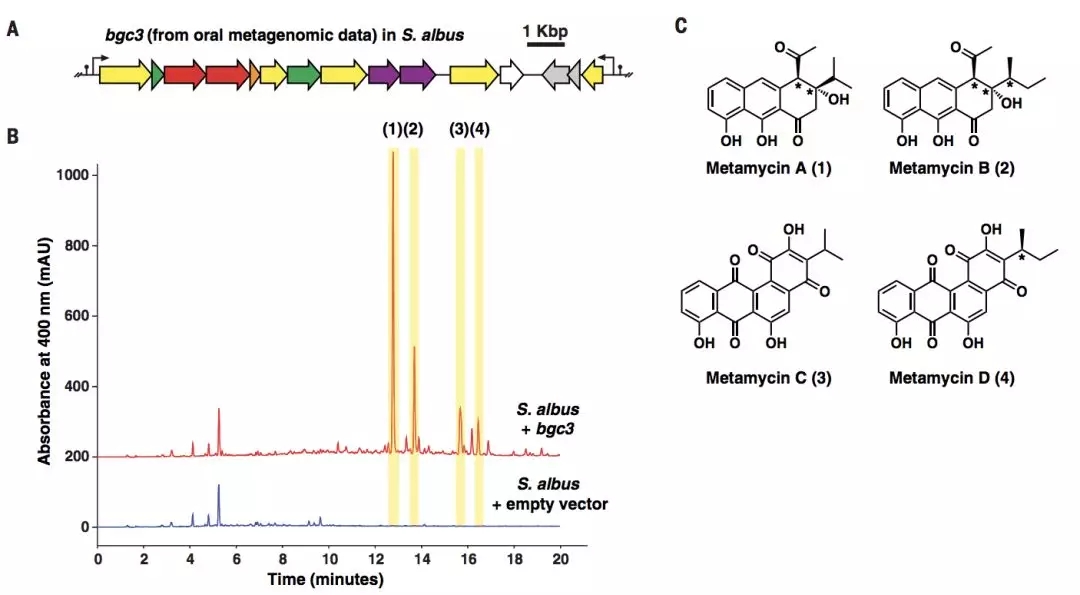
以 bgc3 为例,下图中作者发现在枯草芽孢杆菌中过表达 bgc3 会促进甲霉素的合成,这一结果与计算预测的结果相吻合。
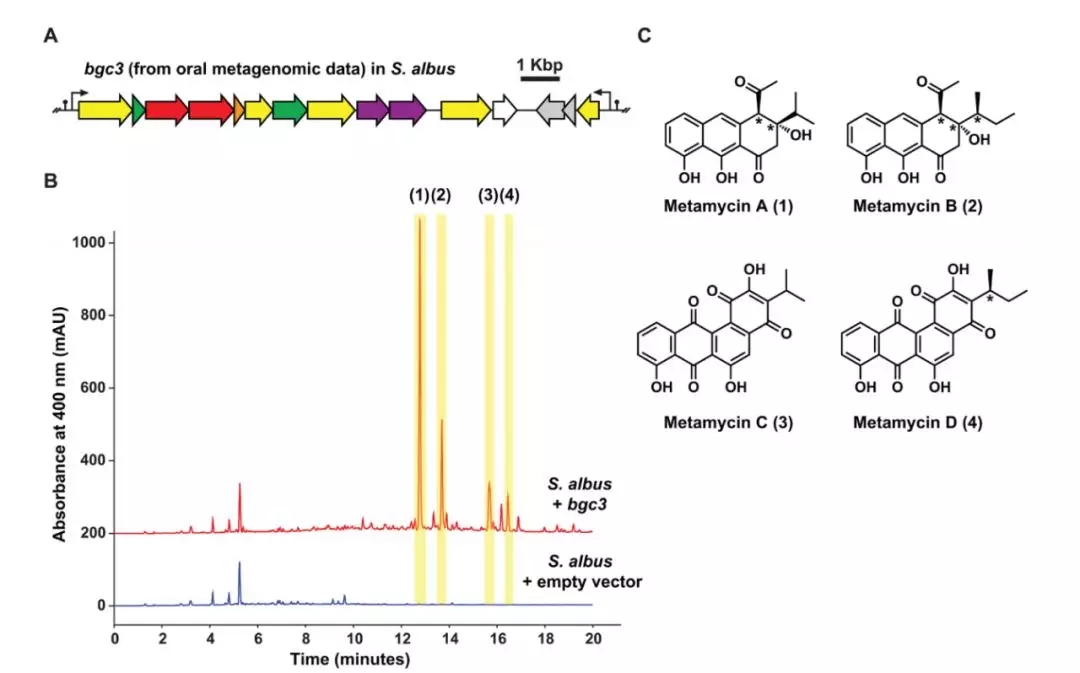
图片来源:Science
该结果表明,这种新型的宏基因组学规模的 BGC 挖掘方法在预测基因代谢产物方面具有高度的准确性。
扩展阅读
Donia 教授的主要研究方向在于通过生物信息学手段发现新的 BGC 以及相关细菌代谢产物。
2014 年,Donia 教授在 Cell 杂志发表论文,首次介绍了利用宏基因组学手段对「人类微生物组计划」中的样本进行分析,并最终得到了 3118 个小分子 BGC。

图片来源:Cell
Fischbach 教授长期致力于肠道微生物代谢产物以及药物开发方面的研究。
2016 年,Fischbach 教授在 Cell Host Microbe 发表文章,描述了通过遗传操纵肠道微生物的基因表达,从而降低宿主血液中有害成分的水平,这将有利于尿毒症患者的症状缓解。

图片来源:Cell Host Microbe
小结
很长时间以来,微生物组学领域的研究仍局限于「相关性分析」。对此,上述两项研究对于引导微生物组学研究向分子层面迈出了重要的一步。
Donia 教授等人的研究表明人体肠道与口腔微生物组会产生临床相关的药物,基于这一认识,将来在筛选小分子库时可能会提供更准确的指导。
另外,基于已有的算法,可以通过反向选择(即排除与已有结构相似的基因簇)寻找新型的生物合成通路,从而发现更多不同结构与功能的细菌代谢产物。
Fischbach 教授等人的研究则提供了一种针对厌氧型肠道微生物的基因编辑工具,这一突破有助于未来更多的微生物组菌株的遗传学操作,从而便于我们深入了解其它基因的代谢产物合成能力以及对宿主的影响。
参考文献
1. Guo C et al. Depletion of microbiome-derived molecules in the host using Clostridium genetics . Science. (2019)
2. Sugimoto Y et al. A metagenomic strategy for harnessing the chemical repertoire of the human microbiome. Science. (2019)
3. Donia MS et al. A systematic analysis of biosynthetic gene clusters in the human microbiome reveals a common family of antibiotics. Cell. 2014 Sep 11;158(6):1402-1414.
4. Devlin AS et al. Modulation of a Circulating Uremic Solute via Rational Genetic Manipulation of the Gut Microbiota. Cell Host Microbe. 2016.14;20(6):709-715.