微生物多样性—16S/18S/ITS测序
研选同类产品更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 用户评价
- 文献和实验
- 技术资料
微生物多样性测序技术是将环境样本中全部微生物的DNA获取后,根据自己的需求对细菌、真菌、功能基因等特异性的区域(16S rDNA/ITS/18S/nifH等)进行PCR扩增,混样建库,并基于高通量测序平台,研究环境样本中可分离培养和不可分离培养的微生物的种类和丰度差异。可分为细菌多样性、真菌多样性和功能基因多样性。在土壤、水体、空气、污泥、粪便、肠道、工业发酵液、生物膜、拭子、口腔、皮肤、昆虫、动植物内生菌等相关领域被广泛应用。
产品优势
一次性获得可培养和不可培养物种多样性信息
20余种样本类型提取建库测序分析项目经验
高水平分析团队,全面的分析内容,专业的售后服务
技术路线

结果展示
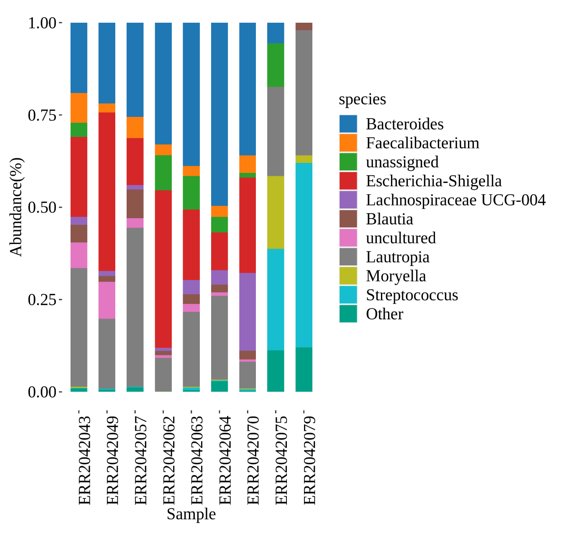
物种分布柱状图
根据物种注释结果,在门(Phylum)、纲(Class)、目(Order)、科(Family)、属(Genus)不同的分类学水平上展示不同样本中的优势菌群及丰度排名前十的物种,可以直观的反馈出样本间物种和丰度的差异。
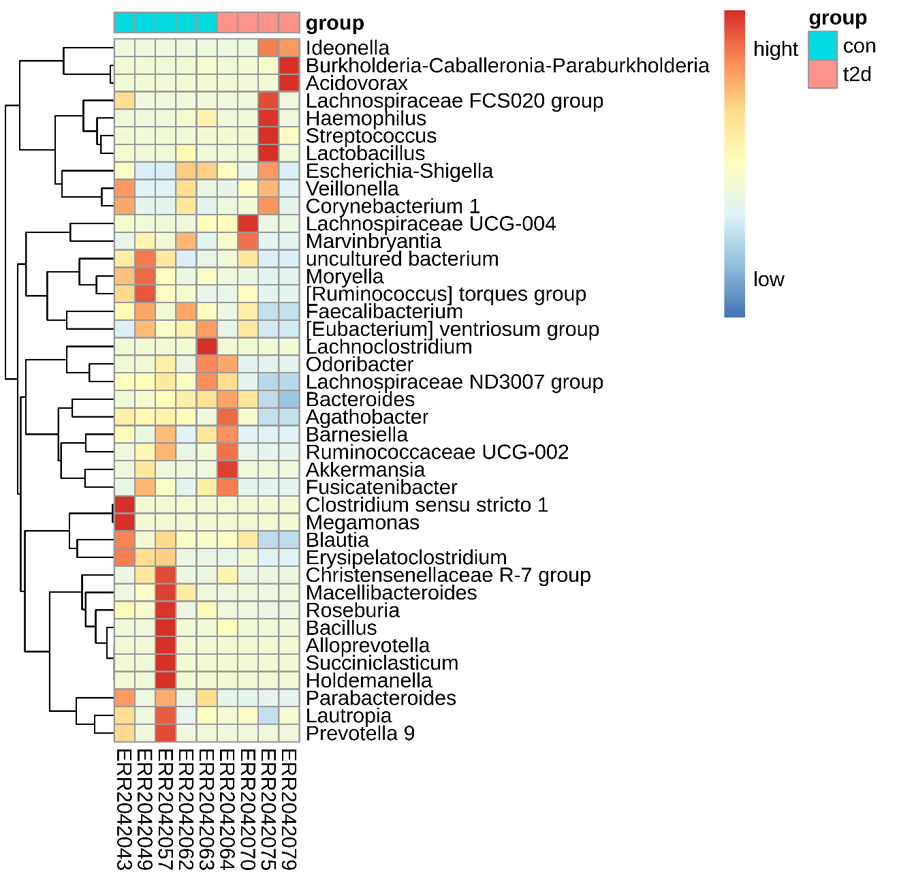
物种聚类热图
热图聚类结果中,颜色代表物种丰度。纵向聚类表示不同物种在各样品间丰度的相似情况,横向聚类表示不同样品的各物种丰度的相似情况,直观反馈同一物种在不同样本中的丰度差异。
Alpha 多样性指数
Alpha多样性(Alpha diversity)反映的是单个样品物种丰度及物种多样性,有多种衡量指标:Chao1、Ace、Shannon、Simpson、PD等。Chao1和Ace指数衡量物种丰度即物种数量的多少。Shannon和Simpson指数用于衡量物种多样性,受样品群落中物种丰度和物种均匀度的影响。

β 多样性分析
使用QIIME软件进行 Beta 多样性(Beta diversity)分析,比较不同样品在物种多样性方面存在的相似程度。Beta多样性分析主要采用 binary jaccard、 bray curtis、 weighted unifrac(限细菌)、 unweighted unifrac (限细菌)等4种算法计算样品间的距离从而获得样本间的β值。

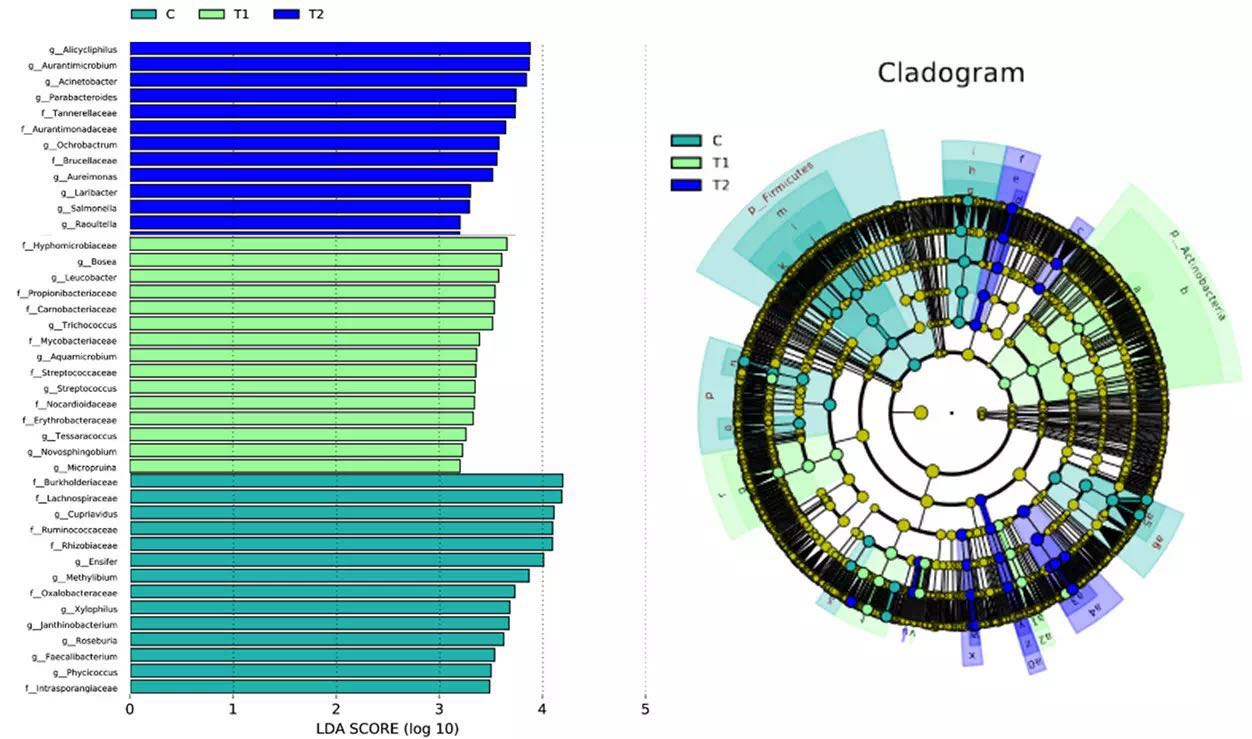
组间显著性差异分析
LEfSe(Line Discriminant Analysis (LDA) Effect Size) 分析:寻找不同组中,每组中显著性差别与其他组的物种,可视为该组样品中的biomarker物种。下图为LDA值分布柱状图和LEfSe分析进化分枝图。
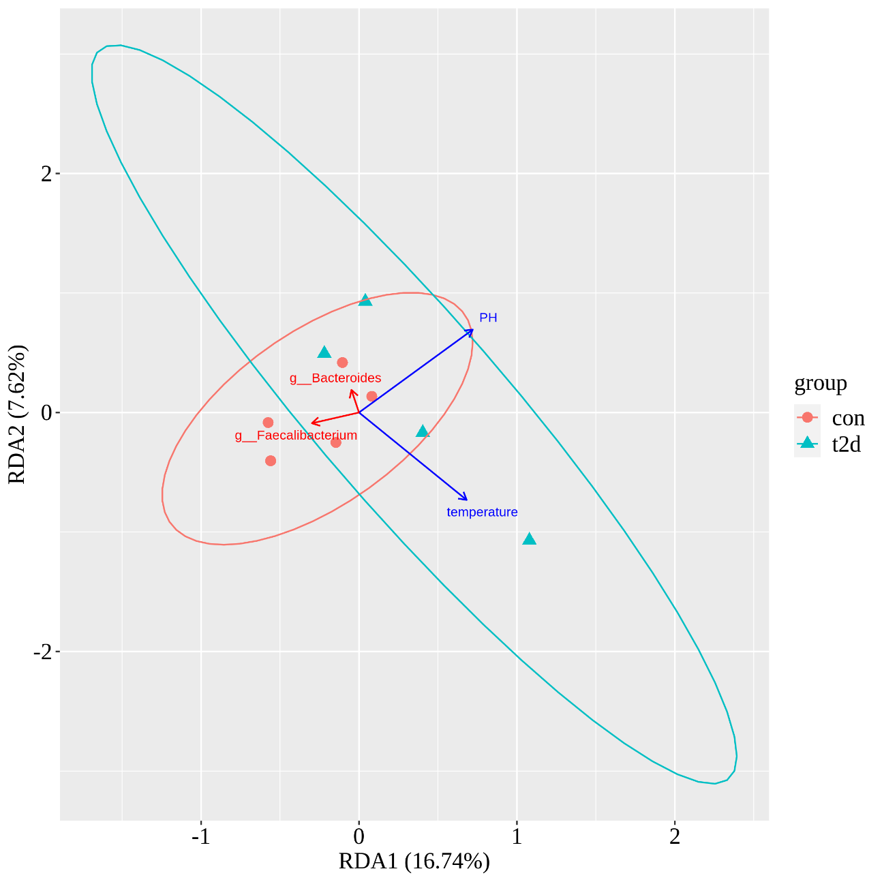
RDA/CCA分析
检测环境因子、样品、菌群三者之间的关系或者两两之间的关系,常用的分析方法有CCA和RDA两种。RDA是基于线性模型,CCA是基于单峰模型。右图是RDA分析,箭头代表不同的环境因子,箭头连线之间的夹角的代表其相关性。
常见问题
Q1、微生物多样性的研究目的
A:主要关注环境样本中物种的组成和丰度,不同样本中物种种类差异和物种丰度差异。
Q2、什么是16S rDNA?
A:16S rDNA编码原核核糖体小亚基 rRNA的DNA序列。在结构上分为10个保守区和9个可变区。保守区反映生物物种间的亲缘关系,可变区反映物种间的差异。
Q3、什么是ITS?
A:ITS是编码真核生物核糖体小亚基rRNA的DNA内转录间隔区序列。由于ITS区在核糖体RNA加工过程中被剪切掉,不发挥功能作用,在进化过程中选择压力较小,进化速率约为18S rDNA的10倍,属于中度保守的区域,利用它可研究种及种以下的分类阶元。
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 用户评价
用户评价 暂无用户评价
暂无用户评价 文献和实验
文献和实验Infants born to mothers with IBD present with altered gut microbiome that transfers abnormalities of the adaptive immune system to germ-free mice. Gut,2019,DOI: 10.1136/gutjnl-2018-317855.
Root exudates drive the soil-borne legacy of aboveground pathogen infection.Microbiome,2018,doi.org/10.1186/s40168-018-0537-x
8 小时限时饮食,可改善高脂饮食导致的肥胖和代谢问题!肠道菌群发挥了关键作用
为了研究高脂饮食(HFD)和限制时间饮食(TRF)对宿主回肠循环活动的影响,他们将小鼠分为三组:限时高脂饮食组(FT)、自由高脂饮食组(FA)和正常饮食组(NA)。喂养 8 周后,研究人员在 24 小时内每 4 h 收集全回肠样本,以研究肠腔和粘附细菌的日间动态。尽管摄入的食物热量相同,TRF 仍能改善与高脂饮食诱导的体重和血糖水平。 经过对宿主样本进行 16S 扩增子测序,发现回肠菌群的组成可因饲喂方式不同而不同,高脂饮食可显著降低回肠菌群的 a-多样性和 β-多样性。限时高脂饮食组和自由高脂
盒,他们利用机械和化学裂解以及DNA纯化步骤,接着对16S rRNA基因进行PCR扩增,最后测序 。但是他们发现,并非所有方法都是相似的,这不但影响结果的质量,还让不同研究之间的结果难以比较。为了解决这些挑战,多个研究小组都在优化DNA提取的效率,并研究不同步骤如何影响DNA的质量和产量。 例如,在上月发表在《PLoS ONE》的一项研究中,科罗拉多大学的微生物学家Gilberto Flores及其同事检验了一种由Sigma-Aldrich提供的直接PCR方法(Extract-N-Amp
范围为15 ℃~80 ℃,较大片段DNA 的Tm 值因为接近80 ℃而使检测难度提高;若电泳条件不适宜,不能完全保证将有序列差异的DNA片段分开,从而出现序列不同的DNA 迁移在同一位置的现象。 3、高通量测序方法 近年来,16S rRNA/DNA为基础的分子生物学技术已成为普遍接受的方法[6,7 ]。研究表明,400~600碱基的序列,足以对环境中微生物的多样性和种群分类进行初步的估计[8],因此454高通量测序的方法因其读长(400~500bp)长和准确性高的特点大量用于微生物多样性
 技术资料
技术资料暂无技术资料 索取技术资料