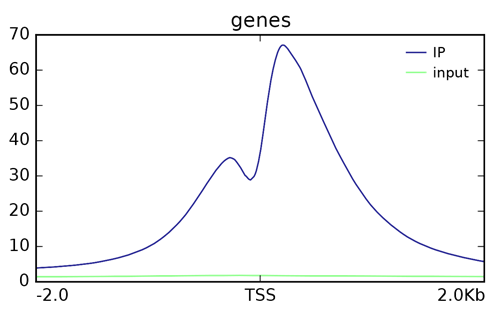
ChIP作图千万张,Reads分布第一张
武汉康测科技有限公司
很多老师关心自己研究的某些关键基因上是否有目的蛋白结合或者是否有组蛋白修饰,别着急,小编今天有备而来,就给大家讲这个。
reads在Peak关联基因上的分布图,是选取筛选到Peak的基因,进行作图,展示比对到该基因的reads的覆盖深度分布,通俗的说,就是该基因上目的蛋白的结合分布,横坐标显示基因位置,纵坐标表示reads数(reads count)。
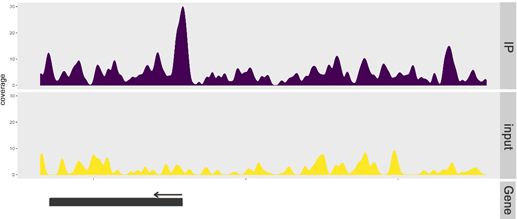
这是项目KC2018-CXX,植物某转录因子ChIP测序的一张reads在Peak关联基因上的分布图,对比IP和input可知,在该基因TSS附近及下游IP显著富集,此处为该转录因子的结合区域。
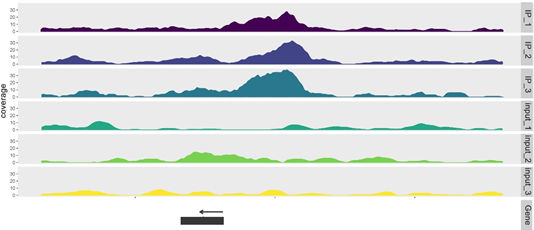
另一个项目KC2018-CXX,动物某转录因子ChIP测序,三个生物学重复,从reads在Peak关联基因上的分布图可以看出,该转录因子主要结合这个基因TSS上游的一段序列。
这张图也是文章中露脸频率非常高的图,放上这张图,就能省掉上千字的解说,还能直线拉升文章档次,啧啧啧。

两组(橙色和蓝色)样本组蛋白修饰在基因CREG1上游的分布比较,橙色样本的CREG1上游组蛋白修饰显著富集。(图片来源:Neuroblastoma is composed of two super-enhancer associated differentiation states)
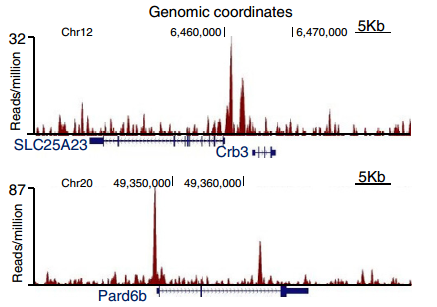
转录因子ZEB1在基因上的结合分布(图片来源:Zeb1 potentiates genome-wide gene transcription
with Lef1 to promote glioblastoma cell invasion)