万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 英文名:
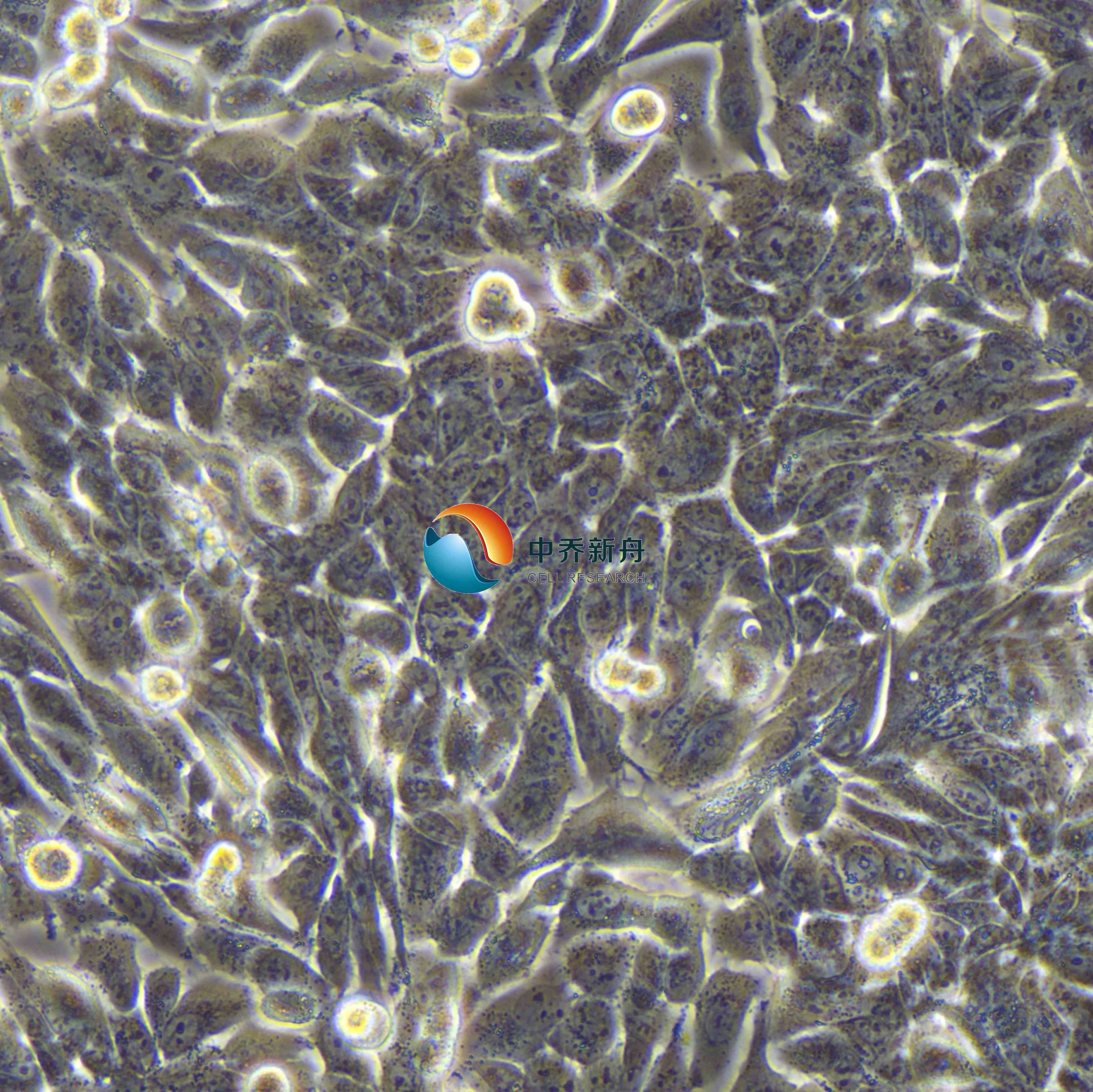
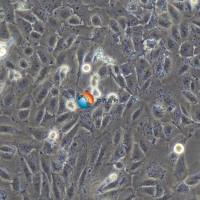
NCI-H358
- 库存:
大量
- 供应商:
中乔新舟
- 细胞类型:
细胞系
- 品系:
human
- 组织来源:
human
- 相关疾病:
否
- 物种来源:
human
- 免疫类型:
否
- 细胞形态:
咨询销售
- 器官来源:
human
- 运输方式:
T25瓶运输
- 年限:
5-10年
- 生长状态:
贴壁生长
- 规格:
5 x 10^5 cells/vial
|
产品名称 |
NCI-H358人非小细胞肺癌细胞 |
|
货号 |
ZQ0384 |
|
产品介绍 |
NCI-H358,也被称为H-358或NCIH358,是一种上皮样细胞系,来源于一名非小细胞肺癌(NSCLC)亚型细支气管肺泡癌患者。超微结构研究表明细胞质中有Clara细胞的特征结构 细胞表达主要的肺表面结合蛋白SP-A的蛋白和RNA。 不表达SP-B和SP-C。 他们在软琼脂中的克隆形成效率为0.83%。NCI-H358细胞在以非小细胞肺癌为重点的癌症研究中尤其重要,特别是在探索肺腺癌的生物学和治疗方面。 |
|
种属 |
人 |
|
性别/年龄 |
男 |
|
组织 |
肺/支气管;来源于转移部位:肺泡 |
|
疾病 |
细支气管肺泡癌;非小细胞肺癌 |
|
细胞类型 |
肿瘤细胞 |
|
形态学 |
上皮细胞 |
|
生长方式 |
贴壁 |
|
倍增时间 |
大约38~76小时 |
|
培养基和添加剂 |
RPMI-1640(品牌:中乔新舟 货号:ZQ-200)+10%FBS(品牌:中乔新舟 货号:ZQ500-A)+1%P/S(中乔新舟 货号:CSP006) |
|
推荐完全培养基货号 |
ZM0384 |
|
生物安全等级 |
BSL-1 |
|
STR位点信息 |
Amelogenin: X,Y CSF1PO: 11,12 D13S317: 8,12 D16S539: 12,13 D5S818: 10,12 D7S820: 10,11 TH01: 6 TPOX: 8,9 vWA: 17 |
|
培养条件 |
95%空气,5%二氧化碳;37℃ |
|
抗原表达/受体表达 |
*** |
|
基因表达 |
*** |
|
保藏机构 |
ATCC; CRL-5807 ECACC; 95111733 |
|
供应限制 |
仅供科研使用 |
上海中乔新舟生物科技有限公司成立于2011年,历经十多年发展,主要专注于细胞生物学产品的研究和开发,专注于为药企、各类科研机构及CRO企业提供符合标准规范的细胞培养服务、细胞培养基、细胞检测试剂盒、细胞培养试剂,胎牛血清和细胞生物学技术服务等。
公司一直致力于为高等院校、研究机构、医院、CRO及CDMO企业提供细胞培养完整解决方案,这些产品旨在满足细胞培养的多样需求,确保实验和研究的有效进行。引用中乔新舟(ZQXZBIO)产品和服务的文献超数千篇。

产品服务
细胞资源:原代细胞、细胞株、干细胞、示踪细胞、耐药株细胞、永生化细胞等基因工程细胞。
试剂产品:胎牛血清、完全培养基(适用于原代细胞及细胞株)、无血清培养基、基础培养基、细胞转染试剂、重组因子、胰酶和双抗等等细胞培养所有实验相关产品。
技术服务:稳转株构建、原代细胞分离、特殊培养基定制服务、细胞检测等。

目前产品已经畅销国内30多个省市,与客户建立长期的合作伙伴关系,共同实现成功。全体员工将不懈努力,继续为科研人员提供优良的产品和服务,致力成为全球细胞培养领域的参与者。
企业愿景
致力于成为国内细胞培养基产业的佼佼者,生物医药领域上游原材料的优良提供商。
企业使命
成长为专业细胞系及原代细胞培养供应商、专业细胞培养基及培养试剂生产商。
企业荣誉


风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验论文标题: NEDD4L-induced ubiquitination mediating UBE2T degradation inhibits progression of lung adenocarcinoma via PI3K-AKT signaling
DOI: 10.1186/s12935-021-02341-9
发表时间: 2021-11-27
期刊: Cancer Cell International
影响因子: 5.722
货号: ZQ0384
产品名称: H358 cells
论文标题: LncRNA PCBP1-AS1 correlated with the functional states of cancer cells and inhibited lung adenocarcinoma metastasis by suppressing the EMT progression
DOI: 10.1093/carcin/bgab047
发表时间: 2021-07-16
期刊: CARCINOGENESIS
影响因子: 4.944
货号: ZQ0384
产品名称: H358 cells
论文标题: High expression of 14‑3‑3ơ indicates poor prognosis and progression of lung adenocarcinoma
DOI: 10.3892/ol.2022.13323
发表时间: 2022-05-12
期刊: Oncology Letters
影响因子: 2.967
货号: ZQ0384
产品名称: NCI-H358 cells
论文标题: Loading MicroRNA-376c in Extracellular Vesicles Inhibits Properties of Non-Small Cell Lung Cancer Cells by Targeting YTHDF1:
DOI: 10.1177/1533033820977525
发表时间: 2020-12-07
期刊: TECHNOLOGY IN CANCER RESEARCH & TREATMENT
影响因子: 2.074
货号: ZQ0384
产品名称: NCI-H358 cells
PubMed=3940644
Brower M., Carney D.N., Oie H.K., Gazdar A.F., Minna J.D.
Growth of cell lines and clinical specimens of human non-small cell lung cancer in a serum-free defined medium.
Cancer Res. 46:798-806(1986)
PubMed=3335022
Alley M.C., Scudiero D.A., Monks A., Hursey M.L., Czerwinski M.J., Fine D.L., Abbott B.J., Mayo J.G., Shoemaker R.H., Boyd M.R.
Feasibility of drug screening with panels of human tumor cell lines using a microculture tetrazolium assay.
Cancer Res. 48:589-601(1988)
PubMed=2388294; DOI=10.1093/jnci/82.17.1420
McLemore T.L., Litterst C.L., Coudert B.P., Liu M.C., Hubbard W.C., Adelberg S., Czerwinski M.J., McMahon N.A., Eggleston J.C., Boyd M.R.
Metabolic activation of 4-ipomeanol in human lung, primary pulmonary carcinomas, and established human pulmonary carcinoma cell lines.
J. Natl. Cancer Inst. 82:1420-1426(1990)
PubMed=2386953
Gazdar A.F., Linnoila R.I., Kurita Y., Oie H.K., Mulshine J.L., Clark J.C., Whitsett J.A.
Peripheral airway cell differentiation in human lung cancer cell lines.
Cancer Res. 50:5481-5487(1990)
PubMed=1311061
Mitsudomi T., Steinberg S.M., Nau M.M., Carbone D.P., D'Amico D., Bodner S.M., Oie H.K., Linnoila R.I., Mulshine J.L., Minna J.D., Gazdar A.F.
p53 gene mutations in non-small-cell lung cancer cell lines and their correlation with the presence of ras mutations and clinical features.
Oncogene 7:171-180(1992)
PubMed=1565469
Bodner S.M., Minna J.D., Jensen S.M., D'Amico D., Carbone D.P., Mitsudomi T., Fedorko J., Buchhagen D.L., Nau M.M., Gazdar A.F., Linnoila R.I.
Expression of mutant p53 proteins in lung cancer correlates with the class of p53 gene mutation.
Oncogene 7:743-749(1992)
PubMed=7972006; DOI=10.1073/pnas.91.23.11045
Okamoto A., Demetrick D.J., Spillare E.A., Hagiwara K., Hussain S.P., Bennett W.P., Forrester K., Gerwin B.I., Serrano M., Beach D.H., Harris C.C.
Mutations and altered expression of p16INK4 in human cancer.
Proc. Natl. Acad. Sci. U.S.A. 91:11045-11049(1994)
PubMed=8806092; DOI=10.1002/jcb.240630505
Phelps R.M., Johnson B.E., Ihde D.C., Gazdar A.F., Carbone D.P., McClintock P.R., Linnoila R.I., Matthews M.J., Bunn P.A. Jr., Carney D.N., Minna J.D., Mulshine J.L.
NCI-Navy Medical Oncology Branch cell line data base.
J. Cell. Biochem. 63 Suppl. 24:32-91(1996)
PubMed=8806101; DOI=10.1002/jcb.240630514
Lokshin A.E., Levitt M.L.
Effect of suramin on squamous differentiation and apoptosis in three human non-small-cell lung cancer cell lines.
J. Cell. Biochem. 63 Suppl. 24:186-197(1996)
PubMed=9649128; DOI=10.1038/bjc.1998.361
Yi S., Chen J.-R., Viallet J., Schwall R.H., Nakamura T., Tsao M.-S.
Paracrine effects of hepatocyte growth factor/scatter factor on non-small-cell lung carcinoma cell lines.
Br. J. Cancer 77:2162-2170(1998)
PubMed=11030152; DOI=10.1038/sj.onc.1203815
Modi S., Kubo A., Oie H.K., Coxon A.B., Rehmatulla A., Kaye F.J.
Protein expression of the RB-related gene family and SV40 large T antigen in mesothelioma and lung cancer.
Oncogene 19:4632-4639(2000)
PubMed=11369051; DOI=10.1016/S0165-4608(00)00363-0
Luk C., Tsao M.-S., Bayani J., Shepherd F.A., Squire J.A.
Molecular cytogenetic analysis of non-small cell lung carcinoma by spectral karyotyping and comparative genomic hybridization.
Cancer Genet. Cytogenet. 125:87-99(2001)
PubMed=11464291; DOI=10.1038/sj.onc.1204557
Shigemitsu K., Sekido Y., Usami N., Mori S., Sato M., Horio Y., Hasegawa Y., Bader S.A., Gazdar A.F., Minna J.D., Hida T., Yoshioka H., Imaizumi M., Ueda Y., Takahashi M., Shimokata K.
Genetic alteration of the beta-catenin gene (CTNNB1) in human lung cancer and malignant mesothelioma and identification of a new 3p21.3 homozygous deletion.
Oncogene 20:4249-4257(2001)
PubMed=12068308; DOI=10.1038/nature00766
Davies H., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
PubMed=18083107; DOI=10.1016/j.cell.2007.11.025
Rikova K., Guo A.-L., Zeng Q.-F., Possemato A., Yu J., Haack H., Nardone J., Lee K., Reeves C., Li Y., Hu Y.-R., Tan Z.-P., Stokes M.P., Sullivan L., Mitchell J., Wetzel R., MacNeill J., Ren J.-M., Yuan J., Bakalarski C.E., Villen J., Kornhauser J.M., Smith B., Li D.-Q., Zhou X.-M., Gygi S.P., Gu T.-L., Polakiewicz R.D., Rush J., Comb M.J.
Global survey of phosphotyrosine signaling identifies oncogenic kinases in lung cancer.
Cell 131:1190-1203(2007)
PubMed=19472407; DOI=10.1002/humu.21028
Blanco R., Iwakawa R., Tang M.-Y., Kohno T., Angulo B., Pio R., Montuenga L.M., Minna J.D., Yokota J., Sanchez-Cespedes M.
A gene-alteration profile of human lung cancer cell lines.
Hum. Mutat. 30:1199-1206(2009)
PubMed=20164919; DOI=10.1038/nature08768
Bignell G.R., Greenman C.D., Davies H., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20557307; DOI=10.1111/j.1349-7006.2010.01622.x
Iwakawa R., Kohno T., Enari M., Kiyono T., Yokota J.
Prevalence of human papillomavirus 16/18/33 infection and p53 mutation in lung adenocarcinoma.
Cancer Sci. 101:1891-1896(2010)
PubMed=22460905; DOI=10.1038/nature11003
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22961666; DOI=10.1158/2159-8290.CD-12-0112
Byers L.A., Wang J., Nilsson M.B., Fujimoto J., Saintigny P., Yordy J., Giri U., Peyton M., Fan Y.-H., Diao L.-X., Masrorpour F., Shen L., Liu W.-B., Duchemann B., Tumula P., Bhardwaj V., Welsh J., Weber S., Glisson B.S., Kalhor N., Wistuba I.I., Girard L., Lippman S.M., Mills G.B., Coombes K.R., Weinstein J.N., Minna J.D., Heymach J.V.
Proteomic profiling identifies dysregulated pathways in small cell lung cancer and novel therapeutic targets including PARP1.
Cancer Discov. 2:798-811(2012)
PubMed=24135919; DOI=10.1038/ncomms3617
Balbin O.A., Prensner J.R., Sahu A., Yocum A., Shankar S., Malik R., Fermin D., Dhanasekaran S.M., Chandler B., Thomas D., Beer D.G., Cao X.-H., Nesvizhskii A.I., Chinnaiyan A.M.
Reconstructing targetable pathways in lung cancer by integrating diverse omics data.
Nat. Commun. 4:2617.1-2617.13(2013)
都是贴壁细胞,在消化传代过程中,步骤如下:倒尽旧的培养液->用无血清的培养基清洗一两次->加入一定量的胰酶,置于37度培养箱中5--10分钟,使细胞悬浮->显微镜下观察,待细胞大部分变圆时,回到超静台->加入一定量的含血清的新培养液,以终止胰酶作用->反复吹打细胞->再置显微镜下观察,直到细胞全部悬浮起来->吸出一部分加入新的培养瓶中->最后再补充加入一定量新的培养液。注意: 1、吹细胞时尽量多吹边角儿,此处细胞生长的多。2、吸出细胞前要混匀,可以剧烈震荡培养瓶。3、我们用的是DMEM
CD44变异体V6、V7/8在非小细胞肺癌中的表达及临床意义
CD44变异体V 6 、V 7/8 在非小细胞肺癌中的表达及临床意义 第三军医大学学报2000年第22卷第7期 孙焰 陈幸华 罗平 贺光友 摘 要 : 目的 探讨CD44 v 6 、V 7/8 与非小细胞肺癌(Non-small cell lung cancer,NSCLC)患者的淋巴结转移及预后的关系。 方法 采用免疫组化方法观察CD44 v 6 、V 7/8 在76例原发性非小细胞肺癌
每年的 11 月 17 日被定为「国际肺癌宣传日」,旨在呼吁世界各国政府重视肺癌的预防,提高人们对肺癌的防治意识,普及肺癌的规范化诊疗知识。肺癌是我国及世界范围内发病率和死亡率较高的恶性肿瘤之一,一般分非小细胞肺癌(non small cell lung cancer, NSCLC)和小细胞肺癌(small cell lung cancer,SCLC)两种亚型,其中非小细胞肺癌约占肺癌的 80~85%[1]。 图 1. 2015 年中国肿瘤发病率统计 目前,已发现的肺癌驱动基因已达数十
 技术资料
技术资料暂无技术资料 索取技术资料