相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 英文名:
MDA-MB-435S
- 库存:
大量
- 供应商:
中乔新舟
- 细胞类型:
细胞系
- 品系:
human
- 组织来源:
human
- 相关疾病:
否
- 物种来源:
human
- 免疫类型:
否
- 细胞形态:
咨询销售
- 器官来源:
human
- 运输方式:
T25瓶运输
- 年限:
5-10年
- 生长状态:
贴壁生长
- 规格:
5 x 10^5 cells/vial
|
产品名称 |
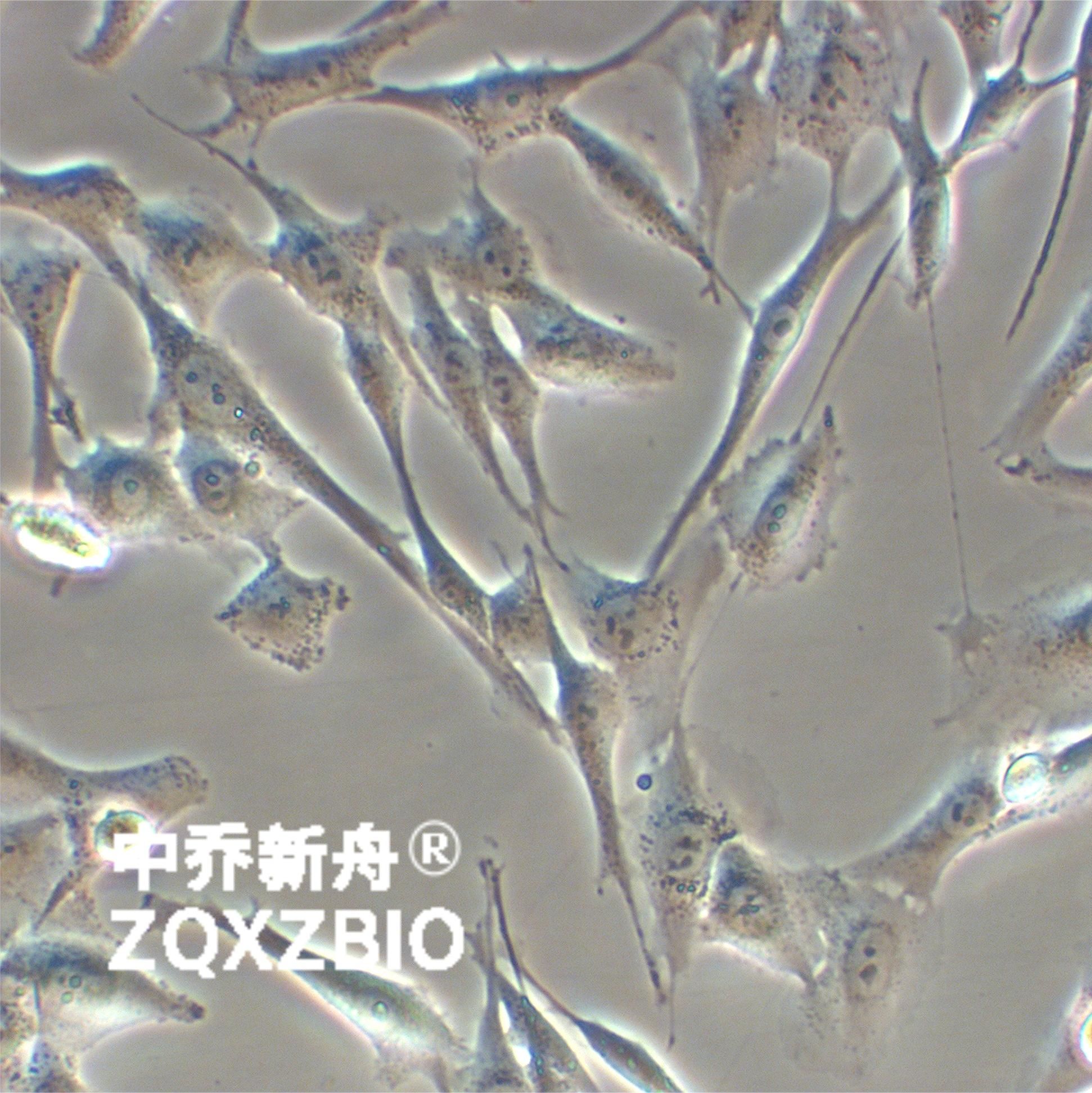
MDA-MB-435S人黑素瘤细胞 |
|
货号 |
ZQ0374 |
|
产品介绍 |
该细胞系最初被描述为 R. Cailleau 等人于 1976 年分离的亲本 MDA-MB-435 菌株的纺锤形变体。来自一名患有转移性乳腺导管腺癌的 31 岁女性的胸腔积液。 然而,最近的研究对亲本细胞系 MDA-MB-435 以及延伸的 HTB-129 的起源产生了疑问。对细胞的基因表达分析产生了微阵列,其中 MDA-MB-435 与黑色素瘤来源的细胞系聚集在一起。 此后的更多研究证实了 MDA-MB-435 的黑素细胞起源,ATCC 对此做出了回应,对该细胞系的身份进行了自己的调查。据报道,MDA-MB-435 交叉污染的细胞系是 M14 黑色素瘤细胞系 [PubMed ID:12354931 和 PubMed ID:17004106]。 |
|
种属 |
人 |
|
性别/年龄 |
女/31岁 |
|
组织 |
先前描述为:乳腺/乳房;来源于转移部位:胸腔积液 |
|
疾病 |
无色素性黑色素瘤 |
|
细胞类型 |
黑色素细胞,黑色素瘤 |
|
形态学 |
纺锤形 |
|
生长方式 |
贴壁 |
|
倍增时间 |
大约48~72小时 |
|
培养基和添加剂(默认) |
DMEM (中乔新舟 货号:ZQ-100)+10%FBS(中乔新舟 货号:ZQ0500)+1%双抗(中乔新舟 货号:CSP006) 注意默认方案培养条件:95%空气,5%CO2;37℃ |
|
培养方案B(可选) |
L-15(中乔新舟 货号:ZQ-1100)+10%FBS(中乔新舟 货号:ZQ500-A)+1%P/S(中乔新舟 货号:CSP006)+0.01mg/mL牛胰岛素+0.01mg/ml谷胱甘肽 注意B方案培养条件:100%空气;37℃ |
|
推荐默认完全培养基货号 |
ZM0374 |
|
B方案完全培养基货号 |
ZM0374-B |
|
生物安全等级 |
BSL-1 |
|
STR位点信息 |
Amelogenin: X CSF1PO: 11 D13S317: 12 D16S539: 13 D5S818: 12 D7S820: 8,10 TH01: 6,7 TPOX: 8,11 vWA: 16,18 |
|
默认方案培养条件 |
95%空气,5%CO2;37℃ |
|
抗原表达/受体表达 |
*** |
|
基因表达 |
*** |
|
保藏机构 |
ATCC; HTB-129 |
|
供应限制 |
仅供科研使用 |
上海中乔新舟生物科技有限公司成立于2011年,历经十多年发展,主要专注于细胞生物学产品的研究和开发,专注于为药企、各类科研机构及CRO企业提供符合标准规范的细胞培养服务、细胞培养基、细胞检测试剂盒、细胞培养试剂,胎牛血清和细胞生物学技术服务等。
公司一直致力于为高等院校、研究机构、医院、CRO及CDMO企业提供细胞培养完整解决方案,这些产品旨在满足细胞培养的多样需求,确保实验和研究的有效进行。引用中乔新舟(ZQXZBIO)产品和服务的文献超数千篇。

产品服务
细胞资源:原代细胞、细胞株、干细胞、示踪细胞、耐药株细胞、永生化细胞等基因工程细胞。
试剂产品:胎牛血清、完全培养基(适用于原代细胞及细胞株)、无血清培养基、基础培养基、细胞转染试剂、重组因子、胰酶和双抗等等细胞培养所有实验相关产品。
技术服务:稳转株构建、原代细胞分离、特殊培养基定制服务、细胞检测等。

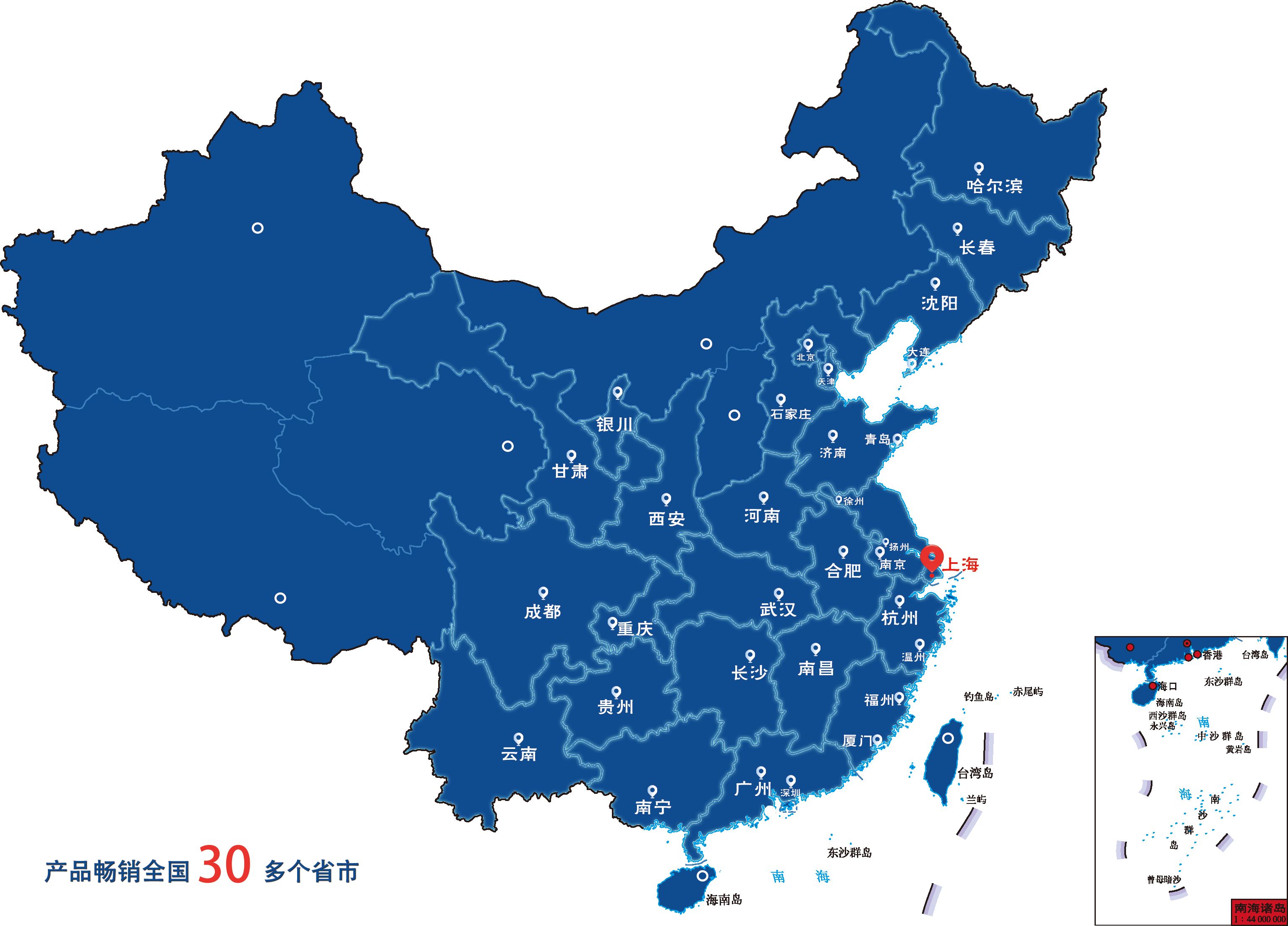
目前产品已经畅销国内30多个省市,与客户建立长期的合作伙伴关系,共同实现成功。全体员工将不懈努力,继续为科研人员提供优良的产品和服务,致力成为全球细胞培养领域的参与者。
企业愿景
致力于成为国内细胞培养基产业的佼佼者,生物医药领域上游原材料的优良提供商。
企业使命
成长为专业细胞系及原代细胞培养供应商、专业细胞培养基及培养试剂生产商。
企业荣誉


风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验PubMed=2007622; DOI=10.1083/jcb.113.1.173
Frixen U.H., Behrens J., Sachs M., Eberle G., Voss B., Warda A., Lochner D., Birchmeier W.
E-cadherin-mediated cell-cell adhesion prevents invasiveness of human carcinoma cells.
J. Cell Biol. 113:173-185(1991)
DOI=10.1016/B978-0-12-333530-2.50009-5
Leibovitz A.
Cell lines from human breast.
(In) Atlas of human tumor cell lines; Hay R.J., Park J.-G., Gazdar A.F. (eds.); pp.161-184; Academic Press; New York (1994)
PubMed=10700188; DOI=10.1038/73536
Gayther S.A., Batley S.J., Linger L., Bannister A.J., Thorpe K., Chin S.-F., Daigo Y., Russell P., Wilson A., Sowter H.M., Delhanty J.D.A., Ponder B.A.J., Kouzarides T., Caldas C.
Mutations truncating the EP300 acetylase in human cancers.
Nat. Genet. 24:300-303(2000)
PubMed=12354931; DOI=10.1136/mp.55.5.294
Ellison G., Klinowska T., Westwood R.F.R., Docter E., French T., Fox J.C.
Further evidence to support the melanocytic origin of MDA-MB-435.
Mol. Pathol. 55:294-299(2002)
PubMed=12661003; DOI=10.1002/gcc.10196
Seitz S., Wassmuth P., Plaschke J., Schackert H.K., Karsten U., Santibanez-Koref M.F., Schlag P.M., Scherneck S.
Identification of microsatellite instability and mismatch repair gene mutations in breast cancer cell lines.
Genes Chromosomes Cancer 37:29-35(2003)
PubMed=15677628; DOI=10.1093/carcin/bgi032
Gorringe K.L., Chin S.-F., Pharoah P.D.P., Staines J.M., Oliveira C., Edwards P.A.W., Caldas C.
Evidence that both genetic instability and selection contribute to the accumulation of chromosome alterations in cancer.
Carcinogenesis 26:923-930(2005)
PubMed=16397213; DOI=10.1158/0008-5472.CAN-05-2853
Elstrodt F., Hollestelle A., Nagel J.H.A., Gorin M., Wasielewski M., van den Ouweland A.M.W., Merajver S.D., Ethier S.P., Schutte M.
BRCA1 mutation analysis of 41 human breast cancer cell lines reveals three new deleterious mutants.
Cancer Res. 66:41-45(2006)
PubMed=16541312; DOI=10.1007/s10549-006-9186-z
Wasielewski M., Elstrodt F., Klijn J.G.M., Berns E.M.J.J., Schutte M.
Thirteen new p53 gene mutants identified among 41 human breast cancer cell lines.
Breast Cancer Res. Treat. 99:97-101(2006)
PubMed=19593635; DOI=10.1007/s10549-009-0460-8
Hollestelle A., Nagel J.H.A., Smid M., Lam S., Elstrodt F., Wasielewski M., Ng S.S., French P.J., Peeters J.K., Rozendaal M.J., Riaz M., Koopman D.G., ten Hagen T.L.M., de Leeuw B.H.C.G.M., Zwarthoff E.C., Teunisse A., van der Spek P.J., Klijn J.G.M., Dinjens W.N.M., Ethier S.P., Clevers H.C., Jochemsen A.G., den Bakker M.A., Foekens J.A., Martens J.W.M., Schutte M.
Distinct gene mutation profiles among luminal-type and basal-type breast cancer cell lines.
Breast Cancer Res. Treat. 121:53-64(2010)
PubMed=20070913; DOI=10.1186/1471-2407-10-15
Tsuji K., Kawauchi S., Saito S., Furuya T., Ikemoto K., Nakao M., Yamamoto S., Oka M., Hirano T., Sasaki K.
Breast cancer cell lines carry cell line-specific genomic alterations that are distinct from aberrations in breast cancer tissues: comparison of the CGH profiles between cancer cell lines and primary cancer tissues.
BMC Cancer 10:15.1-15.10(2010)
PubMed=22460905; DOI=10.1038/nature11003
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=23601657; DOI=10.1186/bcr3415
Riaz M., van Jaarsveld M.T.M., Hollestelle A., Prager-van der Smissen W.J.C., Heine A.A.J., Boersma A.W.M., Liu J.-J., Helmijr J.C.A., Ozturk B., Smid M., Wiemer E.A.C., Foekens J.A., Martens J.W.M.
miRNA expression profiling of 51 human breast cancer cell lines reveals subtype and driver mutation-specific miRNAs.
Breast Cancer Res. 15:R33.1-R33.17(2013)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=28940260; DOI=10.1002/ijc.31067
Korch C.T., Hall E.M., Dirks W.G., Ewing M., Faries M., Varella-Garcia M., Robinson S., Storts D.R., Turner J.A., Wang Y., Burnett E.C., Healy L.E., Kniss D.A., Neve R.M., Nims R.W., Reid Y.A., Robinson W.A., Capes-Davis A.
Authentication of M14 melanoma cell line proves misidentification of MDA-MB-435 breast cancer cell line.
Int. J. Cancer 142:561-572(2017)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. III, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
 技术资料
技术资料暂无技术资料 索取技术资料






![CCRF-CEM [CCRF CEM]人急性淋巴细胞白血病T淋巴细胞](https://img1.dxycdn.com/p/s14/2024/1226/202/8009356417684012781.jpg!wh200)
