相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 品系:
详见细胞说明资料
- 细胞类型:
详见细胞说明资料
- 肿瘤类型:
详见细胞说明资料
- 供应商:
上海冠导生物工程有限公司
- 库存:
≥100瓶
- 生长状态:
详见细胞说明资料
- 年限:
详见细胞说明资料
- 运输方式:
常温运输【复苏细胞】或干冰运输【冻存细胞】
- 器官来源:
详见细胞说明资料
- 是否是肿瘤细胞:
详见细胞说明资料
- 细胞形态:
详见细胞说明资料
- 免疫类型:
详见细胞说明资料
- 物种来源:
详见细胞说明资料
- 相关疾病:
详见细胞说明资料
- 组织来源:
详见细胞说明资料
- 英文名:
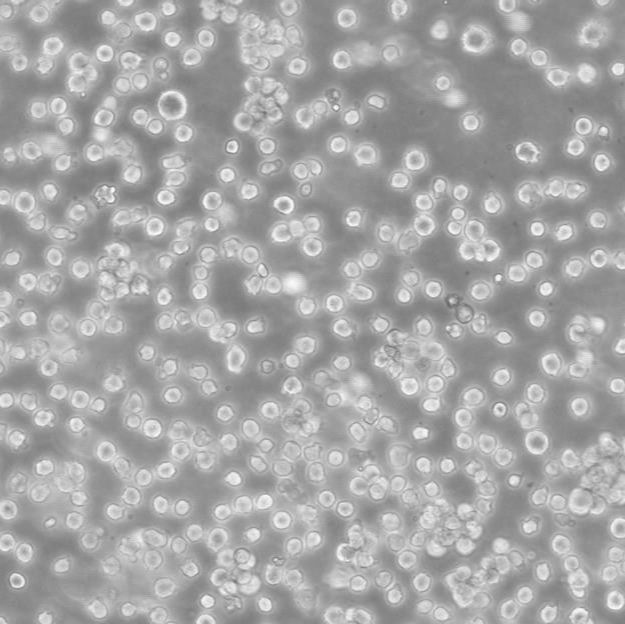
PSN1 Cells人胰腺癌细胞传代培养|STR图谱
- 规格:
1*10(6)Cellls/瓶
物种来源:PSN1 Cells人胰腺癌细胞传代培养|STR图谱来源于人源、鼠源等其它物种来源
生长特性:PSN1 Cells人胰腺癌细胞传代培养|STR图谱贴壁生长
形态特性:PSN1 Cells人胰腺癌细胞传代培养|STR图谱上皮细胞样
在实际应用中,不同细胞系需要定制化的冻存方案。例如,悬浮细胞比贴壁细胞更易冻存;原代细胞比永生化细胞系更脆弱;某些特殊细胞株可能需要特殊的保护剂组合。实验室间的细胞交换更凸显标准化冻存流程的重要性。国际细胞库提供的标准操作流程值得借鉴,包括使用无血清冻存培养基、严格控制降温速率等细节。统计表明,采用标准化冻存流程可使细胞复苏存活率提升至90%以上,而随意操作可能导致存活率不足50%。
传代培养是细胞培养技术中的关键环节,其核心目的是维持细胞的健康增殖并获取稳定的实验材料。当细胞在培养瓶中生长至80%-90%汇合度时,细胞间的接触抑制和营养竞争会导致生长速率下降,此时必须通过传代操作将细胞分散并重新接种。这一过程不仅解决了培养空间限制问题,还能通过细胞扩增建立可持续研究的细胞株系。然而值得注意的是,反复传代可能导致细胞表型改变,例如间充质干细胞多次传代后可能出现分化能力下降,因此需严格控制传代次数和操作规范。
换液周期:PSN1 Cells人胰腺癌细胞传代培养|STR图谱每周2-3次
产品包装:复苏发货:PSN1 Cells人胰腺癌细胞传代培养|STR图谱T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
背景信息:PSN1 Cells人胰腺癌细胞传代培养|STR图谱详见相关文献介绍
在培养细胞时,经常发现它们的状态不太理想,总是像一群顽皮的孩子般扎堆生长。用胰酶消化时,它们更是抱团取暖,形成一个个紧密的细胞团块,即使用移液管反复吹打也难以将它们分散成单个细胞。更令人困扰的是,它们的生长速度犹如雨后春笋,却极不均匀,传代第二天,某些区域就已拥挤得如同早高峰的地铁车厢。
当T25瓶复苏细胞收到货时,请观察好细胞状态后,将T25细胞瓶壁进行75%酒精消毒,将T25瓶置于37度培养箱放置2-4h,以便稳定细胞状态,当细胞密度达80%-90%,即可进行首次传代培养;干冰运输的细胞冻存管收到货后,需立即转入液氮保存或直接进行复苏(第三天换液并检查复苏细胞密度,以便进行下一步)。 能够在实验室条件下进行大量培养和繁殖。这种细胞系在分子生物学和生物技术研究中十分常用。
传代比例:1:2-1:4(PSN1 Cells人胰腺癌细胞传代培养|STR图谱首次传代建议1:2)
来源说明:PSN1 Cells人胰腺癌细胞传代培养|STR图谱主要来源ATCC、ECACC、DSMZ、RCB等细胞库
细胞传代准备工作:预热培养基和PBS:在开始传代前,将完全培养基和磷酸盐缓冲液(PBS),预热至37℃,以确保细胞在操作过程中不会受到温度冲击。准备无菌器材:包括无菌吸管、移液枪、离心管、培养皿、胰蛋白酶(或EDTA)溶液、完全培养基等。检查细胞状态:在显微镜下观察细胞的生长密度和形态,确保细胞处于对数生长期且状态良好。
营养供给是细胞的生命基石。就像新生儿需要母乳或配方奶粉提供全面营养,初代细胞或刚刚复苏的冻存细胞对营养环境有着近乎苛刻的要求。科研人员会为这些""细胞婴儿""精心配制含有10-15%胎牛血清的基础培养基,这相当于为细胞准备的高级""配方奶粉""。血清中富含的生长因子、激素和附着因子,就像母乳中的免疫球蛋白和益生菌,能为细胞提供最贴心的保护。而对于某些娇贵的干细胞或免疫细胞,还需要额外添加EGF、bFGF等""营养强化剂"",这些细胞因子相当于婴幼儿的DHA和维生素补充剂,能显著提高细胞的贴壁率和增殖活性。实验室数据表明,在复苏后24小时内添加适量血清的细胞,其存活率可比常规培养提高30%以上。
KU 812F Cells;背景说明:慢性粒细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:RPMI-8226S细胞、MRC-9细胞、HCC1171细胞
NCI-H78 Cells;背景说明:皮肤;T淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SK LU 1细胞、PK-136细胞、HEL-1细胞
TE 85.T Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BE(2)M17细胞、RPTEC/TERT1细胞、GA-10 (Clone 4)细胞
RT-BM-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成神经细胞;相关产品有:HUC-1细胞、HCC1806细胞、GM 2132细胞
NCI H2106 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:IPLB-SF-21细胞、Human podocyte细胞、TE671/RD细胞
COR-L88 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:FLC-7细胞、JCA1细胞、H-1385细胞
D-324 Med Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:多边形;相关产品有:NCI-SNU-182细胞、HCT-116细胞、H2066细胞
NIH:OVCAR-3-GFP Cells人卵巢癌腺癌细胞库专业复苏|带STR图谱
JHH-1 Cells人肝癌细胞库专业复苏|带STR图谱
SVCT Cells人乳腺上皮细胞库专业复苏|带STR图谱
HET-1A Cells人食管上皮细胞库专业复苏|带STR图谱
PSN1 Cells人胰腺癌细胞传代培养|STR图谱
MS1 Cells小鼠胰岛内皮细胞库专业复苏|带STR图谱
OCM-1A-Luc Cells人眼脉络黑色素瘤细胞库专业复苏|带STR图谱
COLO 205-Luc Cells人结肠癌细胞库专业复苏|带STR图谱
A2780 Cells人卵巢癌细胞库专业复苏|带STR图谱
WI-38 Cells人胚肺细胞库专业复苏|带STR图谱
HCC15 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:FRO81-2细胞、YD-38细胞、MOLT.4细胞
19C5 Cells(提供STR鉴定图谱)
Adeno-293 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:VMRC-LCD细胞、TSCC1细胞、8402细胞
GC-1 Cells;背景说明:精原细胞;SV40转化;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Normal Rat Kidney-52E细胞、Hs 695T细胞、GM05372E细胞
HCT GEO Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ISHI细胞、Rainbow Trout Embryo细胞、T_T_细胞
HT Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:WM35细胞、MIHA细胞、SNU1细胞
HCC38 BL Cells人淋巴母细胞库专业复苏|带STR图谱
SK-LU-1 Cells人低分化肺腺癌细胞库专业复苏|带STR图谱
PSN1 Cells人胰腺癌细胞传代培养|STR图谱
HMC3+RFP Cells人小胶质细胞库专业复苏|带STR图谱
HBL-100 Cells人整合SV40基因的乳腺上皮细胞库专业复苏|带STR图谱
U-937 Cells人组织细胞淋巴瘤细胞库专业复苏|带STR图谱
GCT Cells人巨细胞瘤细胞库专业复苏|带STR图谱
SW13 Cells人肾上腺皮质小细胞癌细胞库专业复苏|带STR图谱
PaTu 8988t Cells人胰腺癌细胞库专业复苏|带STR图谱
LL/2 (LLC1) Cells小鼠肺癌细胞库专业复苏|带STR图谱
SUM149-PT Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MHCC-LM3细胞、Vero E-6细胞、NOMO-1细胞
OAC-P4C Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hut292细胞、Karpas 422细胞、AG06814-M细胞
NCIH64 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Colo-201细胞、HL-1细胞、HOP62细胞
NCI-H508 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:293-H细胞、NCI660细胞、JVM3细胞
GM09931 Cells(提供STR鉴定图谱)
HAP1 BRD2 (-) 3 Cells(提供STR鉴定图谱)
HGC-27+luc Cells人胃癌细胞库专业复苏|带STR图谱
HaCat+luc Cells人永生化表皮细胞库专业复苏|带STR图谱
HEK293 Cells人胚肾细胞库专业复苏|带STR图谱
SK-N-BE(2)-M17 Cells人神经母细胞瘤细胞库专业复苏|带STR图谱
SW48 Cells人结肠腺癌细胞库专业复苏|带STR图谱
NCI-H157 Cells人非小细胞肺腺癌细胞库专业复苏|带STR图谱
BT483 Cells人乳腺导管癌细胞库专业复苏|带STR图谱
SW1710 Cells人膀胱癌细胞库专业复苏|带STR图谱
K562/Adr Cells人慢性髓系白血病细胞库专业复苏|带STR图谱
Case 3 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCIH2106细胞、BGC823细胞、H1666细胞
OCM1A Cells;背景说明:脉络膜黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:686LN-M4e细胞、C-Lu-65细胞、HUT 226细胞
MC3T3-E1 Subclone 14 Cells;背景说明:该细胞是从克隆的但是表型各异的MC3T3-E1细胞系中分离出一系列亚克隆。从含抗坏血酸培养基生长的成骨细胞中选择高或低成骨细胞分化、矿化的亚克隆。 MC3T3亚克隆4(ATCC CRL-2593)和MC3T3亚克隆14(ATCC CRL-2594)在抗坏血酸和3到4mM无机盐中生长表现出高水平的成骨细胞分化。 它们10天后形成一个矿化良好的细胞外基质(ECM)。MC3T3亚克隆24(ATCC CRL-2594)和MC3T3亚克隆30(ATCC CRL-2596)在抗坏血酸中生长表现出很差的成骨细胞分化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:成纤维细胞样;相关产品有:P3/X63/Ag8.653细胞、FLS细胞、DMS 53细胞
F442-A Cells;背景说明:脂肪前体细胞;雄性;Swiss albino;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MOLT-16细胞、DH 82细胞、UCLA-SO-M14细胞
H748 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长 ;形态特性:详见产品说明书;相关产品有:MJ细胞、H-2009细胞、CAL 27细胞
MDAMB435 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIH647细胞、PTK1细胞、UACC 812细胞
Caov-3 Cells人卵巢腺癌细胞库专业复苏|带STR图谱
R 1610 Cells仓鼠肺细胞库专业复苏|带STR图谱
H9/HTLV-IIIB Cells人T淋巴瘤白血病细胞库专业复苏|带STR图谱
T/G HA-VSMC Cells人主动脉血管平滑肌细胞库专业复苏|带STR图谱
PSN1 Cells人胰腺癌细胞传代培养|STR图谱
293GP Cells人胚肾细胞库专业复苏|带STR图谱
TE-10 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NGEC细胞、SK-N-BE(2)-C细胞、SUPB-15细胞
293-GP Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SHG 44细胞、SKES1细胞、Panc2.03细胞
HG03388 Cells(提供STR鉴定图谱)
ID00006 Cells(提供STR鉴定图谱)
Hs 815.Pl Cells人胎盘细胞库专业复苏|带STR图谱
AU565 Cells人乳腺癌细胞库专业复苏|带STR图谱
HUVEC-GFP Cells人脐静脉内皮细胞库专业复苏|带STR图谱
Reh Cells人急性淋巴细胞白血病细胞库专业复苏|带STR图谱
P560 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HuT-78细胞、H889细胞、OVCAR-10细胞
BT 549 Cells;背景说明:该细胞1978年由W.G.Coutinho和E.Y.Lasfargues建系,源自一位72岁患有乳腺导管癌的白人女性,来源组织包括乳头及浸润导管。该细胞形态包括上皮样细胞及多核巨细胞,可分泌一种粘性物质。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:DU4475细胞、HS-729细胞、Mouse podocyte细胞
MM1S Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:混合生长;形态特性:淋巴母细胞样;相关产品有:L-1210细胞、FAT细胞、B5537SKIN细胞
HAP1 ZMYM4 (-) 2 Cells(提供STR鉴定图谱)
MeT 5A Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:SW1783细胞、IPI-2I细胞、Panc 02.03细胞
CAL-78 Cells;背景说明:软骨肉瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1435细胞、COLO 680N细胞、Ontario Cancer Institute-Acute Myeloid Leukemia-5细胞
HTori-3.1 Cells;背景说明:甲状腺;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BEL-7405细胞、TEC细胞、P3HR-1细胞
SK_HEP1 Cells;背景说明:SK-HEP-1细胞系已被鉴定为内皮来源。该细胞系为异倍体女性人(XX),染色体在亚三倍体范围内。在裸鼠中,它能形成与肝癌相一致的大细胞癌;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:WIL2 S细胞、Panc 03.27细胞、Hk-2细胞
MKN45 Cells人胃癌细胞库专业复苏|带STR图谱
L Wnt-3A Cells小鼠皮下结缔组织细胞库专业复苏|带STR图谱
COLO 201 Cells人结直肠腺癌细胞库专业复苏|带STR图谱
PSN1 Cells人胰腺癌细胞传代培养|STR图谱
Reh Cells人急性淋巴细胞白血病细胞库专业复苏|带STR图谱
hFOB 1.19 Cells人成骨细胞库专业复苏|带STR图谱
AT-3-GFP Cells小鼠乳腺癌细胞库专业复苏|带STR图谱
LNCaP-Pro5 Cells(提供STR鉴定图谱)
NCI-H433 Cells(提供STR鉴定图谱)
PathHunter U2OS C3AR1 beta-arrestin Cells(提供STR鉴定图谱)
Ubigene A-549 CD276 KO Cells(提供STR鉴定图谱)
UOK121LN Cells(提供STR鉴定图谱)
" "PubMed=11787853; DOI=10.1007/s004280100474
Moore P.S., Sipos B., Orlandini S., Sorio C., Real F.X., Lemoine N.R., Gress T.M., Bassi C., Kloppel G., Kalthoff H., Ungefroren H., Lohr J.-M., Scarpa A.
Genetic profile of 22 pancreatic carcinoma cell lines. Analysis of K-ras, p53, p16 and DPC4/Smad4.
Virchows Arch. 439:798-802(2001)
PubMed=18380791; DOI=10.1111/j.1349-7006.2008.00779.x; PMCID=PMC11158928
Suzuki A., Shibata T., Shimada Y., Murakami Y., Horii A., Shiratori K., Hirohashi S., Inazawa J., Imoto I.
Identification of SMURF1 as a possible target for 7q21.3-22.1 amplification detected in a pancreatic cancer cell line by in-house array-based comparative genomic hybridization.
Cancer Sci. 99:986-994(2008)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25167228; DOI=10.1038/bjc.2014.475; PMCID=PMC4453732
Hamidi H., Lu M., Chau K., Anderson L., Fejzo M.S., Ginther C., Linnartz R., Zubel A., Slamon D.J., Finn R.S.
KRAS mutational subtype and copy number predict in vitro response of human pancreatic cancer cell lines to MEK inhibition.
Br. J. Cancer 111:1788-1801(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26216984; DOI=10.1073/pnas.1501605112; PMCID=PMC4538616
Daemen A., Peterson D., Sahu N., McCord R., Du X.-N., Liu B., Kowanetz K., Hong R., Moffat J., Gao M., Boudreau A., Mroue R., Corson L., O'Brien T., Qing J., Sampath D., Merchant M., Yauch R.L., Manning G., Settleman J., Hatzivassiliou G., Evangelista M.
Metabolite profiling stratifies pancreatic ductal adenocarcinomas into subtypes with distinct sensitivities to metabolic inhibitors.
Proc. Natl. Acad. Sci. U.S.A. 112:E4410-E4417(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验Moore P.S., Sipos B., Orlandini S., Sorio C., Real F.X., Lemoine N.R., Gress T.M., Bassi C., Kloppel G., Kalthoff H., Ungefroren H., Lohr J.-M., Scarpa A.
Genetic profile of 22 pancreatic carcinoma cell lines. Analysis of K-ras, p53, p16 and DPC4/Smad4.
Virchows Arch. 439:798-802(2001)
PubMed=18380791; DOI=10.1111/j.1349-7006.2008.00779.x; PMCID=PMC11158928
Suzuki A., Shibata T., Shimada Y., Murakami Y., Horii A., Shiratori K., Hirohashi S., Inazawa J., Imoto I.
Identification of SMURF1 as a possible target for 7q21.3-22.1 amplification detected in a pancreatic cancer cell line by in-house array-based comparative genomic hybridization.
Cancer Sci. 99:986-994(2008)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25167228; DOI=10.1038/bjc.2014.475; PMCID=PMC4453732
Hamidi H., Lu M., Chau K., Anderson L., Fejzo M.S., Ginther C., Linnartz R., Zubel A., Slamon D.J., Finn R.S.
KRAS mutational subtype and copy number predict in vitro response of human pancreatic cancer cell lines to MEK inhibition.
Br. J. Cancer 111:1788-1801(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26216984; DOI=10.1073/pnas.1501605112; PMCID=PMC4538616
Daemen A., Peterson D., Sahu N., McCord R., Du X.-N., Liu B., Kowanetz K., Hong R., Moffat J., Gao M., Boudreau A., Mroue R., Corson L., O'Brien T., Qing J., Sampath D., Merchant M., Yauch R.L., Manning G., Settleman J., Hatzivassiliou G., Evangelista M.
Metabolite profiling stratifies pancreatic ductal adenocarcinomas into subtypes with distinct sensitivities to metabolic inhibitors.
Proc. Natl. Acad. Sci. U.S.A. 112:E4410-E4417(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"
分型已被 ICLAC、ATCC 等权威机构作为金标准应用于细胞鉴定,目前越来越多的杂志要求在投稿时提供细胞 STR 分型数据。最佳鉴定时间1、发表文章或申请课题经费前;2、使用细胞进行临床治疗(试验)前;3、准备冻存保种或已冻存多年的细胞;4、一个涉及到细胞试验项目开始 / 结束时;5、新得到的细胞或实验室培养 5 代以上的细胞;6、细胞系表现不稳定或结果与预期差别较大;7、异体细胞移植后嵌合情况检查。科佰优势STR 鉴定的实验方法已经很成熟了,常规都是在 ABI 3730xl 上完成检测,由机器读取峰图,导出
刚刚在中国科学院昆明细胞库看到他们今年最新公布的错误鉴定和交叉污染的细胞,共 60 个细胞系,STR 检测结果和备检细胞系的名称相差甚远,大家赶紧去看看自己的细胞是否在列吧。 很多生物医药的研究都采用培养细胞来进行, 这些细胞可能是从细胞库 (如美国 ATCC,American Type Culture Collection) 得来,也可能是受赠于其它研究人员,尤其在中国,目前细胞的传播太复杂,无序,很多都不是通过正规途径获得,如友情赠送等。 据统计,约有 30% 细胞系被交叉
),(5)PACSU(对照平板)。复制平板经培养以后,每个重组子的基因型根据它们在每种培养基上是否生长而得到鉴定。4、遗传图谱的制作本研究挑选了具有str(链霉素抗性)性状和his+(不需要组氨酸)性状的重组子172个,然后影印到五种“诊断培养基”上。172个重组子在五种“诊断培养基”上得到生长情况如下:在(1)ACSU(不加P)上生长了40个重组子;在(2)PCSU(不加A)上生长了46个;在(3)PASU(不加C)上生长了54个重组子;但在(4)PACS(不加U)上却未发现有生长的重组;,在(5)PACSU
 技术资料
技术资料暂无技术资料 索取技术资料