相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 品系:
详见细胞说明资料
- 细胞类型:
详见细胞说明资料
- 肿瘤类型:
详见细胞说明资料
- 供应商:
上海冠导生物工程有限公司
- 库存:
≥100瓶
- 生长状态:
详见细胞说明资料
- 年限:
详见细胞说明资料
- 运输方式:
常温运输【复苏细胞】或干冰运输【冻存细胞】
- 器官来源:
详见细胞说明资料
- 是否是肿瘤细胞:
详见细胞说明资料
- 细胞形态:
详见细胞说明资料
- 免疫类型:
详见细胞说明资料
- 物种来源:
详见细胞说明资料
- 相关疾病:
详见细胞说明资料
- 组织来源:
详见细胞说明资料
- 英文名:
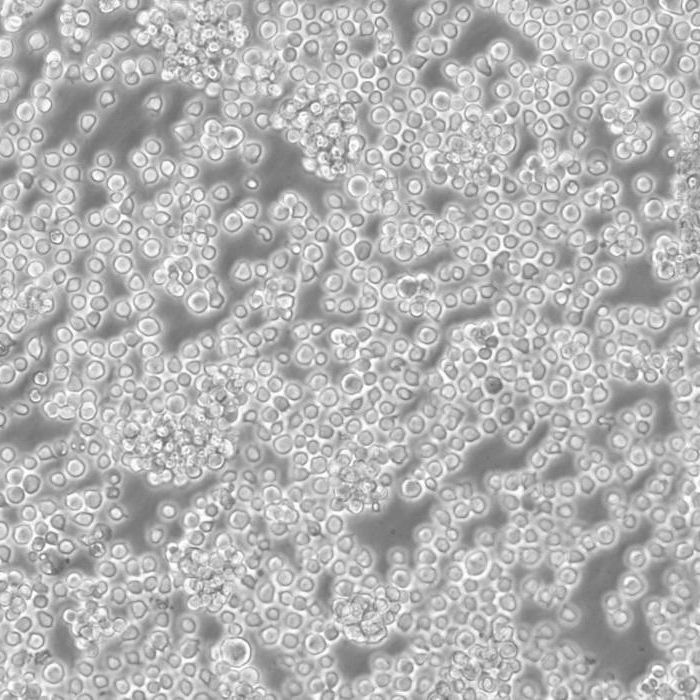
SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱
- 规格:
1*10(6)Cellls/瓶
物种来源:SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱来源于人源、鼠源等其它物种来源
生长特性:SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱贴壁生长
形态特性:SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱上皮细胞样
根据国际细胞培养标准,未开封血清必须贮存于-20℃至-70℃深低温环境。实验证实,在-70℃超低温条件下,血清中的生长因子和蛋白质可保持5年活性稳定;而-20℃常规冷冻也能维持3年有效期限。值得注意的是,若因特殊情况需临时存放于4℃冰箱,必须严格控制在1个月内使用完毕,否则血清中的不稳定成分(如维生素、氨基酸等)会以每天0.5%的速率降解。细胞库的对比数据显示,4℃存放超过30天的血清会使细胞增殖速率降低17%-23%。
细胞培养是生物学和医学研究中不可或缺的技术手段,而温度作为细胞培养环境中的核心参数之一,直接影响细胞的生长、代谢和功能。维持恒定的适宜温度是确保细胞健康生长的关键因素。不同种类的细胞对温度的要求存在差异,而人体细胞的标准培养温度通常设定为36.5℃±0.5℃。这一温度范围模拟了人体内环境的稳态,能够地支持细胞的正常生理活动。
换液周期:SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱每周2-3次
产品包装:复苏发货:SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
背景信息:SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱详见相关文献介绍
细胞传代后需定期观察其生长状态。通常在2-4天内细胞可长成单层,具体时间取决于细胞接种浓度和环境条件。观察时应注意以下几点:传代后24小时内,细胞应贴壁并开始伸展。若贴壁不良,可能是消化过度或培养液配制不当所致。健康细胞应保持原有形态,若出现变形、颗粒增多或空泡化等异常现象,需及时排查原因。定期检查培养液是否浑浊或出现异常漂浮物,以排除细菌或真菌污染的可能。此外,实验人员需详细填写传代记录,包括细胞类型、传代日期、传代比例、培养液配方、操作人员等信息,以便后续实验追溯和质量控制。
细胞代谢随温度降低而逐渐减慢。当温度降至冰点以下时,细胞内的游离水会形成冰晶,导致细胞膜和细胞器结构的机械损伤,最终引发细胞死亡。然而,通过添加冷冻保护剂(如二甲亚砜或甘油),可以显著提高细胞在深低温环境下的存活率。这些保护剂能够降低冰晶的形成概率,并减少低温对细胞结构的破坏。
传代比例:1:2-1:4(SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱首次传代建议1:2)
来源说明:SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱主要来源ATCC、ECACC、DSMZ、RCB等细胞库
细胞传代如同为生命搭建一座崭新的舞台,是细胞培养艺术中的关键一笔。这项精妙的操作犹如园丁移栽幼苗,将蓬勃生长的细胞从拥挤的旧培养瓶温柔地迁入宽敞的新居,让这些微小的生命之花得以继续绽放。在显微镜下,细胞们如同训练有素的舞者,遵循着精密的生命韵律,在新的培养环境中舒展身姿,延续着它们与生俱来的生命密码。这不仅是一次简单的空间转移,更是一场精心编排的生命接力,确保每一代细胞都能完美传承其独特的生物学禀赋。
DSMZ菌株保藏中心成立于1969年,是德国的国家菌种保藏中心。该中心一直致力于细菌、真菌、质粒、抗菌素、人体和动物细胞、植物病毒等的分类、鉴定和保藏工作。DSMZ菌种保藏中心是欧洲规模最大的生物资源中心,保藏有动物细胞500多株。日本Riken BRC(Riken生物资源保藏中心)是全球三大典型培养物收集中心之一。Riken保藏中心提供了很多细胞系。在世界范围内,这些细胞系,都在医学、科学和兽医中具有重要意义。Riken生物资源中心支持了各种学术、健康、食品和兽医机构的研究工作,并在世界各地不同组织的微生物实验室和研究机构中使用。
H-1435 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,每周换液2-3次;生长特性:贴壁生长,松散;形态特性:详见产品说明书;相关产品有:YD15细胞、H-2135细胞、OCI/AML2细胞
Caov-4 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IR983F细胞、Cor L51细胞、SW13细胞
Ca Ski Cells;背景说明:这株细胞是从小肠肠系膜转移灶的细胞中建立的。 据报道,它含有完整的HPV-16(每个细胞大约600个拷贝)和HPV-18相关序列。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Tb 1 Lu (NBL-12)细胞、University of Michigan-Urothelial Carcinoma-14细胞、D-407细胞
CA-OV-3 Cells;背景说明:该细胞1976年建系,源自一位54岁白人女性的卵巢腺癌组织。;传代方法:1:3传代,2—3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HIT T15细胞、VK2 (E6/E7)细胞、L6565细胞
NCIH345 Cells;背景说明:小细胞肺癌;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:COLO 738细胞、MT4细胞、Jurkat clone A3细胞
A-72 Cells;背景说明:癌细胞;金毛猎犬;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MDA-157细胞、aTC1 Clone 6细胞、NU-GC-4细胞
MOLT.4 Cells;背景说明:MOLT-4与MOLT-3来源于一名19岁的男性急性淋巴细胞性白血病的复发患者,该患者前期接受过多种药物联合化疗。MOLT-4细胞系为T淋巴细胞起源,p53基因的第248位密码子有一个G→A突变,不表达p53,不表达免疫球蛋白或EB病毒;可产生高水平的末端脱氧核糖转移酶;表达CD1(49%),CD2(35%),CD3A(26%)B(33%)C(34%),CD4(55%),CD5(72%),CD6(22%),CD7(77%)。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;圆形;相关产品有:H295细胞、A2780S细胞、DCS细胞
DMS 114 Cells人肺癌细胞库专业复苏|带STR图谱
MP41 Cells人眼葡萄膜黑色素瘤细胞库专业复苏|带STR图谱
RS4:11 Cells人急性淋巴白血病细胞库专业复苏|带STR图谱
LS180 Cells人结肠癌细胞库专业复苏|带STR图谱
SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱
Calu-1 Cells人肺癌细胞库专业复苏|带STR图谱
Neuro-2a-Luc Cells小鼠脑神经瘤细胞库专业复苏|带STR图谱
BT-20 Cells人乳腺癌细胞库专业复苏|带STR图谱
NCI-N87-luc Cells人胃癌细胞库专业复苏|带STR图谱
KYSE-140 Cells人食管鳞癌细胞库专业复苏|带STR图谱
NCIH378 Cells;背景说明:小细胞肺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:COLO320-DM细胞、COS-7细胞、EC9706细胞
155-9F6 Cells(提供STR鉴定图谱)
MADB-106 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MNNG/HOS Clone F-5细胞、MUTZ-1细胞、Caov-3细胞
Panc02.03 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H2198细胞、U118MG细胞、UWB1-289细胞
Calu-1 Cells;背景说明:详见相关文献介绍;传代方法:消化10分钟。1:2。3-4天长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:KYSE-140细胞、BCAP37细胞、NCI-BL2141细胞
OACP4C Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:B-cell Acute Lymphoblastic Leukemia-1细胞、EOC 20细胞、NB-4细胞
HeLa 229 Cells人宫颈癌细胞库专业复苏|带STR图谱
Panc 0813-LUC Cells人胰腺癌细胞库专业复苏|带STR图谱
SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱
MEG-01 Cells人成巨核细胞白血病细胞库专业复苏|带STR图谱
MHCC-97H Cells人高转移性肝癌细胞库专业复苏|带STR图谱
SKM-1 Cells人急性髓系白血病细胞库专业复苏|带STR图谱
HOS-Luc Cells人骨肉瘤细胞库专业复苏|带STR图谱
HEK293-EBNA Cells人胚肾细胞库专业复苏|带STR图谱
Malme-3M Cells人皮肤恶性黑色素瘤细胞库专业复苏|带STR图谱
GH3 Cells大鼠垂体瘤细胞库专业复苏|带STR图谱
SK-N-FI Cells;背景说明:详见相关文献介绍;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Tb 1 Lu细胞、P3/X63/Ag8细胞、MCF 10A细胞
PK136 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NFS60细胞、Vero-76细胞、Institute for Medical Research-90细胞
OCI Ly10 Cells;背景说明:弥漫大B细胞淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SCL-1细胞、HD-LM-2细胞、A2780-CP细胞
Be Wo Cells;背景说明:取自人绒癌脑转移组织,在仓鼠颊囊移植传代8年。利用移植瘤组织进行体外培养,建立细胞系。利用不同传代方法建立了不同亚系,JEG-3是其衍生克隆。该细胞可以产生雌激素、孕激素、雌酮、雌二醇、雌三醇、hCG、胎盘催乳素、角蛋白。;传代方法:1:3传代,3-4天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MDAMB330细胞、LNCaP C4-2B细胞、IMR-32细胞
HAP1 AIMP1 (-) 1 Cells(提供STR鉴定图谱)
HAP1 SIRT3 (-) 1 Cells(提供STR鉴定图谱)
Sp2/mIL-6 Cells小鼠骨髓瘤B淋巴细胞库专业复苏|带STR图谱
KYSE-150 Cells人食管鳞癌细胞库专业复苏|带STR图谱
HCE-T Cells人角膜上皮细胞库专业复苏|带STR图谱
OCI-AML-3 Cells人急性髓系白血病细胞库专业复苏|带STR图谱
Hs 578Bst Cells人正常乳腺细胞库专业复苏|带STR图谱
HCC44 Cells人非小细胞肺癌细胞库专业复苏|带STR图谱
HCC-44 Cells人非小细胞肺癌细胞库专业复苏|带STR图谱
ACHN Cells人肾细胞腺癌细胞库专业复苏|带STR图谱
HCC1937 Cells人乳腺癌细胞库专业复苏|带STR图谱
MOLT 16 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:R.K.13细胞、HANK1细胞、NS20-Y细胞
CNE1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:OE33细胞、KMS-11细胞、HEK-293细胞
U87MG Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:OE-19细胞、EFM-192C细胞、BHK 21细胞
CII Cells;背景说明:慢性淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:MLFC细胞、bEnd3细胞、HEL92.1.7细胞
B6Tert-1 Cells;背景说明:滋养层;HGNC-TRET永生;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CEM-0细胞、EL4细胞、SNT8细胞
W256 Cells;背景说明:乳腺癌;雌性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HUC细胞、SNU-739细胞、SKMES1细胞
SNU878 Cells人肝癌细胞库专业复苏|带STR图谱
EBTr (NBL-4) Cells牛胚气管细胞库专业复苏|带STR图谱
JHH-1 Cells人肝癌细胞库专业复苏|带STR图谱
TU212 Cells人喉癌细胞库专业复苏|带STR图谱
SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱
Pfeiffer Cells人弥漫大B细胞淋巴瘤细胞库专业复苏|带STR图谱
CEM C7 Cells;背景说明:急性T淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NPC-TW 039细胞、A2008细胞、BeWo细胞
HL-60 Cells;背景说明:该细胞由CollinsSJ从一位患有急性早幼粒细胞性白血病的36岁白人女性的外周血中分离建立;可自发分化,或在盐、次黄嘌呤、佛波醇肉豆蔻酸(PMA,TPA)、DMSO(1%to1.5%)、D和视黄酸的刺激下发生分化;PMA刺激后可分泌TNF-α。该细胞具有吞噬活性和趋化反应,癌基因myc阳性,表达补体受体和FcR。;传代方法:维持细胞浓度在1×105-1×106/ml,每2-3天换液1次。;生长特性:悬浮生长;形态特性:髓母细胞样;相关产品有:Panc-813细胞、CX-1细胞、KNS62细胞
HyCyte NCI-H460-Luc Cells(提供STR鉴定图谱)
LCL-PI 69 Cells(提供STR鉴定图谱)
REC-1 Cells人淋巴瘤细胞库专业复苏|带STR图谱
COLO 16 Cells人皮肤鳞状细胞癌细胞库专业复苏|带STR图谱
MKN45 Cells人胃癌细胞库专业复苏|带STR图谱
NCI-H661 Cells人大细胞肺癌细胞库专业复苏|带STR图谱
Malme-3M Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:NCI-H920细胞、HEK-EBNA细胞、Hs 343.T细胞
TE9 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:GM03569细胞、NCIH838细胞、RPMI-6666细胞
L 428 Cells;背景说明:霍奇金淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SNU475细胞、HCC-1833细胞、QGP1细胞
HG01050 Cells(提供STR鉴定图谱)
IA-LM Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CCD-19Lu细胞、H2009细胞、H-220细胞
UO31 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MHH-CALL-2细胞、MV-4:11细胞、293 HEK细胞
NCI-H1688 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MOLT3细胞、P 3 HR 1细胞、CCD 1112SK细胞
Sp2/O Cells;背景说明:该细胞是由绵羊红细胞免疫的BALB/c小鼠脾细胞和P3X63Ag8骨髓瘤细胞融合得到的。该细胞不分泌免疫球蛋白,对20μg/ml的8-氮鸟嘌呤有抗性,对HAT比较敏感;该细胞可以作为细胞融合时的B细胞组分用于制备杂交瘤;鼠痘病毒阴性。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;圆形;相关产品有:X63-AG 8.653细胞、OUMS-23细胞、SCC4细胞
RAW264.7-GFP Cells小鼠单核巨噬细胞白血病细胞库专业复苏|带STR图谱
UMR-106 Cells大鼠骨肉瘤细胞库专业复苏|带STR图谱
NCI-H929 Cells人骨髓瘤细胞库专业复苏|带STR图谱
SUIT-2 Cells人胰腺癌细胞传代培养|STR图谱
CCRF-CEM Cells人急性淋巴细胞白血病细胞库专业复苏|带STR图谱
ICR Cells小鼠胚胎成纤维细胞库专业复苏|带STR图谱
LS1034 Cells人结肠腺癌细胞库专业复苏|带STR图谱
NAL1A clone B5SF11 Cells(提供STR鉴定图谱)
p-hiPS02 Cells(提供STR鉴定图谱)
RTiv3 Cells(提供STR鉴定图谱)
Ubigene HCT 116 MKNK1 KO Cells(提供STR鉴定图谱)
WAe001-A-1J Cells(提供STR鉴定图谱)
" "PubMed=11169959; DOI=10.1002/1097-0215(200002)9999:9999<::AID-IJC1049>3.0.CO;2-C
Sirivatanauksorn V., Sirivatanauksorn Y., Gorman P.A., Davidson J.M., Sheer D., Moore P.S., Scarpa A., Edwards P.A.W., Lemoine N.R.
Non-random chromosomal rearrangements in pancreatic cancer cell lines identified by spectral karyotyping.
Int. J. Cancer 91:350-358(2001)
PubMed=11787853; DOI=10.1007/s004280100474
Moore P.S., Sipos B., Orlandini S., Sorio C., Real F.X., Lemoine N.R., Gress T.M., Bassi C., Kloppel G., Kalthoff H., Ungefroren H., Lohr J.-M., Scarpa A.
Genetic profile of 22 pancreatic carcinoma cell lines. Analysis of K-ras, p53, p16 and DPC4/Smad4.
Virchows Arch. 439:798-802(2001)
PubMed=11854916; DOI=10.3748/wjg.v7.i6.855; PMCID=PMC4695609
Liu B., Staren E., Iwamura T., Appert H., Howard J.
Taxotere resistance in SUIT. Taxotere resistance in pancreatic carcinoma cell line SUIT 2 and its sublines.
World J. Gastroenterol. 7:855-859(2001)
PubMed=12800145; DOI=10.1002/gcc.10218
Adelaide J., Huang H.-E., Murati A., Alsop A.E., Orsetti B., Mozziconacci M.-J., Popovici C., Ginestier C., Letessier A., Basset C., Courtay-Cahen C., Jacquemier J., Theillet C., Birnbaum D., Edwards P.A.W., Chaffanet M.
A recurrent chromosome translocation breakpoint in breast and pancreatic cancer cell lines targets the neuregulin/NRG1 gene.
Genes Chromosomes Cancer 37:333-345(2003)
PubMed=19077451; DOI=10.1159/000178871
Harada T., Chelala C., Crnogorac-Jurcevic T., Lemoine N.R.
Genome-wide analysis of pancreatic cancer using microarray-based techniques.
Pancreatology 9:13-24(2009)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26216984; DOI=10.1073/pnas.1501605112; PMCID=PMC4538616
Daemen A., Peterson D., Sahu N., McCord R., Du X.-N., Liu B., Kowanetz K., Hong R., Moffat J., Gao M., Boudreau A., Mroue R., Corson L., O'Brien T., Qing J., Sampath D., Merchant M., Yauch R.L., Manning G., Settleman J., Hatzivassiliou G., Evangelista M.
Metabolite profiling stratifies pancreatic ductal adenocarcinomas into subtypes with distinct sensitivities to metabolic inhibitors.
Proc. Natl. Acad. Sci. U.S.A. 112:E4410-E4417(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验Sirivatanauksorn V., Sirivatanauksorn Y., Gorman P.A., Davidson J.M., Sheer D., Moore P.S., Scarpa A., Edwards P.A.W., Lemoine N.R.
Non-random chromosomal rearrangements in pancreatic cancer cell lines identified by spectral karyotyping.
Int. J. Cancer 91:350-358(2001)
PubMed=11787853; DOI=10.1007/s004280100474
Moore P.S., Sipos B., Orlandini S., Sorio C., Real F.X., Lemoine N.R., Gress T.M., Bassi C., Kloppel G., Kalthoff H., Ungefroren H., Lohr J.-M., Scarpa A.
Genetic profile of 22 pancreatic carcinoma cell lines. Analysis of K-ras, p53, p16 and DPC4/Smad4.
Virchows Arch. 439:798-802(2001)
PubMed=11854916; DOI=10.3748/wjg.v7.i6.855; PMCID=PMC4695609
Liu B., Staren E., Iwamura T., Appert H., Howard J.
Taxotere resistance in SUIT. Taxotere resistance in pancreatic carcinoma cell line SUIT 2 and its sublines.
World J. Gastroenterol. 7:855-859(2001)
PubMed=12800145; DOI=10.1002/gcc.10218
Adelaide J., Huang H.-E., Murati A., Alsop A.E., Orsetti B., Mozziconacci M.-J., Popovici C., Ginestier C., Letessier A., Basset C., Courtay-Cahen C., Jacquemier J., Theillet C., Birnbaum D., Edwards P.A.W., Chaffanet M.
A recurrent chromosome translocation breakpoint in breast and pancreatic cancer cell lines targets the neuregulin/NRG1 gene.
Genes Chromosomes Cancer 37:333-345(2003)
PubMed=19077451; DOI=10.1159/000178871
Harada T., Chelala C., Crnogorac-Jurcevic T., Lemoine N.R.
Genome-wide analysis of pancreatic cancer using microarray-based techniques.
Pancreatology 9:13-24(2009)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26216984; DOI=10.1073/pnas.1501605112; PMCID=PMC4538616
Daemen A., Peterson D., Sahu N., McCord R., Du X.-N., Liu B., Kowanetz K., Hong R., Moffat J., Gao M., Boudreau A., Mroue R., Corson L., O'Brien T., Qing J., Sampath D., Merchant M., Yauch R.L., Manning G., Settleman J., Hatzivassiliou G., Evangelista M.
Metabolite profiling stratifies pancreatic ductal adenocarcinomas into subtypes with distinct sensitivities to metabolic inhibitors.
Proc. Natl. Acad. Sci. U.S.A. 112:E4410-E4417(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"
分型已被 ICLAC、ATCC 等权威机构作为金标准应用于细胞鉴定,目前越来越多的杂志要求在投稿时提供细胞 STR 分型数据。最佳鉴定时间1、发表文章或申请课题经费前;2、使用细胞进行临床治疗(试验)前;3、准备冻存保种或已冻存多年的细胞;4、一个涉及到细胞试验项目开始 / 结束时;5、新得到的细胞或实验室培养 5 代以上的细胞;6、细胞系表现不稳定或结果与预期差别较大;7、异体细胞移植后嵌合情况检查。科佰优势STR 鉴定的实验方法已经很成熟了,常规都是在 ABI 3730xl 上完成检测,由机器读取峰图,导出
刚刚在中国科学院昆明细胞库看到他们今年最新公布的错误鉴定和交叉污染的细胞,共 60 个细胞系,STR 检测结果和备检细胞系的名称相差甚远,大家赶紧去看看自己的细胞是否在列吧。 很多生物医药的研究都采用培养细胞来进行, 这些细胞可能是从细胞库 (如美国 ATCC,American Type Culture Collection) 得来,也可能是受赠于其它研究人员,尤其在中国,目前细胞的传播太复杂,无序,很多都不是通过正规途径获得,如友情赠送等。 据统计,约有 30% 细胞系被交叉
),(5)PACSU(对照平板)。复制平板经培养以后,每个重组子的基因型根据它们在每种培养基上是否生长而得到鉴定。4、遗传图谱的制作本研究挑选了具有str(链霉素抗性)性状和his+(不需要组氨酸)性状的重组子172个,然后影印到五种“诊断培养基”上。172个重组子在五种“诊断培养基”上得到生长情况如下:在(1)ACSU(不加P)上生长了40个重组子;在(2)PCSU(不加A)上生长了46个;在(3)PASU(不加C)上生长了54个重组子;但在(4)PACS(不加U)上却未发现有生长的重组;,在(5)PACSU
 技术资料
技术资料暂无技术资料 索取技术资料






![NCI-H446[H446]人小细胞肺癌传代细胞长期复苏|送STR图谱](https://img1.dxycdn.com/p/s14/2025/0213/765/8442957467425299881.jpg!wh200)
