相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 询价记录
- 文献和实验
- 技术资料
- 库存:
充足
- 英文名:
Protein A/G Magnetic Beads
- 供应商:
南京诺唯赞生物科技股份有限公司
- 保存条件:
2 ~ 8℃保存,根据不同目的地调整运输方式。
- 规格:
1 ml/5 ml
| 规格: | 1 ml | 产品价格: | ¥799.0 |
|---|---|---|---|
| 规格: | 5 ml | 产品价格: | ¥2980.0 |
Protein A/G Magnetic Beads
IP/CoIP/ChIP适用,特异性好背景低!

蛋白纯化、蛋白互作

- 低背景干扰
-
ChIP信噪 比好
- 结合效率高

Protein A/G Magnetic Beads是由聚合物磁性微球与高纯度的重组蛋白A/G共价结合而成,磁珠粒径为1 μm,重组蛋白A/G包含蛋白A的5个Fc结合结构域和蛋白G的2个Fc结合结构域,结合能力较蛋白A或蛋白G更强。本产品特异性表面区域更大,抗体结合位点更多,可以大大缩短抗体吸附时间,减少非特异性结合。

1. 低背景干扰
使用Protein A/G磁珠Vazyme #PB101,以1 mg 293T细胞裂解蛋白为样本,对 phospho-Akt蛋白进行免疫沉淀实验,经1×SDS-PAGE Sample Loading Buffer高温变性洗脱后,搭配抗轻链二抗,通过Western Blot检测phospho-Akt蛋白富集水 平。结 果显示,PB101对蛋白的非特异性吸附低,且无Proetin A/G蛋白掉落干扰,背景更干净。
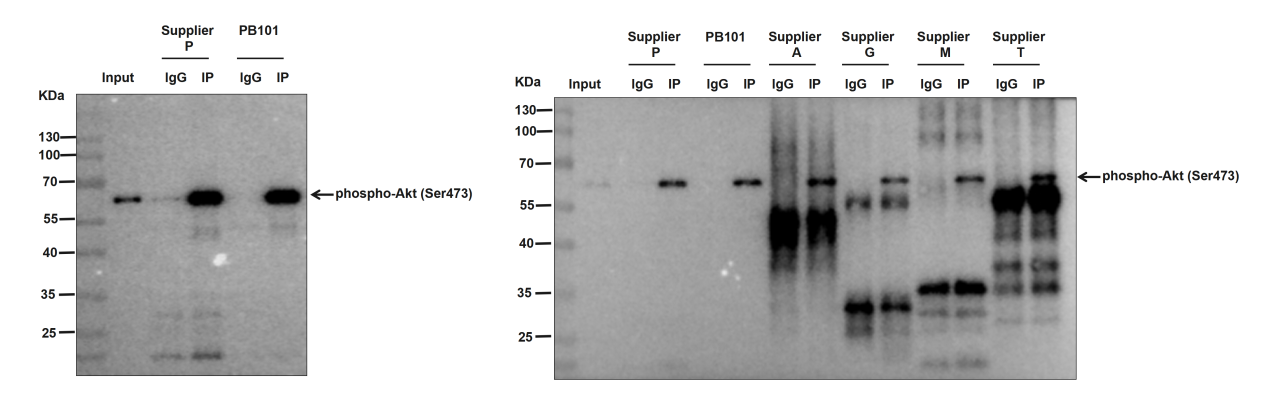
图1.phospho-Akt蛋白的Western Blot检测结果图
2. ChIP信噪比好
使用Protein A/G磁珠Vazyme #PB101,以293T细胞为样本,选择H3K4me3为靶点进行ChIP实验。结果显示,PB101可兼容ChIP-seq实验,信噪比好。
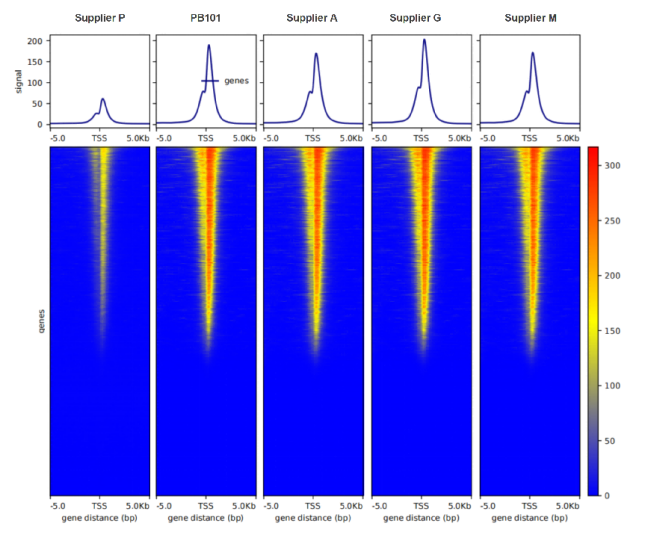
图2.以H3K4me3为靶点的不同Protein A/G磁珠的TSS富集图
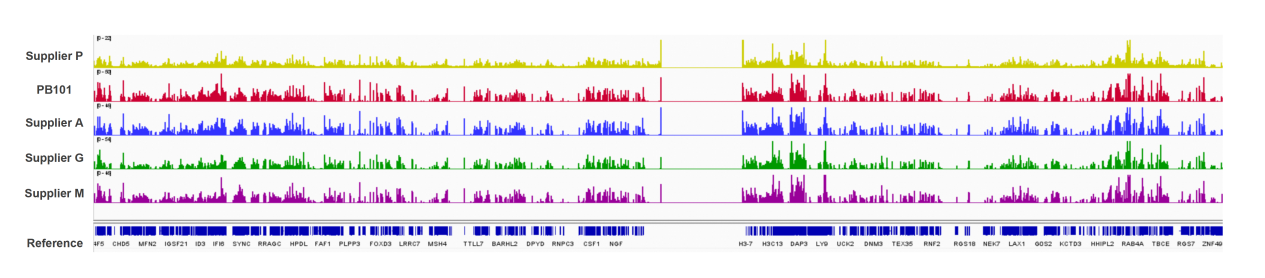
图3.以H3K4me3为靶点的不同Protein A/G磁珠的IGV全局视图
3. 结合效率高
使用Protein A/G磁珠Vazyme #PB101,搭配Vazyme #RA1003,以1 mg 293T细胞裂解蛋白为样本,对 Flag标签蛋白进行免疫沉淀实验,经1×SDS-PAGE Sample Loading Buffer高温变性洗脱后,搭配抗轻链二抗,通过Western Blot检测Flag标签蛋白富集水平。结果显示,PB101蛋白结合效率更高。
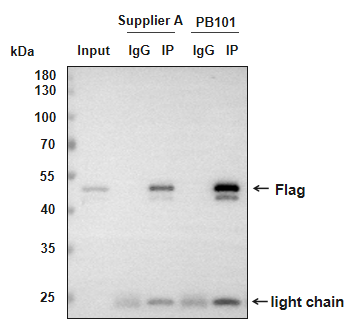
图4.Flag标签蛋白的Western Blot检测结果图



2 ~ 8℃保存,根据不同目的地调整运输方式。
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
- 作者
- 内容
- 询问日期
 文献和实验
文献和实验Combined 3C-ChIP-Cloning (6C) Assay: A Tool to Unravel Protein-Mediated Genome Architecture
antibiotic (see Step 51). Ligation buffer (10X) Dilute the 10X stock to 1.15X for Step 18. Magnetic beads, Protein A-conjugated Magnetic beads, Protein G-conjugated PCR buffer (10X) PCR primers, T3 and T7 Phenol
Detection of Bacterial Protein Toxins by Solid Phase Magnetic Immunocapture and Mass Spectrometry
, can be efficiently enriched by means of magnetic immunocapture using antibody functionalized paramagnetic beads. The method was successfully interfaced by the on-beads and off-beads detection using matrix-assisted laser desorption/ionization time-of-flight mass
Nuclear magnetic resonance (NMR) has become a well-established method to characterize the structures of biomolecules in solution. High-quality structures are now produced, thanks to both experimental and computational developments, allowing