相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 英文名:
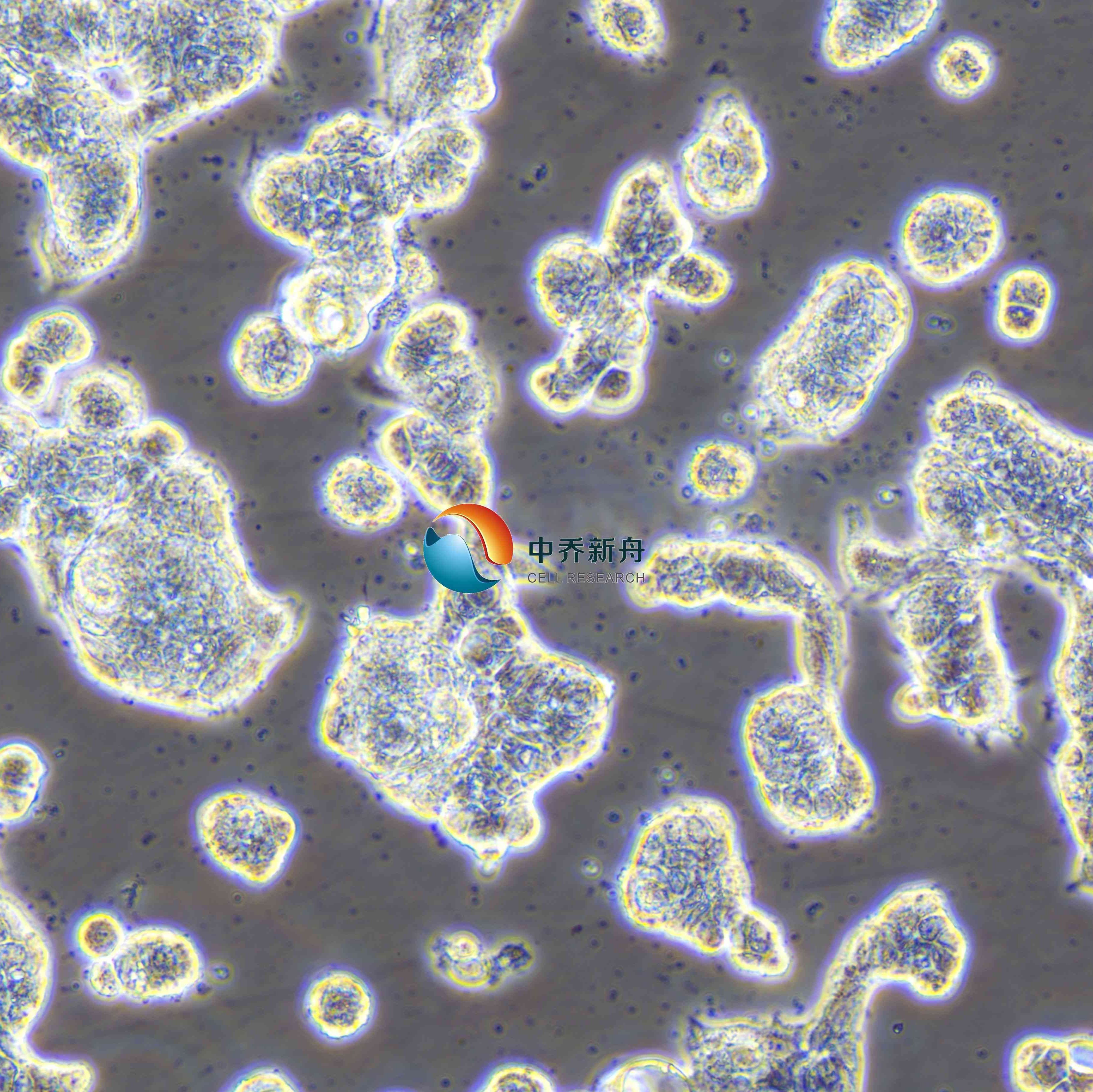
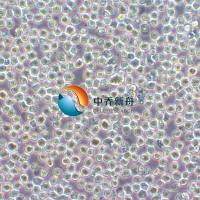
SNU-719
- 库存:
100
- 供应商:
中乔新舟
- 品系:
细胞系
- 运输方式:
常温
- 年限:
液氮长期
- 生长状态:
贴壁生长
- 规格:
T25
|
产品名称 |
SNU-719人胃癌细胞 |
|
货号 |
ZQ0959 |
|
产品介绍 |
这株细胞建立于1984年4月。病人是个吸烟者,100包/年。 SNU-719细胞系可以用于多种生物学研究,包括药物筛选、病理机制研究以及癌症治疗的实验模型。这些细胞在肿瘤生物学研究、药物筛选与研发、病毒感染研究以及细胞生物学研究等领域有着广泛的应用。 |
|
种属 |
人 |
|
性别/年龄 |
男/53岁 |
|
组织 |
胃 |
|
疾病 |
胃癌 |
|
生物安全等级 |
BSL-1 |
|
STR位点信息 |
Amelogenin: X,Y CSF1PO :12 D3S1358 :15 D5S818 :11,12 D7S820: 10,11 D8S1179: 12 D13S317: 10,11 D16S539: 9,10 D18S51: 13,15 D21S11: 29,30 FGA: 21,23 Penta D: 10,11 Penta E: 11,16 TH01: 7,9 TPOX: 8,9 vWA: 16,18 |
|
细胞类型 |
肿瘤细胞 |
|
形态学 |
上皮样 |
|
生长方式 |
贴壁 |
|
倍增时间 |
43 hours (PubMed=29435981) |
|
培养基和添加剂 |
RPMI-1640基础培养基(品牌:中乔新舟 货号:ZQ-200)+10%FBS(货号:ZQ0500)+1%PS(品牌:中乔新舟 货号:CSP006) |
|
推荐完全培养基货号 |
ZM0959 |
|
培养条件 |
95%空气,5%二氧化碳;37℃ |
|
抗原表达/受体表达 |
*** |
|
基因表达 |
*** |
|
保藏机构 |
KCLB |
|
供应限制 |
仅供科研使用 |
上海中乔新舟生物科技有限公司成立于2011年,历经十多年发展,主要专注于细胞生物学产品的研究和开发,专注于为药企、各类科研机构及CRO企业提供符合标准规范的细胞培养服务、细胞培养基、细胞检测试剂盒、细胞培养试剂,胎牛血清和细胞生物学技术服务等。
公司一直致力于为高等院校、研究机构、医院、CRO及CDMO企业提供细胞培养完整解决方案,这些产品旨在满足细胞培养的多样需求,确保实验和研究的有效进行。引用中乔新舟(ZQXZBIO)产品和服务的文献超数千篇。

产品服务
细胞资源:原代细胞、细胞株、干细胞、示踪细胞、耐药株细胞、永生化细胞等基因工程细胞。
试剂产品:胎牛血清、完全培养基(适用于原代细胞及细胞株)、无血清培养基、基础培养基、细胞转染试剂、重组因子、胰酶和双抗等等细胞培养所有实验相关产品。
技术服务:稳转株构建、原代细胞分离、特殊培养基定制服务、细胞检测等。

目前产品已经畅销国内30多个省市,与客户建立长期的合作伙伴关系,共同实现成功。全体员工将不懈努力,继续为科研人员提供优良的产品和服务,致力成为全球细胞培养领域的参与者。
企业愿景
致力于成为国内细胞培养基产业的佼佼者,生物医药领域上游原材料的优良提供商。
企业使命
成长为专业细胞系及原代细胞培养供应商、专业细胞培养基及培养试剂生产商。
企业荣誉

风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验PubMed=9033653; DOI=10.1002/(SICI)1097-0215(19970207)70:4<443::AID-IJC12>3.0.CO;2-G
Park J.-G., Yang H.-K., Kim W.-H., Chung J.-K., Kang M.-S., Lee J.-H., Oh J.H., Park H.-S., Yeo K.-S., Kang S.H., Song S.-Y., Kang Y.K., Bang Y.-J., Kim Y.I., Kim J.-P.
Establishment and characterization of human gastric carcinoma cell lines.
Int. J. Cancer 70:443-449(1997)
PubMed=10812166; DOI=10.1016/S0165-4608(99)00217-4
Chun Y.-H., Kil J.-I., Suh Y.-S., Kim S.-H., Kim H., Park S.-H.
Characterization of chromosomal aberrations in human gastric carcinoma cell lines using chromosome painting.
Cancer Genet. Cytogenet. 119:18-25(2000)
PubMed=15016554; DOI=10.1016/j.virol.2003.12.005
Oh S.T., Seo J.S., Moon U.Y., Kang K.H., Shin D.-J., Yoon S.K., Kim W.-H., Park J.-G., Lee S.K.
A naturally derived gastric cancer cell line shows latency I Epstein-Barr virus infection closely resembling EBV-associated gastric cancer.
Virology 320:330-336(2004)
PubMed=15723654; DOI=10.1111/j.1349-7006.2005.00016.x
Takada H., Imoto I., Tsuda H., Sonoda I., Ichikura T., Mochizuki H., Okanoue T., Inazawa J.
Screening of DNA copy-number aberrations in gastric cancer cell lines by array-based comparative genomic hybridization.
Cancer Sci. 96:100-110(2005)
PubMed=19956504; DOI=10.4143/crt.2005.37.1.1
Ku J.-L., Park J.-G.
Biology of SNU cell lines.
Cancer Res. Treat. 37:1-19(2005)
PubMed=22460905; DOI=10.1038/nature11003
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=24807215; DOI=10.1038/ncomms4830
Liu J.-F., McCleland M.L., Stawiski E.W., Gnad F., Mayba O., Haverty P.M., Durinck S., Chen Y.-J., Klijn C., Jhunjhunwala S., Lawrence M., Liu H.-B., Wan Y.-N., Chopra V.S., Yaylaoglu M.B., Yuan W.-L., Ha C., Gilbert H.N., Reeder J., Pau G., Stinson J., Stern H.M., Manning G., Wu T.D., Neve R.M., de Sauvage F.J., Modrusan Z., Seshagiri S., Firestein R., Zhang Z.-M.
Integrated exome and transcriptome sequencing reveals ZAK isoform usage in gastric cancer.
Nat. Commun. 5:3830.1-3830.8(2014)
PubMed=25960936; DOI=10.4161/21624011.2014.954893
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26889033; DOI=10.1128/JVI.00060-16
Kanda T., Furuse Y., Oshitani H., Kiyono T.
Highly efficient CRISPR/Cas9-mediated cloning and functional characterization of gastric cancer-derived Epstein-Barr virus strains.
J. Virol. 90:4383-4393(2016)
PubMed=29435981; DOI=10.1002/ijc.31304
Kim H.J., Kang S.K., Kwon W.S., Kim T.S., Jeong I., Jeung H.-C., Kragh M., Horak I.D., Chung H.C., Rha S.Y.
Forty-nine gastric cancer cell lines with integrative genomic profiling for development of c-MET inhibitor.
Int. J. Cancer 143:151-159(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. III, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. III, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)
 技术资料
技术资料暂无技术资料 索取技术资料




![MCF7 [MCF-7]-luc人乳腺癌细胞-荧光素酶标记](https://img1.dxycdn.com/p/s14/2025/0407/833/6180717660432219091.jpg!wh200)


