相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 英文名:
KMS-11
- 库存:
100
- 供应商:
中乔新舟
- 品系:
细胞系
- 运输方式:
常温
- 年限:
液氮长期
- 生长状态:
半贴半悬
- 规格:
T25
|
产品名称 |
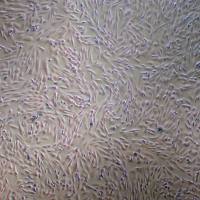
KMS-11人骨髓瘤细胞 |
|
货号 |
ZQ0840 |
|
产品介绍 |
KMS-11是一种人多发性骨髓瘤克隆细胞系,源自一名67岁患有骨髓瘤的女性的胸腔积液。在癌症研究中有着广泛的应用,包括药物筛选、病理机制研究和基因功能分析。它们是理解骨髓瘤发展和寻找新治疗方法的重要工具。 |
|
种属 |
人 |
|
性别/年龄 |
女/67岁 |
|
组织 |
胸腔积液 |
|
疾病 |
多发性骨髓瘤 |
|
细胞类型 |
肿瘤细胞 |
|
形态学 |
淋巴样细胞 |
|
生长方式 |
半贴半悬 |
|
倍增时间 |
大约36 hours (PubMed=2519219); 70 hours (PubMed=25984343); 50 hours (Note=Lot 03062008), ~2 days (Note=Lot 10142009), ~31 hours (Note=Lot 10072016), ~39 hours (Note=Lot 10222021) (JCRB=JCRB1179) |
|
培养基和添加剂 |
RPMI-1640(品牌:中乔新舟 货号:ZQ-200)+10%胎牛血清(中乔新舟 货号:ZQ0500)+1%P/S(中乔新舟 货号:CSP006) |
|
推荐完全培养基货号 |
ZM0840 |
|
生物安全等级 |
BSL-1 |
|
培养条件 |
95%空气,5%二氧化碳;37℃ |
|
STR位点信息 |
Amelogenin: X CSF1PO: 13 D3S1358: 15 D5S818: 10,12 D7S820: 11,12 D8S1179: 13 D13S317: 12 D16S539 :9,13 D18S51: 13,14 D21S11: 28,30 FGA: 22,24 Penta D: 11,12 Penta E: 15,16 TH01: 6,9 TPOX: 12 vWA :17,18 |
|
抗原表达/受体表达 |
*** |
|
基因表达 |
*** |
|
保藏机构 |
JCRB; JCRB1179 |
|
供应限制 |
仅供科研使用 |
上海中乔新舟生物科技有限公司成立于2011年,历经十多年发展,主要专注于细胞生物学产品的研究和开发,专注于为药企、各类科研机构及CRO企业提供符合标准规范的细胞培养服务、细胞培养基、细胞检测试剂盒、细胞培养试剂,胎牛血清和细胞生物学技术服务等。
公司一直致力于为高等院校、研究机构、医院、CRO及CDMO企业提供细胞培养完整解决方案,这些产品旨在满足细胞培养的多样需求,确保实验和研究的有效进行。引用中乔新舟(ZQXZBIO)产品和服务的文献超数千篇。

产品服务
细胞资源:原代细胞、细胞株、干细胞、示踪细胞、耐药株细胞、永生化细胞等基因工程细胞。
试剂产品:胎牛血清、完全培养基(适用于原代细胞及细胞株)、无血清培养基、基础培养基、细胞转染试剂、重组因子、胰酶和双抗等等细胞培养所有实验相关产品。
技术服务:稳转株构建、原代细胞分离、特殊培养基定制服务、细胞检测等。

目前产品已经畅销国内30多个省市,与客户建立长期的合作伙伴关系,共同实现成功。全体员工将不懈努力,继续为科研人员提供优良的产品和服务,致力成为全球细胞培养领域的参与者。
企业愿景
致力于成为国内细胞培养基产业的佼佼者,生物医药领域上游原材料的优良提供商。
企业使命
成长为专业细胞系及原代细胞培养供应商、专业细胞培养基及培养试剂生产商。
企业荣誉


风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验
PubMed=2519219
Ohtsuki T., Yawata Y., Namba M.
Establishment and characterization of five human myeloma cell lines.
Hum. Cell 2:297-303(1989)
PubMed=2768132; DOI=10.1007/BF02623725
Namba M., Ohtsuki T., Mori M., Togawa A., Wada H., Sugihara T., Yawata Y., Kimoto T.
Establishment of five human myeloma cell lines.
In Vitro Cell. Dev. Biol. 25:723-729(1989)
PubMed=8943038; DOI=10.1073/pnas.93.24.13931
Bergsagel P.L., Chesi M., Nardini E., Brents L.A., Kirby S.L., Kuehl W.M.
Promiscuous translocations into immunoglobulin heavy chain switch regions in multiple myeloma.
Proc. Natl. Acad. Sci. U.S.A. 93:13931-13936(1996)
PubMed=9787181; DOI=10.1182/blood.V92.9.3410
Sakai A., Thieblemont C., Wellmann A., Jaffe E.S., Raffeld M.
PTEN gene alterations in lymphoid neoplasms.
Blood 92:3410-3415(1998)
DOI=10.1002/1438-826X(200005)1:1<48::AID-GNFD48>3.0.CO;2-B
Otsuki T., Yamada O., Yata K., Nakazawa N., Taniwaki M., Sakaguchi H., Yawata Y., Ueki A.
Genetic and biological characterization of human myeloma cell lines: an overview of the lines established at Kawasaki Medical School.
Gene Funct. Dis. 1:48-56(2000)
DOI=10.1007/0-306-46877-8_4
Jernberg-Wiklund H., Nilsson K.
Multiple myeloma cell lines.
(In) Human cell culture. Vol. 3. Cancer cell lines part 3; Masters J.R.W., Palsson B.O. (eds.); pp.81-155; Kluwer Academic Publishers; New York (2000)
PubMed=10936422; DOI=10.1016/S0145-2126(99)00195-2
Drexler H.G., Matsuo Y.
Malignant hematopoietic cell lines: in vitro models for the study of multiple myeloma and plasma cell leukemia.
Leuk. Res. 24:681-703(2000)
DOI=10.1016/B978-0-12-221970-2.50457-5
Drexler H.G.
The leukemia-lymphoma cell line factsbook.
(In) ISBN 9780122219702; pp.1-733; Academic Press; London (2001)
PubMed=11157491; DOI=10.1182/blood.V97.3.729
Chesi M., Brents L.A., Ely S.A., Bais C., Robbiani D.F., Mesri E.A., Kuehl W.M., Bergsagel P.L.
Activated fibroblast growth factor receptor 3 is an oncogene that contributes to tumor progression in multiple myeloma.
Blood 97:729-736(2001)
PubMed=15215163; DOI=10.1016/S0002-9440(10)63276-2
Inoue J., Otsuki T., Hirasawa A., Imoto I., Matsuo Y., Shimizu S., Taniwaki M., Inazawa J.
Overexpression of PDZK1 within the 1q12-q22 amplicon is likely to be associated with drug-resistance phenotype in multiple myeloma.
Am. J. Pathol. 165:71-81(2004)
PubMed=16956823
Bataille R., Jego G., Robillard N., Barille-Nion S., Harousseau J.-L., Moreau P., Amiot M., Pellat-Deceunynck C.
The phenotype of normal, reactive and malignant plasma cells Identification of 'many and multiple myelomas' and of new targets for myeloma therapy.
Haematologica 91:1234-1240(2006)
PubMed=17171682; DOI=10.1002/gcc.20404
Lombardi L., Poretti G., Mattioli M., Fabris S., Agnelli L., Bicciato S., Kwee I., Rinaldi A., Ronchetti D., Verdelli D., Lambertenghi-Deliliers G., Bertoni F., Neri A.
Molecular characterization of human multiple myeloma cell lines by integrative genomics: insights into the biology of the disease.
Genes Chromosomes Cancer 46:226-238(2007)
PubMed=17692805; DOI=10.1016/j.ccr.2007.07.003
Keats J.J., Fonseca R., Chesi M., Schop R., Baker A., Chng W.-J., Van Wier S., Tiedemann R., Shi C.-X., Sebag M., Braggio E., Henry T., Zhu Y.-X., Fogle H., Price-Troska T.L., Ahmann G.J., Mancini C., Brents L.A., Kumar S.K., Greipp P., Dispenzieri A., Bryant B., Mulligan G., Bruhn L., Barrett M.T., Valdez R., Trent J.M., Stewart A.K., Carpten J.D., Bergsagel P.L.
Promiscuous mutations activate the noncanonical NF-kappaB pathway in multiple myeloma.
Cancer Cell 12:131-144(2007)
PubMed=18647998; DOI=10.1093/jncimonographs/lgn011
Dib A., Gabrea A., Glebov O.K., Bergsagel P.L., Kuehl W.M.
Characterization of MYC translocations in multiple myeloma cell lines.
J. Natl. Cancer Inst. Monogr. 39:25-31(2008)
PubMed=18700954; DOI=10.1186/1755-8794-1-37
Ronchetti D., Lionetti M., Mosca L., Agnelli L., Andronache A., Fabris S., Lambertenghi-Deliliers G., Neri A.
An integrative genomic approach reveals coordinated expression of intronic miR-335, miR-342, and miR-561 with deregulated host genes in multiple myeloma.
BMC Med. Genomics 1:37.1-37.9(2008)
PubMed=19306352; DOI=10.1002/gcc.20660
Lionetti M., Agnelli L., Mosca L., Fabris S., Andronache A., Todoerti K., Ronchetti D., Lambertenghi-Deliliers G., Neri A.
Integrative high-resolution microarray analysis of human myeloma cell lines reveals deregulated miRNA expression associated with allelic imbalances and gene expression profiles.
Genes Chromosomes Cancer 48:521-531(2009)
PubMed=21173094; DOI=10.3324/haematol.2010.033456
Moreaux J., Klein B., Bataille R., Descamps G., Maiga S., Hose D., Goldschmidt H., Jauch A., Reme T., Jourdan M., Amiot M., Pellat-Deceunynck C.
A high-risk signature for patients with multiple myeloma established from the molecular classification of human myeloma cell lines.
Haematologica 96:574-582(2011)
PubMed=22460905; DOI=10.1038/nature11003
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25984343; DOI=10.1038/sdata.2014.35
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25688540; DOI=10.1002/cyto.a.22643
Maiga S., Brosseau C., Descamps G., Dousset C., Gomez-Bougie P., Chiron D., Menoret E., Kervoelen C., Vie H., Cesbron A., Moreau-Aubry A., Amiot M., Pellat-Deceunynck C.
A simple flow cytometry-based barcode for routine authentication of multiple myeloma and mantle cell lymphoma cell lines.
Cytometry A 87:285-288(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yua
【求助】骨髓瘤细胞的体内接种问题 参与者:qj_82 请教各位:骨髓瘤细胞如果要进行乳鼠或者大鼠的体内接种,是采用腹腔注射,还是皮下注射?还是直接注射在脾脏上?细胞密度要达到多少才有比较理想的接种效果? 参与者:zhangzhanli2008 皮下1*10的7次方-3*10的7次方/ml 腹腔1*10的6次方/ml
1、脾细胞和骨髓瘤细胞一般按2:1-5:1的密度混合,有的报道可以到10:1,不过总体来说我们常选3:1-5:1,记住,原则是脾细胞的数量比骨髓瘤的多。2、融合前,一般用HAT培养基做饲养细胞铺板,这是融合前的准备工作,因为饲养细胞分泌的某些细胞因子有助与融合的杂交瘤细胞生长,并且提供一定的细胞密度。通常是可以融合前一天做饲养细胞,老鼠选ICR或是BALB/C与ICR杂交的鼠,一只老鼠可以铺2-3块96孔板。3、我们融合选用96孔细胞培养板。融合时将脾细胞与骨髓瘤混合后离心,然后用手掌稍稍搓
骨髓瘤细胞能产生并分泌大量的免疫球蛋白,这样的瘤细胞融合后,可能影响或降低所分泌抗体的滴度,所以必须选育出非分泌免疫球蛋白缺陷型的骨髓瘤细胞。 选择骨髓瘤细胞的条件:①该瘤细胞系的来源应与制备脾细胞小鼠为同一品系,以便两者的组织相容性抗原一致;②骨髓瘤细胞必须是静息状态,不产生γ球蛋白或不分泌到细胞外;③骨髓瘤细胞生长需要一个较高的细胞密度,最好106个细胞/ml;④生长速度快,繁殖时间短。 目前常用的Balb/c小鼠产生的骨髓瘤细胞株有:①S194/5XXO・BU・1;②SP2/
 技术资料
技术资料暂无技术资料 索取技术资料






![CV-1 [Part of the Wistar Special Collection]非洲绿猴肾细胞](https://img1.dxycdn.com/p/s14/2024/1226/077/8615103296157512781.jpg!wh200)