相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 保存条件:
-20
- 保质期:
2年
- 英文名:
pBBR1MCS2
- 库存:
100
- 供应商:
上海烜雅生物科技有限公司
- 规格:
干粉/液体
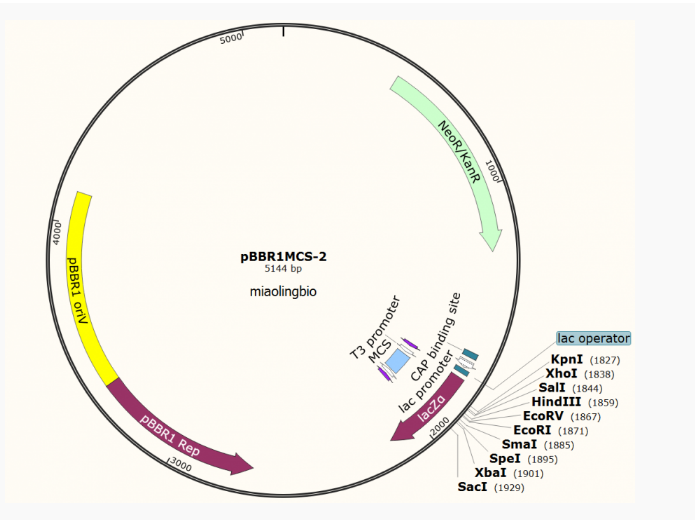
名称:pBBR1MCS-2广宿主表达质粒
别称: pBBR1MCS2
| 启动子: | Lac |
|---|---|
| 复制子: | pBBR1 |
| 质粒分类: | pBBR1MCS系列质粒 |
| 质粒大小: | 5144bp |
| 质粒标签: | LacZ |
| 原核抗性: | Kan |
| 克隆菌株: | DH5a |
| 培养条件: | 37度 |
| 表达宿主: | 广宿主 |
| 培养条件: | 参考相关文献 |
| 5'测序引物: | M13R:CAGGAAACAGCTATGACC |
| 3'测序引物: | M13F:TGTAAAACGACGGCCAGT |
| 备注: | 低拷贝质粒 |
质粒属性
| 载体宿主: | 广宿主 |
|---|---|
| 载体用途: | 蛋白表达 |
| 基因种属: | 空载体 |
| 基因类型: | ORF |
| 原核抗性: | Kan |
| 筛选标记: | |
| 荧光蛋白: |
质粒简介
广宿主穿梭质粒pBBR1MCS-2是基于质粒pBBR1MCS构建的[1],该质粒是由Kovach等构建,已经证实可以在许多革兰氏阴性菌中进行复制[2],包括Acetobacter xylinum、Alcaligenes eutrophus、Bartonella bacilliformis、百日咳杆菌(Bordetella spp.)、布鲁氏菌(Brucella spp.)、Caulobacter crescentus、大肠杆菌、Paracoccous denitrificans、荧光假单胞菌(Pseudomonas fluorescens)、恶臭假单胞菌(P. putida)、苜蓿根瘤菌(Rhizobium meliloti)、R. leguminosarum by. Viciae、球形红假单胞菌(Rhodobacter sphaeroides)、鼠伤寒沙门氏菌(Salmonella typhimurium)、霍乱弧菌(Vibrio cholerae)和Xanthomonas campestris等。
质粒图谱
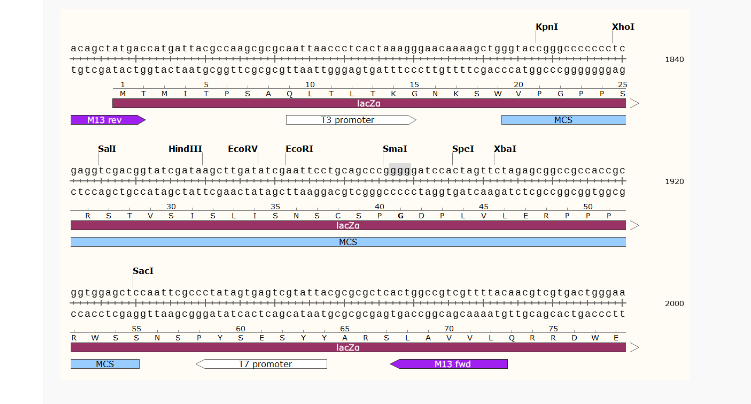
质粒序列
LOCUS Exported 5144 bp ds-DNA circular SYN 10-SEP-2016
DEFINITION synthetic circular DNA
ACCESSION .
VERSION .
KEYWORDS pBBR1MCS-2
SOURCE synthetic DNA construct
ORGANISM synthetic DNA construct
REFERENCE 1 (bases 1 to 5144)
AUTHORS .
TITLE Direct Submission
JOURNAL Exported Saturday, September 10, 2016 from SnapGene Viewer 3.1.4
http://www.miaolingbio.com
FEATURES Location/Qualifiers
source 1..5144
/organism="synthetic DNA construct"
/mol_type="other DNA"
CDS 458..1252
/codon_start=1
/gene="aph(3')-II (or nptII)"
/product="aminoglycoside phosphotransferase from Tn5"
/note="NeoR/KanR"
/note="confers resistance to neomycin, kanamycin, and G418
(Geneticin(R))"
/translation="MIEQDGLHAGSPAAWVERLFGYDWAQQTIGCSDAAVFRLSAQGRP
VLFVKTDLSGALNELQDEAARLSWLATTGVPCAAVLDVVTEAGRDWLLLGEVPGQDLLS
SHLAPAEKVSIMADAMRRLHTLDPATC-PFDHQAKHRIERARTRMEAGLVDQDDLDEEHQ
GLAPAELFARLKARMPDGEDLVVTHGDACLPNIMVENGRFSGFIDCGRLGVADRYQDIA
LATRDIAEELGGEWADRFLVLYGIAAPDSQRIAFYRLLDEFF"
protein_bind 1657..1678
/bound_moiety="E. coli catabolite activator protein"
/note="CAP binding site"
/note="CAP binding activates transcription in the presence
of cAMP."
promoter 1693..1723
/note="lac promoter"
/note="promoter for the E. coli lac operon"
protein_bind 1731..1747
/bound_moiety="lac repressor encoded by lacI"
/note="lac operator"
/note="The lac repressor binds to the lac operator to
inhibit transcription in E. coli. This inhibition can be
relieved by adding lactose or
isopropyl-beta-D-thiogalactopyranoside (IPTG)."
primer_bind 1755..1771
/note="M13 rev"
/note="common sequencing primer, one of multiple similar
variants"
CDS 1767..2132
/codon_start=1
/gene="lacZ fragment"
/product="LacZ-alpha fragment of beta-galactosidase"
/note="lacZ-alpha"
/translation="MTMITPSAQLTLTKGNKSWVPGPPSRSTVSISLISNSCSPGDPLV
LERPPPRWSSNSPYSESYYARSLAVVLQRRDWENPGVTQLNRLAAHPPFASWRNSEEAR
TDRPSQQLRSLNGEWKL"
promoter 1792..1810
/note="T3 promoter"
/note="promoter for bacteriophage T3 RNA polymerase"
misc_feature 1823..1930
/note="MCS"
/note="pBluescript multiple cloning site"
primer_bind 1840..1856
/note="KS primer"
/note="common sequencing primer, one of multiple similar
variants"
primer_bind complement(1890..1906)
/note="SK primer"
/note="common sequencing primer, one of multiple similar
variants"
promoter complement(1939..1957)
/note="T7 promoter"
/note="promoter for bacteriophage T7 RNA polymerase"
primer_bind complement(1967..1983)
/note="M13 fwd"
/note="common sequencing primer, one of multiple similar
variants"
CDS complement(2690..3352)
/codon_start=1
/product="replication protein for the broad-host-range
plasmid pBBR1 from Bordetella bronchiseptica"
/note="pBBR1 Rep"
/translation="MATQSREIGIQAKNKPGHWVQTERKAHEAWAGLIARKPTAAMLLH
HLVAQMGHQNAVVVSQKTLSKLIGRSLRTVQYAVKDLVAERWISVVKLNGPGTVSAYVV
NDRVAWGQPRDQLRLSVFSAAVVVDHDDQDESLLGHGDLRRIPTLYPGEQQLPTGPGEE
PPSQPGIPGMEPDLPALTETEEWERRGQQRLPMPDEPCFLDDGEPLEPPTRVTLPRR"
rep_origin 3353..4122
/note="pBBR1 oriV"
/note="replication origin of the broad-host-range plasmid
pBBR1 from Bordetella bronchiseptica; requires the pBBR1
Rep protein for replication"
ORIGIN
1 accttcggga gcgcctgaag cccgttctgg acgccctggg gccgttgaat cgggatatgc
61 aggccaaggc cgccgcgatc atcaaggccg tgggcgaaaa gctgctgacg gaacagcggg
121 aagtccagcg ccagaaacag gcccagcgcc agcaggaacg cgggcgcgca catttccccg
181 aaaagtgcca cctgggatga atgtcagcta ctgggctatc tggacaaggg aaaacgcaag
241 cgcaaagaga aagcaggtag cttgcagtgg gcttacatgg cgatagctag actgggcggt
301 tttatggaca gcaagcgaac cggaattgcc agctggggcg ccctctggta aggttgggaa
361 gccctgcaaa gtaaactgga tggctttctt gccgccaagg atctgatggc gcaggggatc
421 aagatctgat caagagacag gatgaggatc gtttcgcatg attgaacaag atggattgca
481 cgcaggttct ccggccgctt gggtggagag gctattcggc tatgactggg cacaacagac
541 aatcggctgc tctgatgccg ccgtgttccg gctgtcagcg caggggcgcc cggttctttt
601 tgtcaagacc gacctgtccg gtgccctgaa tgaactgcag gacgaggcag cgcggctatc
661 gtggctggcc acgacgggcg ttccttgcgc agctgtgctc gacgttgtca ctgaagcggg
721 aagggactgg ctgctattgg gcgaagtgcc ggggcaggat ctcctgtcat ctcaccttgc
781 tcctgccgag aaagtatcca tcatggctga tgcaatgcgg cggctgcata cgcttgatcc
841 ggctacctgc ccattcgacc accaagcgaa acatcgcatc gagcgagcac gtactcggat
901 ggaagccggt cttgtcgatc aggatgatct ggacgaagag catcaggggc tcgcgccagc
961 cgaactgttc gccaggctca aggcgcgcat gcccgacggc gaggatctcg tcgtgaccca
1021 tggcgatgcc tgcttgccga atatcatggt ggaaaatggc cgcttttctg gattcatcga
1081 ctgtggccgg ctgggtgtgg cggaccgcta tcaggacata gcgttggcta cccgtgatat
1141 tgctgaagag cttggcggcg aatgggctga ccgcttcctc gtgctttacg gtatcgccgc
1201 tcccgattcg cagcgcatcg ccttctatcg ccttcttgac gagttcttct gagcgggact
1261 ctggggttcg aaatgaccga ccaagcgacg cccaacctgc catcacgaga tttcgattcc
1321 accgccgcct tctatgaaag gttgggcttc ggaatcgttt tccgggacgc cggctggatg
1381 atcctccagc gcggggatct catgctggag ttcttcgccc acccccatgg gcaaatatta
1441 tacgcaaggc gacaaggtgc tgatgccgct ggcgattcag gttcatcatg ccgtttgtga
1501 tggcttccat gtcggcagaa tgcttaatga attacaacag tttttatgca tgcgcccaat
1561 acgcaaaccg cctctccccg cgcgttggcc gattcattaa tgcagctggc acgacaggtt
1621 tcccgactgg aaagcgggca gtgagcgcaa cgcaattaat gtgagttagc tcactcatta
1681 ggcaccccag gctttacact ttatgcttcc ggctcgtatg ttgtgtggaa ttgtgagcgg
1741 ataacaattt cacacaggaa acagctatga ccatgattac gccaagcgcg caattaaccc
1801 tcactaaagg gaacaaaagc tgggtaccgg gccccccctc gaggtcgacg gtatcgataa
1861 gcttgatatc gaattcctgc agcccggggg atccactagt tctagagcgg ccgccaccgc
1921 ggtggagctc caattcgccc tatagtgagt cgtattacgc gcgctcactg gccgtcgttt
1981 tacaacgtcg tgactgggaa aaccctggcg ttacccaact taatcgcctt gcagcacatc
2041 cccctttcgc cagctggcgt aatagcgaag aggcccgcac cgatcgccct tcccaacagt
2101 tgcgcagcct gaatggcgaa tggaaattgt aagcgttaat attttgttaa aattcgcgtt
2161 aaatttttgt taaatcagct cattttttaa ccaataggcc gactgcgatg agtggcaggg
2221 cggggcgtaa tttttttaag gcagttattg gtgcccttaa acgcctggtg ctacgcctga
2281 ataagtgata ataagcggat gaatggcaga aattcgaaag caaattcgac ccggtcgtcg
2341 gttcagggca gggtcgttaa atagccgctt atgtctattg ctggtttacc ggtttattga
2401 ctaccggaag cagtgtgacc gtgtgcttct caaatgcctg aggccagttt gctcaggctc
2461 tccccgtgga ggtaataatt gacgatatga tcatttattc tgcctcccag agcctgataa
2521 aaacggtgaa tccgttagcg aggtgccgcc ggcttccatt caggtcgagg tggcccggct
2581 ccatgcaccg cgacgcaacg cggggaggca gacaaggtat agggcggcga ggcggctaca
2641 gccgatagtc tggaacagcg cacttacggg ttgctgcgca acccaagtgc taccggcgcg
2701 gcagcgtgac ccgtgtcggc ggctccaacg gctcgccatc gtccagaaaa cacggctcat
2761 cgggcatcgg caggcgctgc tgcccgcgcc gttcccattc ctccgtttcg gtcaaggctg
2821 gcaggtctgg ttccatgccc ggaatgccgg gctggctggg cggctcctcg ccggggccgg
2881 tcggtagttg ctgctcgccc ggatacaggg tcgggatgcg gcgcaggtcg ccatgcccca
2941 acagcgattc gtcctggtcg tcgtgatcaa ccaccacggc ggcactgaac accgacaggc
3001 gcaactggtc gcggggctgg ccccacgcca cgcggtcatt gaccacgtag gccgacacgg
3061 tgccggggcc gttgagcttc acgacggaga tccagcgctc ggccaccaag tccttgactg
3121 cgtattggac cgtccgcaaa gaacgtccga tgagcttgga aagtgtcttc tggctgacca
3181 ccacggcgtt ctggtggccc atctgcgcca cgaggtgatg cagcagcatt gccgccgtgg
3241 gtttcctcgc aataagcccg gcccacgcct catgcgcttt gcgttccgtt tgcacccagt
3301 gaccgggctt gttcttggct tgaatgccga tttctctgga ctgcgtggcc atgcttatct
3361 ccatgcggta gggtgccgca cggttgcggc accatgcgca atcagctgca acttttcggc
3421 agcgcgacaa caattatgcg ttgcgtaaaa gtggcagtca attacagatt ttctttaacc
3481 tacgcaatga gctattgcgg ggggtgccgc aatgagctgt tgcgtacccc ccttttttaa
3541 gttgttgatt tttaagtctt tcgcatttcg ccctatatct agttctttgg tgcccaaaga
3601 agggcacccc tgcggggttc ccccacgcct tcggcgcggc tccccctccg gcaaaaagtg
3661 gcccctccgg ggcttgttga tcgactgcgc ggccttcggc cttgcccaag gtggcgctgc
3721 ccccttggaa cccccgcact cgccgccgtg aggctcgggg ggcaggcggg cgggcttcgc
3781 cttcgactgc ccccactcgc ataggcttgg gtcgttccag gcgcgtcaag gccaagccgc
3841 tgcgcggtcg ctgcgcgagc cttgacccgc cttccacttg gtgtccaacc ggcaagcgaa
3901 gcgcgcaggc cgcaggccgg aggcttttcc ccagagaaaa ttaaaaaaat tgatggggca
3961 aggccgcagg ccgcgcagtt ggagccggtg ggtatgtggt cgaaggctgg gtagccggtg
4021 ggcaatccct gtggtcaagc tcgtgggcag gcgcagcctg tccatcagct tgtccagcag
4081 ggttgtccac gggccgagcg aagcgagcca gccggtggcc gctcgcggcc atcgtccaca
4141 tatccacggg ctggcaaggg agcgcagcga ccgcgcaggg cgaagcccgg agagcaagcc
4201 cgtagggcgc cgcagccgcc gtaggcggtc acgactttgc gaagcaaagt ctagtgagta
4261 tactcaagca ttgagtggcc cgccggaggc accgccttgc gctgcccccg tcgagccggt
4321 tggacaccaa aagggagggg caggcatggc ggcatacgcg atcatgcgat gcaagaagct
4381 ggcgaaaatg ggcaacgtgg cggccagtct caagcacgcc taccgcgagc gcgagacgcc
4441 caacgctgac gccagcagga cgccagagaa cgagcactgg gcggccagca gcaccgatga
4501 agcgatgggc cgactgcgcg agttgctgcc agagaagcgg cgcaaggacg ctgtgttggc
4561 ggtcgagtac gtcatgacgg ccagcccgga atggtggaag tcggccagcc aagaacagca
4621 ggcggcgttc ttcgagaagg cgcacaagtg gctggcggac aagtacgggg cggatcgcat
4681 cgtgacggcc agcatccacc gtgacgaaac cagcccgcac atgaccgcgt tcgtggtgcc
4741 gctgacgcag gacggcaggc tgtcggccaa ggagttcatc ggcaacaaag cgcagatgac
4801 ccgcgaccag accacgtttg cggccgctgt ggccgatcta gggctgcaac ggggcatcga
4861 gggcagcaag gcacgtcaca cgcgcattca ggcgttctac gaggccctgg agcggccacc
4921 agtgggccac gtcaccatca gcccgcaagc ggtcgagcca cgcgcctatg caccgcaggg
4981 attggccgaa aagctgggaa tctcaaagcg cgttgagacg ccggaagccg tggccgaccg
5041 gctgacaaaa gcggttcggc aggggtatga gcctgcccta caggccgccg caggagcgcg
5101 tgagatgcgc aagaaggccg atcaagccca agagacggcc cgag
//
质粒菌株产品操作说明书
一、扩增流程
收到产品后,请先根据产品管壁标签来判断产品形式,并在扩增前准确查找该质粒菌株的抗性、感受态和培养温度。
1、质粒干粉(常温运输,存于-20度,90天保质期,请务必转化挑单克隆培养,不要直接使用和测序)
①收到质粒干粉后请先5000rpm离心1min,再加入20μl ddH2O去离子水溶解质粒;
②取1支100μl 感受态于冰上解冻10min,加入2μl质粒,再冰浴30min后,42℃热激60s,不要搅动,再冰浴2min;(从第二步开始均要在超净工作台中无菌操作)
③加入900μl无抗的LB液体培养基,180rpm震荡37℃培养45min;
④6000rpm离心5min,仅留100μl上清液重悬细菌沉淀,并涂布至目标质粒抗性的LB平板上;(可使用本平台的平板涂布专用玻璃珠进行涂布,可以比传统涂布方法获得更多转化子)
⑤将平板正向培养1h,再倒置37℃培养14h。如果要求是30度则培养20h;
(菌落过多则将质粒稀释后再转化。没有菌落则加入10μl质粒转化。另不要直接转表达感受态,要先转克隆感受态,重提质粒后再导入表达感受态)
⑥挑取单菌落至LB液体培养基中,加入对应抗生素,220rpm震荡培养14h,根据实验需要和质粒提取试剂盒说明书提取质粒。
2、甘油菌种(冰袋运输,存于-80℃,保质期90天,请务必划线挑单克隆培养)
四区划线后挑单菌落培养,酵母菌需要先液体复苏再四区划线,再挑单菌落液体培养。
3、穿刺菌种(冰袋运输,存于4℃,保质期7天)
穿刺接种,液体培养后四区划线,再挑单菌落液体培养。
4、菌落平板(冰袋运输,存于4℃,保质期7天)
直接挑取单菌落至液体培养基中。
5、液体质粒(冰袋运输,存于-20℃,保质期90天)
单独提取的液体质粒收到后可直接使用。
6、滤纸质粒(常温运输,存于-20度,90天保质期,请务必转化挑单克隆培养,不要直接使用和测序)
收到货后将滤纸画圈部分剪下放入EP管中,加100ul无菌水将滤纸浸湿并浸泡5min,吸取5ul质粒转化,离心全涂。
二、转化图片

| P2434/pSV40-Trpc4ap-m(892-3394bp)小鼠基因质粒 |
| P2435/pSV40-Icam5-m(1136-2754bp)小鼠基因质粒 |
| P2436/RN3P-KDM5B-FLAG小鼠基因质粒 |
| P2832/pGEM-CXXC5-m小鼠基因质粒 |
| P4843/pGEM-ucp1-A8G-m(1同义突变)小鼠基因质粒 |
| P3216/pCMV-RUNX2-m小鼠基因质粒 |
| P3429/pMD18-FGFR1-m(1同义突变)小鼠基因质粒 |
| P4932/pUC19-Pparg-m小鼠基因质粒 |
| P1063/pET26b-TNFα-m-His小鼠基因质粒 |
| P1064/pBV220-TNFα-m小鼠基因质粒 |
| P2437/pRSETB-ECFP-GABABR-m-Ypet小鼠基因质粒 |
| P0508/mATF4小鼠基因质粒 |
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验Amp pBBR1MCS-1 广宿主质粒 Cam pBBR1MCS-2 广宿主质粒 Kan http://www.ncbi.nlm.nih.gov/nuccore/U23751.1 pBBR1MCS-3 广宿主质粒
作为细胞生物学专业的博士生,在基因功能研究上已经有五年的经验了,今天我给大家讲讲其中经常用到并且非常重要的一种技术--慢病毒包装的过表达体系。下面我就从引物设计,慢病毒表达质粒构建等方面详细讲述。下面以 plex-MCS 载体为例。1. 选择酶切位点,为了避免移码突变,上游的酶切位点选择起始密码子 ATG 前面的 spel,下游选择 xho1。2. 构建方法的选择。连接法对于连接法,首先设计引物将目的基因 PCR 出来,即酶切位点加基因引物,此时应注意,为了防止酶切将基因破坏,通常习惯在两端
,TaKaRa,有木有;pGM-T,天根,有木有;一些表达载体,使用最广泛的,pET 系列,Novagen 公司(默克)的;有双 MCS 的 Duet 系列载体,Novagen 的;毕赤酵母表达质粒 pPIC3.5k,pPIC9k,pPICZA,pPICZaA 等等,都是 invitrogen 的;另外一些 pYES 酿酒酵母表达系统,也是 invitrogen 的,因此知道常见的质粒是哪个公司的,去他们公司网站上肯定可以找到该质粒的相关信息。在他们公司主页搜索栏直接搜索质粒名称,得到质粒序列,图谱什么的