相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 保存条件:
-20
- 保质期:
2年
- 英文名:
pCDNA6.2-GW/EmGFP-miR-Control
- 库存:
100
- 供应商:
上海烜雅生物科技有限公司
- 规格:
干粉/液体
名称:pCDNA6.2-GW/EmGFP-miR-Control哺乳调控质粒
别称: pCDNA6.2-GW/EmGFP-miR-Control
| 启动子: | CMV |
|---|---|
| 复制子: | pUC |
| 终止子: | HSV TK poly(A) signal |
| 质粒分类: | 哺乳细胞质粒;哺乳编辑质粒;干扰RNA质粒 |
| 质粒大小: | 5759bp |
| 原核抗性: | Str |
| 筛选标记: | Blast |
| 克隆菌株: | DH5a |
| 培养条件: | 37度 |
| 表达宿主: | 293T等哺乳细胞 |
| 培养条件: | 37℃,有氧,5%CO2 |
| 诱导方式: | 无需诱导 |
| 5'测序引物: | pEGFP-C-5 |
| 3'测序引物: | HSVtk-pA-R:GTCTCCTTCCGTGTTTCAG |
| 备注: | 可连入miRNA片段 |
质粒属性
| 载体宿主: | 哺乳细胞 |
|---|---|
| 载体用途: | 转录调控 |
| 基因种属: | |
| 基因类型: | miRNA |
| 原核抗性: | Spe |
| 真核抗性: | Blast |
| 荧光蛋白: | 绿色 |
质粒简介
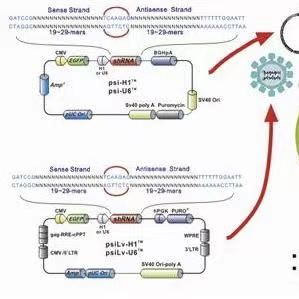
pCDNA6.2-GW-EmGFP-miR-Control质粒是在pcDNA6.2-GW/EmGFP-miR线性化载体的基础上连入了一个小片段而成。如果需要连入其它miRNA片段,可通过引物退火加酶切连接的方式连入。 转染后可立即表达 miRNA。 所表达的 miRNA 在核内经内源细胞机制(包括 Drosha 酶)加工后,转运至细胞质,由 Dicer 酶进一步加工)。 然后,完成加工的 miRNA 结合到 RISC,在此处其功能类似于 siRNA,使 mRNA 靶标被切割。
带有 EmGFP 的 BLOCK-iT™ Pol II miR RNAi 表达载体试剂盒将传统 RNAi 载体的优点(表达稳定并能够使用病毒导入)以及组织特异性表达和同一转录本多靶点抑制的功能集于一身。 带有 EmGFP 的 BLOCK-iT™ Pol II miR RNAi 表达载体试剂盒含有的 pcDNA6.2-GW/EmGFP-miR 载体设计用于表达人工 miRNA,该 miRNA 经工程化改造,与目的基因序列有 100% 的同源性并能引起靶标分子的切割。 该载体包括内源性 miRNA 的侧翼序列和环序列,内源性 miRNA 可指导从较长的 Pol II 转录本 (pre-miRNA) 上切除工程化的 miRNA。 使用 的 BLOCK-iT RNAi Designer,超过 70% 的重组体能产生超过 70% 的抑制效率。 工程化 miRNA 的 Pol II 表达可实现:
在 CMV 即刻早期启动子作用下高效表达,可通过 MultiSite Gateway重组选择组织特异性启动子和其它受调控的启动子;
在 pcDNA™6.2-GW/EmGFP-miR 载体中同-顺反子表达 Emerald GFP (EmGFP) 报告基因,使 EmGFP 的表达与目的 miRNA 抑制活性之间具有极强的相关性;
与用于基因表达的多种 Invitrogen Gateway® 目的 (DEST) 载体兼容;包括用于稳定转导分裂细胞、非分裂细胞和原代细胞类型的慢病毒载体,用于染色体单位点整合的 Flp-In™ 系统,以及替代报告基因融合构建体;
可在同一转录本表达的工程化 miRNA 多于一种,从而可同时抑制多个基因并产生合成表型;
质粒图谱
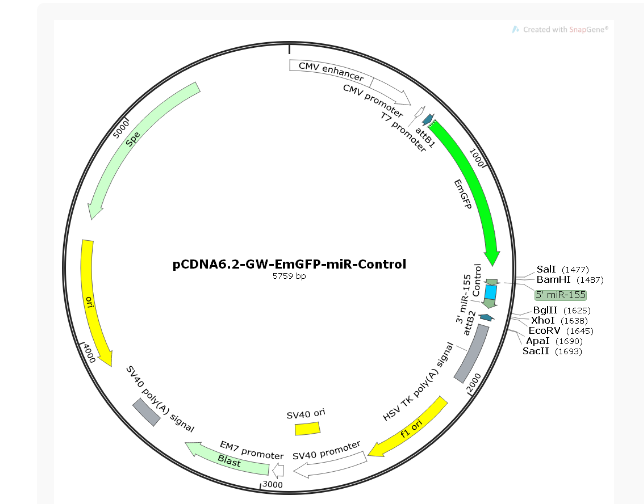

质粒序列
LOCUS Exported 5759 bp ds-DNA circular SYN 19-SEP-2017
DEFINITION synthetic circular DNA
KEYWORDS pCDNA6.2-GW-EmGFP-miR-Control
SOURCE synthetic DNA construct
ORGANISM synthetic DNA construct
REFERENCE 1 (bases 1 to 5759)
TITLE Direct Submission
JOURNAL Exported Sep 19, 2017 from MiaoLingPlasmid
http://www.miaolingbio.com
FEATURES Location/Qualifiers
source 1..5759
/organism="synthetic DNA construct"
/mol_type="other DNA"
enhancer 4..383
/label=CMV enhancer
/note="human cytomegalovirus immediate early enhancer"
promoter 384..587
/label=CMV promoter
/note="human cytomegalovirus (CMV) immediate early
promoter"
promoter 632..650
/label=T7 promoter
/note="promoter for bacteriophage T7 RNA polymerase"
protein_bind 680..704
/gene="mutant version of attB"
/label=attB1
/bound_moiety="BP Clonase(TM)"
/note="recombination site for the Gateway(R) BP reaction"
CDS 713..1432
/codon_start=1
/product="Emerald GFP"
/label=EmGFP
/note="mammalian codon-optimized"
/translation="MVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTL
KFICTTGKLPVPWPTLVTTFTYGVQCFARYPDHMKQHDFFKSAMPEGYVQERTIFFKDD
GNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHKVYITADKQKNGIK
VNFKTRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLL
EFVTAAGITLGMDELYK"
ncRNA 1492..1518
/label=5' miR-155
/note="sequence upstream of the precursor of mouse miR-155
microRNA (Uva et al., 2013)"
/ncRNA_class="miRNA"
misc_feature 1519..1581
/label=Control
ncRNA 1582..1623
/label=3' miR-155
/note="sequence downstream of the precursor of mouse
miR-155 microRNA (Uva et al., 2013)"
/ncRNA_class="miRNA"
protein_bind complement(1652..1676)
/gene="mutant version of attB"
/label=attB2
/bound_moiety="BP Clonase(TM)"
/note="recombination site for the Gateway(R) BP reaction"
polyA_signal 1705..1976
/label=HSV TK poly(A) signal
/note="herpes simplex virus thymidine kinase
polyadenylation signal (Cole and Stacy, 1985)"
rep_origin 2088..2516
/direction=RIGHT
/label=f1 ori
/note="f1 bacteriophage origin of replication; arrow
indicates direction of (+) strand synthesis"
promoter 2530..2859
/label=SV40 promoter
/note="SV40 enhancer and early promoter"
rep_origin 2710..2845
/label=SV40 ori
/note="SV40 origin of replication"
promoter 2907..2954
/label=EM7 promoter
/note="synthetic bacterial promoter "
CDS 2973..3371
/codon_start=1
/gene="Aspergillus terreus BSD"
/product="blasticidin S deaminase"
/label=Blast
/note="confers resistance to blasticidin"
/translation="MAKPLSQEESTLIERATATINSIPISEDYSVASAALSSDGRIFTG
VNVYHFTGGPCAELVVLGTAAAAAAGNLTCIVAIGNENRGILSPCGRCRQVLLDLHPGI
KAIVKDSDGQPTAVGIRELLPSGYVWEG"
polyA_signal 3529..3659
/label=SV40 poly(A) signal
/note="SV40 polyadenylation signal"
rep_origin complement(3856..4444)
/direction=LEFT
/label=ori
/note="high-copy-number ColE1/pMB1/pBR322/pUC origin of
replication"
CDS complement(4540..5331)
/codon_start=1
/gene="aadA"
/product="aminoglycoside adenylyltransferase (Murphy,
1985)"
/label=Spe
/note="confers resistance to spectinomycin and
streptomycin"
/translation="MREAVIAEVSTQLSEVVGVIERHLEPTLLAVHLYGSAVDGGLKPH
SDIDLLVTVTVRLDETTRRALINDLLETSASPGESEILRAVEVTIVVHDDIIPWRYPAK
RELQFGEWQRNDILAGIFEPATIDIDLAILLTKAREHSVALVGPAAEELFDPVPEQDLF
EALNETLTLWNSPPDWAGDERNVVLTLSRIWYSAVTGKIAPKDVAADWAMERLPAQYQP
VILEARQAYLGQEEDRLASRADQLEEFVHYVKGEITKVVGK"
ORIGIN
1 gttgacattg attattgact agttattaat agtaatcaat tacggggtca ttagttcata
61 gcccatatat ggagttccgc gttacataac ttacggtaaa tggcccgcct ggctgaccgc
121 ccaacgaccc ccgcccattg acgtcaataa tgacgtatgt tcccatagta acgccaatag
181 ggactttcca ttgacgtcaa tgggtggagt atttacggta aactgcccac ttggcagtac
241 atcaagtgta tcatatgcca agtacgcccc ctattgacgt caatgacggt aaatggcccg
301 cctggcatta tgcccagtac atgaccttat gggactttcc tacttggcag tacatctacg
361 tattagtcat cgctattacc atggtgatgc ggttttggca gtacatcaat gggcgtggat
421 agcggtttga ctcacgggga tttccaagtc tccaccccat tgacgtcaat gggagtttgt
481 tttggcacca aaatcaacgg gactttccaa aatgtcgtaa caactccgcc ccattgacgc
541 aaatgggcgg taggcgtgta cggtgggagg tctatataag cagagctctc tggctaacta
601 gagaacccac tgcttactgg cttatcgaaa ttaatacgac tcactatagg gagtcccaag
661 ctggctagtt aagctatcaa caagtttgta caaaaaagca ggctttaaaa ccatggtgag
721 caagggcgag gagctgttca ccggggtggt gcccatcctg gtcgagctgg acggcgacgt
781 aaacggccac aagttcagcg tgtccggcga gggcgagggc gatgccacct acggcaagct
841 gaccctgaag ttcatctgca ccaccggcaa gctgcccgtg ccctggccca ccctcgtgac
901 caccttcacc tacggcgtgc agtgcttcgc ccgctacccc gaccacatga agcagcacga
961 cttcttcaag tccgccatgc ccgaaggcta cgtccaggag cgcaccatct tcttcaagga
1021 cgacggcaac tacaagaccc gcgccgaggt gaagttcgag ggcgacaccc tggtgaaccg
1081 catcgagctg aagggcatcg acttcaagga ggacggcaac atcctggggc acaagctgga
1141 gtacaactac aacagccaca aggtctatat caccgccgac aagcagaaga acggcatcaa
1201 ggtgaacttc aagacccgcc acaacatcga ggacggcagc gtgcagctcg ccgaccacta
1261 ccagcagaac acccccatcg gcgacggccc cgtgctgctg cccgacaacc actacctgag
1321 cacccagtcc gccctgagca aagaccccaa cgagaagcgc gatcacatgg tcctgctgga
1381 gttcgtgacc gccgccggga tcactctcgg catggacgag ctgtacaagt aagctaagca
1441 cttcgtggcc gtcgatcgtt taaagggagg tagtgagtcg accagtggat cctggaggct
1501 tgctgaaggc tgtatgctgt gtcagtttgt caaatacccc gttttggcca ctgactgacg
1561 gggtattaca aactgacaca ggacacaagg cctgttacta gcactcacat ggaacaaatg
1621 gcccagatct ggccgcactc gagatatcta gacccagctt tcttgtacaa agtggttgat
1681 ctagagggcc cgcggttcgc tgatggggga ggctaactga aacacggaag gagacaatac
1741 cggaaggaac ccgcgctatg acggcaataa aaagacagaa taaaacgcac gggtgttggg
1801 tcgtttgttc ataaacgcgg ggttcggtcc cagggctggc actctgtcga taccccaccg
1861 tgaccccatt ggggccaata cgcccgcgtt tcttcctttt ccccacccca ccccccaagt
1921 tcgggtgaag gcccagggct cgcagccaac gtcggggcgg caggccctgc catagcatcc
1981 cctatagtga gtcgtattac atggtcatag ctgtttcctg gcagctctgg cccgtgtctc
2041 aaaatctctg atggatctgc gcagctgggg ctctaggggg tatccccacg cgccctgtag
2101 cggcgcatta agcgcggcgg gtgtggtggt tacgcgcagc gtgaccgcta cacttgccag
2161 cgccctagcg cccgctcctt tcgctttctt cccttccttt ctcgccacgt tcgccggctt
2221 tccccgtcaa gctctaaatc gggggctccc tttagggttc cgatttagtg ctttacggca
2281 cctcgacccc aaaaaacttg attagggtga tggttcacgt agtgggccat cgccctgata
2341 gacggttttt cgccctttga cgttggagtc cacgttcttt aatagtggac tcttgttcca
2401 aactggaaca acactcaacc ctatctcggt ctattctttt gatttataag ggattttgcc
2461 gatttcggcc tattggttaa aaaatgagct gatttaacaa aaatttaacg cgaattaatt
2521 ctgtggaatg tgtgtcagtt agggtgtgga aagtccccag gctccccagc aggcagaagt
2581 atgcaaagca tgcatctcaa ttagtcagca accaggtgtg gaaagtcccc aggctcccca
2641 gcaggcagaa gtatgcaaag catgcatctc aattagtcag caaccatagt cccgccccta
2701 actccgccca tcccgcccct aactccgccc agttccgccc attctccgcc ccatggctga
2761 ctaatttttt ttatttatgc agaggccgag gccgcctctg cctctgagct attccagaag
2821 tagtgaggag gcttttttgg aggcctaggc ttttgcaaaa agctcccggg agcttgtata
2881 tccattttcg gatctgatca gcacgtgttg acaattaatc atcggcatag tatatcggca
2941 tagtataata cgacaaggtg aggaactaaa ccatggccaa gcctttgtct caagaagaat
3001 ccaccctcat tgaaagagca acggctacaa tcaacagcat ccccatctct gaagactaca
3061 gcgtcgccag cgcagctctc tctagcgacg gccgcatctt cactggtgtc aatgtatatc
3121 attttactgg gggaccttgt gcagaactcg tggtgctggg cactgctgct gctgcggcag
3181 ctggcaacct gacttgtatc gtcgcgatcg gaaatgagaa caggggcatc ttgagcccct
3241 gcggacggtg ccgacaggtg cttctcgatc tgcatcctgg gatcaaagcc atagtgaagg
3301 acagtgatgg acagccgacg gcagttggga ttcgtgaatt gctgccctct ggttatgtgt
3361 gggagggcta agcacttcgt ggccgaggag caggactgac acgtgctacg agatttcgat
3421 tccaccgccg ccttctatga aaggttgggc ttcggaatcg ttttccggga cgccggctgg
3481 atgatcctcc agcgcgggga tctcatgctg gagttcttcg cccaccccaa cttgtttatt
3541 gcagcttata atggttacaa ataaagcaat agcatcacaa atttcacaaa taaagcattt
3601 ttttcactgc attctagttg tggtttgtcc aaactcatca atgtatctta tcatgtctgt
3661 ataccgtcgc tcttccgctg cttcctcgct cactgactcg ctgcgctcgg tcgttcggct
3721 gcggcgagcg gtatcagctc actcaaaggc ggtaatacgg ttatccacag aatcagggga
3781 taacgcagga aagaacatgt gagcaaaagg ccagcaaaag gccaggaacc gtaaaaaggc
3841 cgcgttgctg gcgtttttcc ataggctccg cccccctgac gagcatcaca aaaatcgacg
3901 ctcaagtcag aggtggcgaa acccgacagg actataaaga taccaggcgt ttccccctgg
3961 aagctccctc gtgcgctctc ctgttccgac cctgccgctt accggatacc tgtccgcctt
4021 tctcccttcg ggaagcgtgg cgctttctca tagctcacgc tgtaggtatc tcagttcggt
4081 gtaggtcgtt cgctccaagc tgggctgtgt gcacgaaccc cccgttcagc ccgaccgctg
4141 cgccttatcc ggtaactatc gtcttgagtc caacccggta agacacgact tatcgccact
4201 ggcagcagcc actggtaaca ggattagcag agcgaggtat gtaggcggtg ctacagagtt
4261 cttgaagtgg tggcctaact acggctacac tagaagaaca gtatttggta tctgcgctct
4321 gctgaagcca gttaccttcg gaaaaagagt tggtagctct tgatccggca aacaaaccac
4381 cgctggtagc ggtggttttt ttgtttgcaa gcagcagatt acgcgcagaa aaaaaggatc
4441 tcaagaagat cctttgatct tttctacggg gtctgacgct cagtggaacg acgcgtaact
4501 cacgttaagg gattttggtc atgggtggct cgacgagggt tatttgccga ctaccttggt
4561 gatctcgcct ttcacgtagt ggacaaattc ttccaactga tctgcgcgcg aggccaagcg
4621 atcttcttct tgtccaagat aagcctgtct agcttcaagt atgacgggct gatactgggc
4681 cggcaggcgc tccattgccc agtcggcagc gacatccttc ggcgcgattt tgccggttac
4741 tgcgctgtac caaatgcggg acaacgtaag cactacattt cgctcatcgc cagcccagtc
4801 gggcggcgag ttccatagcg ttaaggtttc atttagcgcc tcaaatagat cctgttcagg
4861 aaccggatca aagagttcct ccgccgctgg acctaccaag gcaacgctat gttctcttgc
4921 ttttgtcagc aagatagcca gatcaatgtc gatcgtggct ggctcgaaga tacctgcaag
4981 aatgtcattg cgctgccatt ctccaaattg cagttcgcgc ttagctggat aacgccacgg
5041 aatgatgtcg tcgtgcacaa caatggtgac ttctacagcg cggagaatct cgctctctcc
5101 aggggaagcc gaagtttcca aaaggtcgtt gatcaaagct cgccgcgttg tttcatcaag
5161 ccttacggtc accgtaacca gcaaatcaat atcactgtgt ggcttcaggc cgccatccac
5221 tgcggagccg tacaaatgta cggccagcaa cgtcggttcg agatggcgct cgatgacgcc
5281 aactacctct gatagttgag tcgatacttc ggcgatcacc gcttccctca taatgtttaa
5341 ctttgtttta gggcgactgc cctgctgcgt aacatcgttg ctgctccata acatcaaaca
5401 tcgacccacg gcgtaacgcg cttgctgctt ggatgcccga ggcatagact gtaccccaaa
5461 aaaacagtca taacaagcca tgaaaaccgc cactgcgccg ttaccaccgc tgcgttcggt
5521 caaggttctg gaccagttgc gtgagcgcat acgctacttg cattacagct tacgaaccga
5581 acaggcttat gtccactggg ttcgtgcctt catccgtttc cacggtgtgc gtcacccggc
5641 aaccttgggt agcagcgaag tcgaggcatt tctgtcctgg ctggtctaga attgcatgaa
5701 gaatctgctt agggttaggc gttttgcgct gcttcgcgat gtacgggcca gatatacgc
//
质粒菌株产品操作说明书
一、扩增流程
收到产品后,请先根据产品管壁标签来判断产品形式,并在扩增前准确查找该质粒菌株的抗性、感受态和培养温度。
1、质粒干粉(常温运输,存于-20度,90天保质期,请务必转化挑单克隆培养,不要直接使用和测序)
①收到质粒干粉后请先5000rpm离心1min,再加入20μl ddH2O去离子水溶解质粒;
②取1支100μl 感受态于冰上解冻10min,加入2μl质粒,再冰浴30min后,42℃热激60s,不要搅动,再冰浴2min;(从第二步开始均要在超净工作台中无菌操作)
③加入900μl无抗的LB液体培养基,180rpm震荡37℃培养45min;
④6000rpm离心5min,仅留100μl上清液重悬细菌沉淀,并涂布至目标质粒抗性的LB平板上;(可使用本平台的平板涂布专用玻璃珠进行涂布,可以比传统涂布方法获得更多转化子)
⑤将平板正向培养1h,再倒置37℃培养14h。如果要求是30度则培养20h;
(菌落过多则将质粒稀释后再转化。没有菌落则加入10μl质粒转化。另不要直接转表达感受态,要先转克隆感受态,重提质粒后再导入表达感受态)
⑥挑取单菌落至LB液体培养基中,加入对应抗生素,220rpm震荡培养14h,根据实验需要和质粒提取试剂盒说明书提取质粒。
2、甘油菌种(冰袋运输,存于-80℃,保质期90天,请务必划线挑单克隆培养)
四区划线后挑单菌落培养,酵母菌需要先液体复苏再四区划线,再挑单菌落液体培养。
3、穿刺菌种(冰袋运输,存于4℃,保质期7天)
穿刺接种,液体培养后四区划线,再挑单菌落液体培养。
4、菌落平板(冰袋运输,存于4℃,保质期7天)
直接挑取单菌落至液体培养基中。
5、液体质粒(冰袋运输,存于-20℃,保质期90天)
单独提取的液体质粒收到后可直接使用。
6、滤纸质粒(常温运输,存于-20度,90天保质期,请务必转化挑单克隆培养,不要直接使用和测序)
收到货后将滤纸画圈部分剪下放入EP管中,加100ul无菌水将滤纸浸湿并浸泡5min,吸取5ul质粒转化,离心全涂。
二、转化图片

| P4664/pCMV-SPORT6-PBX3人源基因质粒 |
| P4665/pCMV-SPORT6-PFKFB4人源基因质粒 |
| P4666/pCMV-SPORT6-WDR41人源基因质粒 |
| P4667/pCMV-SPORT6-TAPBP(1点突变)人源基因质粒 |
| P4668/pCMV-SPORT6-ST3GAL4人源基因质粒 |
| P4669/pCMV-SPORT6-IMPDH2人源基因质粒 |
| P4670/pCMV-SPORT6-AK5人源基因质粒 |
| P4671/pCMV-SPORT6-IQSEC1人源基因质粒 |
| P4672/pCMV-SPORT6-NET1人源基因质粒 |
| P4673/pCMV-SPORT6-IRF6人源基因质粒 |
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验本人持有以下载体及基因:(原则上只交换不赠送,欢迎广大站友互通有无) QQ 2283524599 luckamily@126.com 一、载体 1)、miRNA载体 Promega miRNA靶标克隆载体 psiCHECK-2 psiCHECK2 amp Ambion miRNA靶标克隆载体 pmiR-report pmiRreport amp Invitrogen miRNA过表达载体 PCDNA TM 6.2-GW/EmGFP-miR
2)。 图2 使用BLOCK-iT™ Complete Dicer RNAi Kit得到>80%的靶定基因阻断 经Dicer酶切的,来源于β-半乳糖苷酶或者荧光素酶的dsRNA产生siRNA(d-siRNA)库,用来确定d-siRNA特异阻断基因活性的效果。GripTite™ 293 MSR 细胞共转染pcDNA™1.2/V5-GW/lacZ阳性对照质粒和pcDNA™5/FRT/luc,分别独自表达β-gal或者荧光素酶报告子,或者50ng luc 或者lacZ d-siRNA。对于每一种报告子
pmCherry-C1 哺乳细胞质粒 Amp http://www.yrgene.com/product/vector/15552 pCDNA3 哺乳细胞质粒 Amp pCDNA3-HA 哺乳细胞质粒 Amp