Olink Proteomics品牌商
1 年
手机商铺
- NaN
- 0
- 1
- 2
- 1
推荐产品
公司新闻/正文
Stabl 机器学习助力组学标志物优中选优(Nature子刊好文回顾)
80 人阅读发布时间:2025-10-09 11:00
研究背景
高通量组学技术结合机器学习算法已经被广泛应用于临床研究,并产出了大量的候选生物标志物。然而,将候选标志物转化为临床应用的生物标志物依然具有挑战。
在生物标志物发现研究的统计学分析中,临床转化往往包含三个必需的任务:
(1)通过识别具有高预测性能(预测性)的多变量模型来预测临床终点;
(2)选择有限数量的特征作为候选临床生物标志物(稀疏性);
(3)确保所选特征与结果真实相关的置信度(可靠性)。
目前,多种机器学习方法可以提供适应p ≫ n多组学数据集的预测模型架构。数据融合方法,如LASSO等也可以集成多种数据集。然而,由于学习阶段通常依赖于有限数量的样本,训练数据中的小扰动可能会导致具有广泛差异的选定特征集,从而破坏与相关性的置信度。因此需要开发能够选择出稀疏而可靠的候选生物标志物的方法。

斯坦福大学的研究人员在Nature Biotechnology(IF=46.9)上发表了一项研究,开发了一种强大的机器学习算法Stabl。Stabl是一种有监督的机器学习框架,通过将噪声注入和数据驱动的信噪比阈值集成到多变量预测建模中,来识别一组稀疏,可靠的生物标志物集。
研究人员在合成数据集和五项独立的临床研究上评估了Stabl的功能,结果表明,Stabl可提高生物标志物的稀疏性和可靠性,并同时保持预测性能。使用Stabl能够将包含1,400-35,000个特征的数据集提炼为4-34个候选生物标志物。将Stabl扩展到多组学整合任务中,能够对复杂的预测模型进行生物学解释。
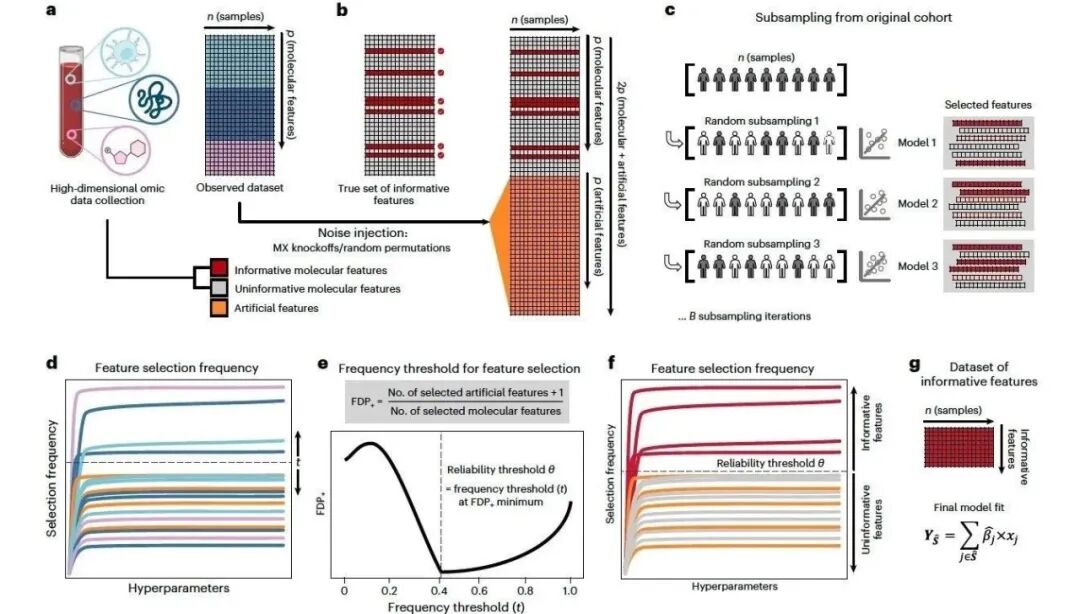
Stabl算法概述
a,通过测量n个样本中每个样本的p个分子特征,得到大小为n × p的原始数据集。
b,在观察到的特征中,一些是有信息的(与结果相关,红色),另一些是无信息的(与结果无关,灰色)。将P个人工特征(橙色)注入到原始数据集中,得到一个大小为n × 2p的新数据集。人工特征是使用MX仿制品或随机排列构建的。从大小为n的原始队列中进行.
c,B次子样本迭代。在每次迭代k时,正则化参数(s) λ变化的SRM模型被拟合到子样本上,导致每次迭代所选择的特征集不同。
d,对于给定的λ,总共生成B组选定的特征。特征i所在集合的比例定义特征选择频率fi(λ)。绘制fi(λ)与1/λ的关系得到一个稳定路径图。在最终模型中选择最大频率大于频率阈值(t)的特征。
f&g,选择频率大于θ的特征集(即可靠特征)包含在最终的预测模型中。
研究人员将Stabl SRM应用于两个单组学临床数据集。数据集一包含大规模血浆cfRNA(37,184个特征),旨在将妊娠分为血压正常或先兆子痫(PE)(下图a,b)。数据集二涉及高复合血浆蛋白质组学(由Olink Explore 1536测定的1,463个特征),旨在对SARS-CoV-2阳性患者的两个独立队列(训练队列和验证队列)进行COVID-19严重程度分类(下图c,d)。
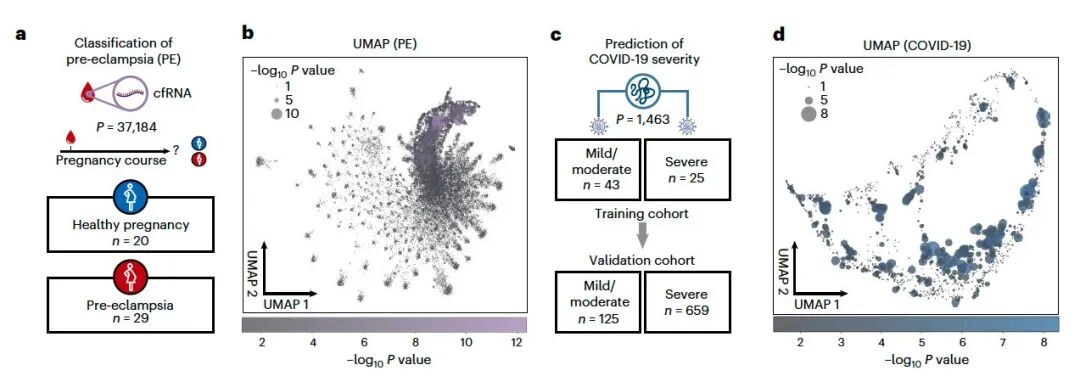
尽管LASSO和EN模型在这些案例中都得到了非常好的预测性能(AUROC = 0.74-0.84),这表明它们具有强大的生物标志物诊断潜力,但模型稀疏度和可靠性的缺乏阻碍了候选生物标志物的识别,需要额外的特征选择方法,这些方法与预测建模过程相分离。
与使用合成数据获得的结果一致,StablL、StablEN和StablAL分别比LASSO、EN和AL表现出更高的稀疏性(下图e、f)。对于PE数据集,Stabl SRM选择的特征比LASSO或EN少20倍以上,比AL少8倍(下图e)。
对于COVID-19分类,Stabl SRM对LASSO、EN和AL分别以1.9、>20和1.25的因子减少特征数量(下图f)。值得注意的是,StablL, StablEN和StablAL在两个数据集上保持了与各自SRM相似的预测性能(下图h)。
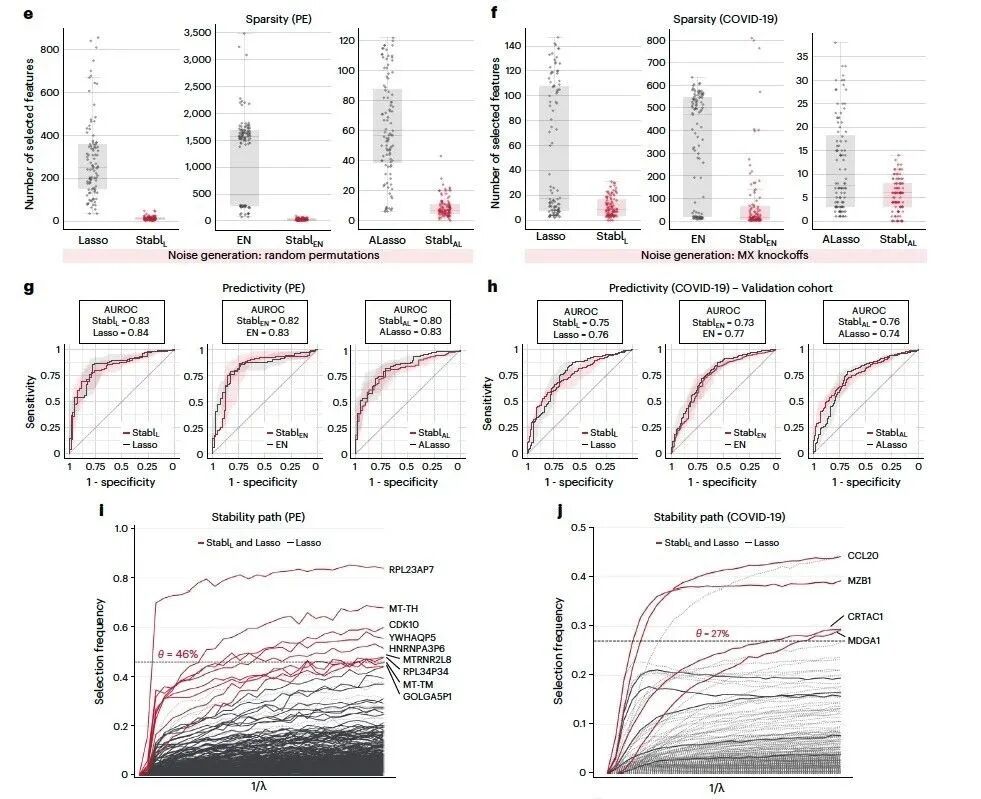
对于COVID-19数据集,StablL确定了与宿主炎症反应的关键病理生物学机制相呼应的特征,例如CCL20,这是COVID-19细胞因子风暴的已知元素;新发现的肺功能标志物CRTAC1,是一种与COVID-19感染后高中和抗体滴度相关的蛋白质(上图j)。StablL模型还选择了MDGA1,这是一种以前未知的COVID-19严重程度的候选生物标志物。
写在最后
Stabl 是一个机器学习框架,旨在促进高维组学生物标志物研究的临床转化。Stabl可以在多变量预测建模架构中实现数据驱动的稀疏和可靠的生物标志物候选选择。当应用于跨越不同组学技术、单组学和多组学数据集以及临床终点的现实世界生物标志物发现任务时,Stabl 始终显示出其在可靠选择生物可解释生物标志物候选物方面的适应性和有效性,有助于进一步的临床转化。
参考文献: