相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 品系:
详见细胞说明资料
- 细胞类型:
详见细胞说明资料
- 肿瘤类型:
详见细胞说明资料
- 供应商:
上海冠导生物工程有限公司
- 库存:
≥100瓶
- 生长状态:
详见细胞说明资料
- 年限:
详见细胞说明资料
- 运输方式:
常温运输【复苏细胞】或干冰运输【冻存细胞】
- 器官来源:
详见细胞说明资料
- 是否是肿瘤细胞:
详见细胞说明资料
- 细胞形态:
详见细胞说明资料
- 免疫类型:
详见细胞说明资料
- 物种来源:
详见细胞说明资料
- 相关疾病:
详见细胞说明资料
- 组织来源:
详见细胞说明资料
- 英文名:
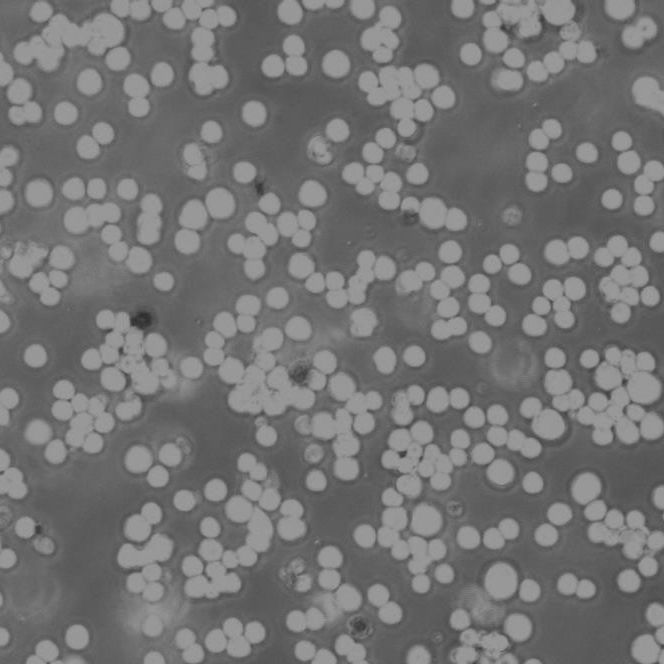
KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱
- 规格:
1*10(6)Cellls/瓶
"KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱
物种来源:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱来源于人源、鼠源等其它物种来源
生长特性:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱贴壁生长
形态特性:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱上皮细胞样
实验人员需佩戴防爆面罩及双层无菌手套,尤其当发现冻存管液氮渗入时(管壁出现冰晶凝结为典型标志)。建议使用专门设计的液氮罐提取器,避免徒手接触-196℃的冻存管导致低温灼伤。解冻前准备37℃恒温水浴锅,水位需完全浸没冻存管液面线以上,并预热至温度稳定(±0.5℃波动范围)。
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。
换液周期:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱每周2-3次
产品包装:复苏发货:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
背景信息:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱详见相关文献介绍
在生命科学研究的微观世界里,细胞培养技术犹如搭建了一座精密运转的舞台,而细胞消化过程则是这台大戏中至关重要的场景转换环节。当科研人员需要为细胞进行传代培养、开展单细胞分离或执行精确计数时,胰蛋白酶便化身成为舞台幕后的魔法师,用其精妙的生物化学机制完成这场细胞集体的""温柔离散""。
细胞培养瓶破裂或渗漏是实验室最紧急的情况之一。当发现容器破损时,应立即执行三级处理流程:首先将破损培养瓶放入生物安全柜,用消毒剂浸泡的纱布覆盖裂口;随后将培养物转移至备用培养瓶,转移过程需严格保证无菌操作;最后对工作台面进行彻底消毒。值得注意的是,聚乙烯材质的培养瓶在反复冻融后更易产生微裂纹,建议使用前进行透光检查。对于珍贵细胞系,可预先分装冻存备份,并建立定期镜检制度,每周至少两次检查培养容器完整性。
传代比例:1:2-1:4(KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱首次传代建议1:2)
来源说明:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱主要来源ATCC、ECACC、DSMZ、RCB等细胞库
CO₂细胞培养箱作为细胞培养的核心设备,通过精确控制温度(通常37℃)、湿度(95%左右)和CO₂浓度(5%),模拟体内生理环境。现代培养箱采用HEPA过滤系统实现空气净化,部分型号配备紫外灯或铜质内胆增强抑菌能力。值得注意的是,有些新型智能CO₂培养箱已实现远程监控,通过传感器实时反馈箱内O₂含量和pH值变化,大幅降低污染风险。使用时需定期校准气体浓度传感器,并避免频繁开门导致环境波动。
准确判断胰酶消化是否完成,首先,观察细胞形态变化是判断胰酶消化程度的重要依据。在加入胰酶后,随着消化的进行,原本贴壁生长的细胞会逐渐变圆。当大部分细胞呈现出圆形,且细胞之间的连接变得松散时,说明消化正在进行中。如果细胞完全脱离培养瓶底部,变成单个的圆形细胞悬浮在消化液中,这通常是消化完成的一个重要标志。此时,在显微镜下观察,细胞应该是圆润、饱满且边界清晰的。其次,可以通过轻轻拍打培养瓶或轻轻晃动培养板来辅助判断。如果细胞很容易从瓶底或板底脱落并在液体中悬浮,说明消化程度可能已经比较合适。但要注意动作要轻柔,避免过度剧烈的晃动对细胞造成损伤。
Hs888Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:COLO 829细胞、Y79细胞、SUM52PE细胞
OE21 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCI-H508细胞、ECV304细胞、H64细胞
N-Tera-2 Cells;背景说明:畸胎瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RBL-2H3细胞、IEC 18细胞、PC-3M IE8细胞
HRIF Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CV-1细胞、Madin-Darby Bovine Kidney细胞、MCF-10A细胞
D562 Cells;背景说明:器官:咽头 疾病:癌 取材转移灶:胸水;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:MIA-PaCa-2细胞、HFT8810细胞、SHSY5Y细胞
CD-18 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:B16 melanoma F10细胞、NCIH1573细胞、RAEC细胞
KU-812-F Cells;背景说明:慢性粒细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SEM细胞、Mevo细胞、OCIAML2细胞
HD11 Cells鸡巨噬细胞库专业复苏|带STR图谱
LX-2-GFP Cells人肝星状细胞库专业复苏|带STR图谱
A-253 Cells人颌下腺表皮癌细胞库专业复苏|带STR图谱
HT-29-Luc Cells人结肠癌细胞库专业复苏|带STR图谱
KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱
CX-1 Cells人结肠癌细胞库专业复苏|带STR图谱
THLE-2 Cells人肝细胞库专业复苏|带STR图谱
SiHa Cells人子宫颈鳞癌细胞库专业复苏|带STR图谱
Hep 3B2.1-7 Cells人肝癌细胞库专业复苏|带STR图谱
HFL1 Cells人胚肺成纤维细胞库专业复苏|带STR图谱
NCIH2106 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:GM03190细胞、RCF细胞、MN 60细胞
Ba/F3 KIF5B-RET Cells(提供STR鉴定图谱)
Z138 Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:15传代;每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:NCI-H520细胞、HeLa 229细胞、KATOIII细胞
UMNSAH-DF1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维母细胞样;相关产品有:PC10细胞、BE(2)-M17细胞、CAL-12T细胞
RCC10 RGB Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC1187细胞、HEC-1B细胞、Rat Skin 1细胞
SUDHL6 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,3—4天换液1次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:H841细胞、RAOEC细胞、ATN-1细胞
AGS Cells人胃腺癌细胞库专业复苏|带STR图谱
L2 Cells大鼠肺上皮细胞库专业复苏|带STR图谱
KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱
PT-K75 Cells猪鼻甲黏膜成纤维细胞库专业复苏|带STR图谱
A375-RFP Cells人黑色素瘤细胞库专业复苏|带STR图谱
Y79 Cells人视网膜母细胞瘤细胞库专业复苏|带STR图谱
SW626 Cells人卵巢癌细胞库专业复苏|带STR图谱
K7M2wt+luc Cells小鼠骨肉瘤成骨细胞库专业复苏|带STR图谱
NCI-H69 Cells人小细胞肺癌细胞库专业复苏|带STR图谱
CEM/C1 Cells人急性淋巴细胞白血病细胞库专业复苏|带STR图谱
639-V Cells;背景说明:膀胱癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PA-TU-8988S细胞、CAL27细胞、NCI-H1435细胞
U-2 OS Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CL 1-0细胞、HSAS3细胞、MMQ细胞
MDAMB175 Cells;背景说明:该细胞源自一位54岁患有乳腺导管癌白人女性的胸腔积液。;传代方法:1:2—1:6传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:FCCH1018细胞、Reuber H-35细胞、H292细胞
MPC5 Cells;背景说明:肾足细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:COLO-HSR细胞、CCRF.CEM细胞、H-2052细胞
GM12410 Cells(提供STR鉴定图谱)
HAP1 ELK1 (-) 1 Cells(提供STR鉴定图谱)
GC-1spg Cells小鼠精原细胞库专业复苏|带STR图谱
OK Cells负鼠肾细胞库专业复苏|带STR图谱
AtT-20 Cells小鼠垂体瘤细胞库专业复苏|带STR图谱
PC-12-GFP Cells大鼠肾上腺嗜铬细胞瘤细胞库专业复苏|带STR图谱
OV-1063 Cells人卵巢上皮细胞癌细胞库专业复苏|带STR图谱
RWPE-2 Cells人前列腺正常细胞库专业复苏|带STR图谱
HKb20 Cells人肾上皮细胞库专业复苏|带STR图谱
LLC-MK2 Cells猴肾细胞库专业复苏|带STR图谱
NCI-N87 Cells人胃癌细胞库专业复苏|带STR图谱
HCT 116 Cells;背景说明:结肠腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H1694细胞、MOLT-16细胞、Metastatic Variant-522细胞
HepG2-luc Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SKNO1细胞、TE 671细胞、NRK clone 49F细胞
IEC-18 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:McA-RH7777细胞、CTV1细胞、Kit 225细胞
Huh-7.5 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEK 293-H细胞、NCI-H510细胞、MA104细胞
IB-RS2 Cells;背景说明:肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW-1417细胞、MKN-45细胞、OUMS-23细胞
HeLa.S3 Cells;背景说明:该细胞是1955年由PuckTT,MarcusPI和CieciuraSJ建系的,含HPV-18序列;角蛋白阳性;可用于与染色体突变、细胞营养、集落形成相关的哺乳动物细胞的克隆分析。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:ND7/23细胞、Ocular Choroidal Melanoma-1细胞、BT474细胞
NCI-H1734 Cells人非小细胞肺癌细胞库专业复苏|带STR图谱
Toledo Cells人弥漫大B细胞淋巴瘤细胞库专业复苏|带STR图谱
P3X63Ag8.653 Cells小鼠骨髓瘤细胞库专业复苏|带STR图谱
MADB106 Cells大鼠乳腺癌细胞库专业复苏|带STR图谱
KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱
NCI-H1734 Cells人非小细胞肺癌细胞库专业复苏|带STR图谱
GA-10-Clone-4 Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:MGH-U3细胞、NCIH2135细胞、SKLMS-1细胞
H3396 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1693细胞、KYSE 410细胞、WSU-DLCL(2)细胞
HIO107C6 Cells(提供STR鉴定图谱)
IPLB-LdFB clone 4 Cells(提供STR鉴定图谱)
mlMCD-3 Cells小鼠肾髓质细胞库专业复苏|带STR图谱
NCI-H446 Cells人肺癌细胞库专业复苏|带STR图谱
P3X63Ag8 Cells小鼠骨髓瘤细胞库专业复苏|带STR图谱
MG-63 Cells人骨肉瘤细胞库专业复苏|带STR图谱
HEK293E Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:10传代;每周2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:RPMI-7951细胞、H920细胞、Tn-5B1-4细胞
SUM 102 Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Swiss-3T3细胞、ID8/MOSEC细胞、NFS60细胞
KYSE30 Cells;背景说明:来源于一位64岁,患有高分化的中段食管鳞癌的男性患者。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:MCF7/WT细胞、Lymph Node Carcinoma of the prostate细胞、HCA7细胞
HAP1 YAP1 (-) 1 Cells(提供STR鉴定图谱)
HPDEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COLO_320DM细胞、Jurkat Clone E6-1细胞、HRGEC细胞
RPE D407 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:WM115F细胞、MGHU3细胞、SW-480细胞
HSKMC Cells;背景说明:骨骼肌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:C8166-CD4细胞、MD Anderson-Metastatic Breast-157细胞、SIRC细胞
L6 Cells;背景说明:该细胞是Yaffe在甲基胆蒽存在的情况下从大鼠大腿肌原代培养的最初两代细胞中分离得到的;在培养基中融合形成多核的肌管和横纹肌纤维,细胞融合的程度随着代数的增加而下降,因此细胞应低代次冷冻并周期性地重新克隆以选择融合能力强的细胞。鼠痘病毒阴性。该细胞应在达汇合状态前传代,以延缓细胞分化能力的丧失。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成肌细胞样;相关产品有:SKCO 1细胞、DU4475细胞、Michigan Cancer Foundation-12F细胞
SK-GT-4 Cells人食道癌肿瘤细胞库专业复苏|带STR图谱
HeLa-RFP Cells人宫颈癌细胞库专业复苏|带STR图谱
HPAC Cells人胰腺腺泡上皮癌细胞库专业复苏|带STR图谱
KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱
LS180 Cells人结肠癌细胞库专业复苏|带STR图谱
GBC-SD Cells人胆囊癌细胞库专业复苏|带STR图谱
RSC96 Cells大鼠雪旺细胞库专业复苏|带STR图谱
MasterShef10 Cells(提供STR鉴定图谱)
ND09990 Cells(提供STR鉴定图谱)
PJS5JTO Cells(提供STR鉴定图谱)
TR-LCC-1 Cells(提供STR鉴定图谱)
UIV-SL-373 Cells(提供STR鉴定图谱)
" "PubMed=9033652; DOI=10.1002/(SICI)1097-0215(19970207)70:4<437::AID-IJC11>3.0.CO;2-C
Tanaka H., Shimada Y., Imamura M., Shibagaki I., Ishizaki K.
Multiple types of aberrations in the p16 (INK4a) and the p15(INK4b) genes in 30 esophageal squamous-cell-carcinoma cell lines.
Int. J. Cancer 70:437-442(1997)
PubMed=11092977; DOI=10.1111/j.1349-7006.2000.tb00895.x; PMCID=PMC5926289
Pimkhaokham A., Shimada Y., Fukuda Y., Kurihara N., Imoto I., Yang Z.-Q., Imamura M., Nakamura Y., Amagasa T., Inazawa J.
Nonrandom chromosomal imbalances in esophageal squamous cell carcinoma cell lines: possible involvement of the ATF3 and CENPF genes in the 1q32 amplicon.
Jpn. J. Cancer Res. 91:1126-1133(2000)
PubMed=15172977; DOI=10.1158/0008-5472.CAN-04-0172
Sonoda I., Imoto I., Inoue J., Shibata T., Shimada Y., Chin K., Imamura M., Amagasa T., Gray J.W., Hirohashi S., Inazawa J.
Frequent silencing of low density lipoprotein receptor-related protein 1B (LRP1B) expression by genetic and epigenetic mechanisms in esophageal squamous cell carcinoma.
Cancer Res. 64:3741-3747(2004)
PubMed=16045545; DOI=10.1111/j.0959-9673.2005.00431.x; PMCID=PMC2517430
Ban S., Michikawa Y., Ishikawa K.-i., Sagara M., Watanabe K., Shimada Y., Inazawa J., Imai T.
Radiation sensitivities of 31 human oesophageal squamous cell carcinoma cell lines.
Int. J. Exp. Pathol. 86:231-240(2005)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"
物种来源:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱来源于人源、鼠源等其它物种来源
生长特性:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱贴壁生长
形态特性:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱上皮细胞样
实验人员需佩戴防爆面罩及双层无菌手套,尤其当发现冻存管液氮渗入时(管壁出现冰晶凝结为典型标志)。建议使用专门设计的液氮罐提取器,避免徒手接触-196℃的冻存管导致低温灼伤。解冻前准备37℃恒温水浴锅,水位需完全浸没冻存管液面线以上,并预热至温度稳定(±0.5℃波动范围)。
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。
换液周期:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱每周2-3次
产品包装:复苏发货:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
背景信息:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱详见相关文献介绍
在生命科学研究的微观世界里,细胞培养技术犹如搭建了一座精密运转的舞台,而细胞消化过程则是这台大戏中至关重要的场景转换环节。当科研人员需要为细胞进行传代培养、开展单细胞分离或执行精确计数时,胰蛋白酶便化身成为舞台幕后的魔法师,用其精妙的生物化学机制完成这场细胞集体的""温柔离散""。
细胞培养瓶破裂或渗漏是实验室最紧急的情况之一。当发现容器破损时,应立即执行三级处理流程:首先将破损培养瓶放入生物安全柜,用消毒剂浸泡的纱布覆盖裂口;随后将培养物转移至备用培养瓶,转移过程需严格保证无菌操作;最后对工作台面进行彻底消毒。值得注意的是,聚乙烯材质的培养瓶在反复冻融后更易产生微裂纹,建议使用前进行透光检查。对于珍贵细胞系,可预先分装冻存备份,并建立定期镜检制度,每周至少两次检查培养容器完整性。
传代比例:1:2-1:4(KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱首次传代建议1:2)
来源说明:KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱主要来源ATCC、ECACC、DSMZ、RCB等细胞库
CO₂细胞培养箱作为细胞培养的核心设备,通过精确控制温度(通常37℃)、湿度(95%左右)和CO₂浓度(5%),模拟体内生理环境。现代培养箱采用HEPA过滤系统实现空气净化,部分型号配备紫外灯或铜质内胆增强抑菌能力。值得注意的是,有些新型智能CO₂培养箱已实现远程监控,通过传感器实时反馈箱内O₂含量和pH值变化,大幅降低污染风险。使用时需定期校准气体浓度传感器,并避免频繁开门导致环境波动。
准确判断胰酶消化是否完成,首先,观察细胞形态变化是判断胰酶消化程度的重要依据。在加入胰酶后,随着消化的进行,原本贴壁生长的细胞会逐渐变圆。当大部分细胞呈现出圆形,且细胞之间的连接变得松散时,说明消化正在进行中。如果细胞完全脱离培养瓶底部,变成单个的圆形细胞悬浮在消化液中,这通常是消化完成的一个重要标志。此时,在显微镜下观察,细胞应该是圆润、饱满且边界清晰的。其次,可以通过轻轻拍打培养瓶或轻轻晃动培养板来辅助判断。如果细胞很容易从瓶底或板底脱落并在液体中悬浮,说明消化程度可能已经比较合适。但要注意动作要轻柔,避免过度剧烈的晃动对细胞造成损伤。
Hs888Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:COLO 829细胞、Y79细胞、SUM52PE细胞
OE21 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCI-H508细胞、ECV304细胞、H64细胞
N-Tera-2 Cells;背景说明:畸胎瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RBL-2H3细胞、IEC 18细胞、PC-3M IE8细胞
HRIF Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CV-1细胞、Madin-Darby Bovine Kidney细胞、MCF-10A细胞
D562 Cells;背景说明:器官:咽头 疾病:癌 取材转移灶:胸水;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:MIA-PaCa-2细胞、HFT8810细胞、SHSY5Y细胞
CD-18 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:B16 melanoma F10细胞、NCIH1573细胞、RAEC细胞
KU-812-F Cells;背景说明:慢性粒细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SEM细胞、Mevo细胞、OCIAML2细胞
HD11 Cells鸡巨噬细胞库专业复苏|带STR图谱
LX-2-GFP Cells人肝星状细胞库专业复苏|带STR图谱
A-253 Cells人颌下腺表皮癌细胞库专业复苏|带STR图谱
HT-29-Luc Cells人结肠癌细胞库专业复苏|带STR图谱
KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱
CX-1 Cells人结肠癌细胞库专业复苏|带STR图谱
THLE-2 Cells人肝细胞库专业复苏|带STR图谱
SiHa Cells人子宫颈鳞癌细胞库专业复苏|带STR图谱
Hep 3B2.1-7 Cells人肝癌细胞库专业复苏|带STR图谱
HFL1 Cells人胚肺成纤维细胞库专业复苏|带STR图谱
NCIH2106 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:GM03190细胞、RCF细胞、MN 60细胞
Ba/F3 KIF5B-RET Cells(提供STR鉴定图谱)
Z138 Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:15传代;每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:NCI-H520细胞、HeLa 229细胞、KATOIII细胞
UMNSAH-DF1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维母细胞样;相关产品有:PC10细胞、BE(2)-M17细胞、CAL-12T细胞
RCC10 RGB Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC1187细胞、HEC-1B细胞、Rat Skin 1细胞
SUDHL6 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,3—4天换液1次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:H841细胞、RAOEC细胞、ATN-1细胞
AGS Cells人胃腺癌细胞库专业复苏|带STR图谱
L2 Cells大鼠肺上皮细胞库专业复苏|带STR图谱
KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱
PT-K75 Cells猪鼻甲黏膜成纤维细胞库专业复苏|带STR图谱
A375-RFP Cells人黑色素瘤细胞库专业复苏|带STR图谱
Y79 Cells人视网膜母细胞瘤细胞库专业复苏|带STR图谱
SW626 Cells人卵巢癌细胞库专业复苏|带STR图谱
K7M2wt+luc Cells小鼠骨肉瘤成骨细胞库专业复苏|带STR图谱
NCI-H69 Cells人小细胞肺癌细胞库专业复苏|带STR图谱
CEM/C1 Cells人急性淋巴细胞白血病细胞库专业复苏|带STR图谱
639-V Cells;背景说明:膀胱癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PA-TU-8988S细胞、CAL27细胞、NCI-H1435细胞
U-2 OS Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CL 1-0细胞、HSAS3细胞、MMQ细胞
MDAMB175 Cells;背景说明:该细胞源自一位54岁患有乳腺导管癌白人女性的胸腔积液。;传代方法:1:2—1:6传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:FCCH1018细胞、Reuber H-35细胞、H292细胞
MPC5 Cells;背景说明:肾足细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:COLO-HSR细胞、CCRF.CEM细胞、H-2052细胞
GM12410 Cells(提供STR鉴定图谱)
HAP1 ELK1 (-) 1 Cells(提供STR鉴定图谱)
GC-1spg Cells小鼠精原细胞库专业复苏|带STR图谱
OK Cells负鼠肾细胞库专业复苏|带STR图谱
AtT-20 Cells小鼠垂体瘤细胞库专业复苏|带STR图谱
PC-12-GFP Cells大鼠肾上腺嗜铬细胞瘤细胞库专业复苏|带STR图谱
OV-1063 Cells人卵巢上皮细胞癌细胞库专业复苏|带STR图谱
RWPE-2 Cells人前列腺正常细胞库专业复苏|带STR图谱
HKb20 Cells人肾上皮细胞库专业复苏|带STR图谱
LLC-MK2 Cells猴肾细胞库专业复苏|带STR图谱
NCI-N87 Cells人胃癌细胞库专业复苏|带STR图谱
HCT 116 Cells;背景说明:结肠腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H1694细胞、MOLT-16细胞、Metastatic Variant-522细胞
HepG2-luc Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SKNO1细胞、TE 671细胞、NRK clone 49F细胞
IEC-18 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:McA-RH7777细胞、CTV1细胞、Kit 225细胞
Huh-7.5 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEK 293-H细胞、NCI-H510细胞、MA104细胞
IB-RS2 Cells;背景说明:肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW-1417细胞、MKN-45细胞、OUMS-23细胞
HeLa.S3 Cells;背景说明:该细胞是1955年由PuckTT,MarcusPI和CieciuraSJ建系的,含HPV-18序列;角蛋白阳性;可用于与染色体突变、细胞营养、集落形成相关的哺乳动物细胞的克隆分析。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:ND7/23细胞、Ocular Choroidal Melanoma-1细胞、BT474细胞
NCI-H1734 Cells人非小细胞肺癌细胞库专业复苏|带STR图谱
Toledo Cells人弥漫大B细胞淋巴瘤细胞库专业复苏|带STR图谱
P3X63Ag8.653 Cells小鼠骨髓瘤细胞库专业复苏|带STR图谱
MADB106 Cells大鼠乳腺癌细胞库专业复苏|带STR图谱
KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱
NCI-H1734 Cells人非小细胞肺癌细胞库专业复苏|带STR图谱
GA-10-Clone-4 Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:MGH-U3细胞、NCIH2135细胞、SKLMS-1细胞
H3396 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1693细胞、KYSE 410细胞、WSU-DLCL(2)细胞
HIO107C6 Cells(提供STR鉴定图谱)
IPLB-LdFB clone 4 Cells(提供STR鉴定图谱)
mlMCD-3 Cells小鼠肾髓质细胞库专业复苏|带STR图谱
NCI-H446 Cells人肺癌细胞库专业复苏|带STR图谱
P3X63Ag8 Cells小鼠骨髓瘤细胞库专业复苏|带STR图谱
MG-63 Cells人骨肉瘤细胞库专业复苏|带STR图谱
HEK293E Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:10传代;每周2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:RPMI-7951细胞、H920细胞、Tn-5B1-4细胞
SUM 102 Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Swiss-3T3细胞、ID8/MOSEC细胞、NFS60细胞
KYSE30 Cells;背景说明:来源于一位64岁,患有高分化的中段食管鳞癌的男性患者。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:MCF7/WT细胞、Lymph Node Carcinoma of the prostate细胞、HCA7细胞
HAP1 YAP1 (-) 1 Cells(提供STR鉴定图谱)
HPDEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COLO_320DM细胞、Jurkat Clone E6-1细胞、HRGEC细胞
RPE D407 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:WM115F细胞、MGHU3细胞、SW-480细胞
HSKMC Cells;背景说明:骨骼肌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:C8166-CD4细胞、MD Anderson-Metastatic Breast-157细胞、SIRC细胞
L6 Cells;背景说明:该细胞是Yaffe在甲基胆蒽存在的情况下从大鼠大腿肌原代培养的最初两代细胞中分离得到的;在培养基中融合形成多核的肌管和横纹肌纤维,细胞融合的程度随着代数的增加而下降,因此细胞应低代次冷冻并周期性地重新克隆以选择融合能力强的细胞。鼠痘病毒阴性。该细胞应在达汇合状态前传代,以延缓细胞分化能力的丧失。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成肌细胞样;相关产品有:SKCO 1细胞、DU4475细胞、Michigan Cancer Foundation-12F细胞
SK-GT-4 Cells人食道癌肿瘤细胞库专业复苏|带STR图谱
HeLa-RFP Cells人宫颈癌细胞库专业复苏|带STR图谱
HPAC Cells人胰腺腺泡上皮癌细胞库专业复苏|带STR图谱
KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱
LS180 Cells人结肠癌细胞库专业复苏|带STR图谱
GBC-SD Cells人胆囊癌细胞库专业复苏|带STR图谱
RSC96 Cells大鼠雪旺细胞库专业复苏|带STR图谱
MasterShef10 Cells(提供STR鉴定图谱)
ND09990 Cells(提供STR鉴定图谱)
PJS5JTO Cells(提供STR鉴定图谱)
TR-LCC-1 Cells(提供STR鉴定图谱)
UIV-SL-373 Cells(提供STR鉴定图谱)
" "PubMed=9033652; DOI=10.1002/(SICI)1097-0215(19970207)70:4<437::AID-IJC11>3.0.CO;2-C
Tanaka H., Shimada Y., Imamura M., Shibagaki I., Ishizaki K.
Multiple types of aberrations in the p16 (INK4a) and the p15(INK4b) genes in 30 esophageal squamous-cell-carcinoma cell lines.
Int. J. Cancer 70:437-442(1997)
PubMed=11092977; DOI=10.1111/j.1349-7006.2000.tb00895.x; PMCID=PMC5926289
Pimkhaokham A., Shimada Y., Fukuda Y., Kurihara N., Imoto I., Yang Z.-Q., Imamura M., Nakamura Y., Amagasa T., Inazawa J.
Nonrandom chromosomal imbalances in esophageal squamous cell carcinoma cell lines: possible involvement of the ATF3 and CENPF genes in the 1q32 amplicon.
Jpn. J. Cancer Res. 91:1126-1133(2000)
PubMed=15172977; DOI=10.1158/0008-5472.CAN-04-0172
Sonoda I., Imoto I., Inoue J., Shibata T., Shimada Y., Chin K., Imamura M., Amagasa T., Gray J.W., Hirohashi S., Inazawa J.
Frequent silencing of low density lipoprotein receptor-related protein 1B (LRP1B) expression by genetic and epigenetic mechanisms in esophageal squamous cell carcinoma.
Cancer Res. 64:3741-3747(2004)
PubMed=16045545; DOI=10.1111/j.0959-9673.2005.00431.x; PMCID=PMC2517430
Ban S., Michikawa Y., Ishikawa K.-i., Sagara M., Watanabe K., Shimada Y., Inazawa J., Imai T.
Radiation sensitivities of 31 human oesophageal squamous cell carcinoma cell lines.
Int. J. Exp. Pathol. 86:231-240(2005)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验该产品被引用文献
"PubMed=9033652; DOI=10.1002/(SICI)1097-0215(19970207)70:4<437::AID-IJC11>3.0.CO;2-C
Tanaka H., Shimada Y., Imamura M., Shibagaki I., Ishizaki K.
Multiple types of aberrations in the p16 (INK4a) and the p15(INK4b) genes in 30 esophageal squamous-cell-carcinoma cell lines.
Int. J. Cancer 70:437-442(1997)
PubMed=11092977; DOI=10.1111/j.1349-7006.2000.tb00895.x; PMCID=PMC5926289
Pimkhaokham A., Shimada Y., Fukuda Y., Kurihara N., Imoto I., Yang Z.-Q., Imamura M., Nakamura Y., Amagasa T., Inazawa J.
Nonrandom chromosomal imbalances in esophageal squamous cell carcinoma cell lines: possible involvement of the ATF3 and CENPF genes in the 1q32 amplicon.
Jpn. J. Cancer Res. 91:1126-1133(2000)
PubMed=15172977; DOI=10.1158/0008-5472.CAN-04-0172
Sonoda I., Imoto I., Inoue J., Shibata T., Shimada Y., Chin K., Imamura M., Amagasa T., Gray J.W., Hirohashi S., Inazawa J.
Frequent silencing of low density lipoprotein receptor-related protein 1B (LRP1B) expression by genetic and epigenetic mechanisms in esophageal squamous cell carcinoma.
Cancer Res. 64:3741-3747(2004)
PubMed=16045545; DOI=10.1111/j.0959-9673.2005.00431.x; PMCID=PMC2517430
Ban S., Michikawa Y., Ishikawa K.-i., Sagara M., Watanabe K., Shimada Y., Inazawa J., Imai T.
Radiation sensitivities of 31 human oesophageal squamous cell carcinoma cell lines.
Int. J. Exp. Pathol. 86:231-240(2005)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"
Tanaka H., Shimada Y., Imamura M., Shibagaki I., Ishizaki K.
Multiple types of aberrations in the p16 (INK4a) and the p15(INK4b) genes in 30 esophageal squamous-cell-carcinoma cell lines.
Int. J. Cancer 70:437-442(1997)
PubMed=11092977; DOI=10.1111/j.1349-7006.2000.tb00895.x; PMCID=PMC5926289
Pimkhaokham A., Shimada Y., Fukuda Y., Kurihara N., Imoto I., Yang Z.-Q., Imamura M., Nakamura Y., Amagasa T., Inazawa J.
Nonrandom chromosomal imbalances in esophageal squamous cell carcinoma cell lines: possible involvement of the ATF3 and CENPF genes in the 1q32 amplicon.
Jpn. J. Cancer Res. 91:1126-1133(2000)
PubMed=15172977; DOI=10.1158/0008-5472.CAN-04-0172
Sonoda I., Imoto I., Inoue J., Shibata T., Shimada Y., Chin K., Imamura M., Amagasa T., Gray J.W., Hirohashi S., Inazawa J.
Frequent silencing of low density lipoprotein receptor-related protein 1B (LRP1B) expression by genetic and epigenetic mechanisms in esophageal squamous cell carcinoma.
Cancer Res. 64:3741-3747(2004)
PubMed=16045545; DOI=10.1111/j.0959-9673.2005.00431.x; PMCID=PMC2517430
Ban S., Michikawa Y., Ishikawa K.-i., Sagara M., Watanabe K., Shimada Y., Inazawa J., Imai T.
Radiation sensitivities of 31 human oesophageal squamous cell carcinoma cell lines.
Int. J. Exp. Pathol. 86:231-240(2005)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"
 技术资料
技术资料暂无技术资料 索取技术资料
文献支持
KYSE-140 Cells人食管鳞癌细胞传代培养|STR图谱
¥850 - 5500