相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 品系:
详见细胞说明资料
- 细胞类型:
详见细胞说明资料
- 肿瘤类型:
详见细胞说明资料
- 供应商:
上海冠导生物工程有限公司
- 库存:
≥100瓶
- 生长状态:
详见细胞说明资料
- 年限:
详见细胞说明资料
- 运输方式:
常温运输【复苏细胞】或干冰运输【冻存细胞】
- 器官来源:
详见细胞说明资料
- 是否是肿瘤细胞:
详见细胞说明资料
- 细胞形态:
详见细胞说明资料
- 免疫类型:
详见细胞说明资料
- 物种来源:
详见细胞说明资料
- 相关疾病:
详见细胞说明资料
- 组织来源:
详见细胞说明资料
- 英文名:
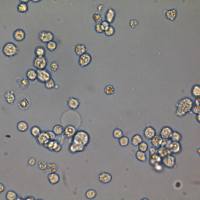
NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱
- 规格:
1*10(6)Cellls/瓶
"NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱
RS1 Cells大鼠皮肤成纤维样细胞保种中心|STR图谱
L929-GFP Cells小鼠成纤维细胞保种中心|STR图谱
KG-1 Cells人急性骨髓白血病细胞保种中心|STR图谱
HEK293-FT Cells人胚肾细胞保种中心|STR图谱
HepG2-GS Cells人肝癌细胞保种中心|STR图谱
KHYG-1 Cells人自然杀伤细胞淋巴细胞白血病细胞保种中心|STR图谱
MFC-Luc Cells小鼠前胃癌细胞保种中心|STR图谱
SV40 MES 13 Cells小鼠肾小球系膜细胞保种中心|STR图谱
物种来源:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱来源于人源、鼠源等其它物种来源
生长特性:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱贴壁生长
标准复苏流程包括:快速解冻:将冻存管从液氮中取出后立即置于37℃水浴,持续摇动直至仅剩少量冰晶(通常需1-2分钟),梯度稀释:将细胞悬液缓慢加入预热的完全培养基(含10%血清),逐步降低DMSO浓度离心清洗:1000rpm离心5分钟去除冻存保护剂,重悬接种:按适当密度接种于预处理好的培养器皿。
细胞系在贴壁的过程中仍然会聚集,这个是贴壁培养的细胞,尤其是肿瘤细胞的一个特性,你可以尝试,准备100%的单个细胞悬液,贴壁后观察细胞,仍然是几个几个细胞聚集在一起。一些悬浮培养细胞也是如此,容易聚集,不要过几个小时就拿出来吹打成单细胞悬液。细胞只要能从基质上脱离下来,即使是成片的(比如Calu-3细胞),吹打不超过20次(一般10次即可),成小规模聚集(10个细胞左右)是正常的,不要再去延长消化时间,等待单细胞悬液出现。比较难消化的细胞:润洗方法5min还不能消化,以结肠癌细胞为例,比如:HCT15、LS411和KM12细胞,胰酶消化,一般10 cm培养皿,一次加入300ul-500ul就足够了。即使这样难消化的细胞,一般不超过5min,即可见细胞成片移动,就应该停止消化。
形态特性:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱上皮细胞样
换液周期:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱每周2-3次
产品包装:复苏发货:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
细胞传代培养作为实验室常规操作,看似简单的消化步骤实则暗藏玄机。许多研究者都曾遇到过这样的困惑:细胞在最初几代生长状态良好,但随着传代次数增加,细胞状态逐渐恶化,出现增殖缓慢、形态改变、贴壁能力下降等问题。这往往并非细胞本身的问题,而是消化环节操作不当导致的累积性损伤。深入理解消化过程的生物学机制,掌握正确的操作技巧,对维持细胞长期稳定的生长状态至关重要。
虽然1×10^6 cells/mL是通用推荐值,但特定细胞可能需要调整:原代细胞:如肝细胞或神经元,通常需要更高密度(2-5×10^6 cells/mL)以维持功能完整性;悬浮细胞:如淋巴细胞,对密度耐受性较强,但低于5×10^5 cells/mL仍会导致活性显著降低;干细胞:间充质干细胞(MSCs)在1.5×10^6 cells/mL时表现出最佳分化潜能保留。值得注意的是,密度过高(如>5×10^6 cells/mL)可能引起冻存液渗透压失衡,反而降低复苏效率。因此,优化需结合具体细胞特性。
背景信息:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱详见相关文献介绍
传代比例:1:2-1:4(NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱首次传代建议1:2)
来源说明:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱主要来源ATCC、ECACC、DSMZ、RCB等细胞库
准确判断胰酶消化是否完成,首先,观察细胞形态变化是判断胰酶消化程度的重要依据。在加入胰酶后,随着消化的进行,原本贴壁生长的细胞会逐渐变圆。当大部分细胞呈现出圆形,且细胞之间的连接变得松散时,说明消化正在进行中。如果细胞完全脱离培养瓶底部,变成单个的圆形细胞悬浮在消化液中,这通常是消化完成的一个重要标志。此时,在显微镜下观察,细胞应该是圆润、饱满且边界清晰的。其次,可以通过轻轻拍打培养瓶或轻轻晃动培养板来辅助判断。如果细胞很容易从瓶底或板底脱落并在液体中悬浮,说明消化程度可能已经比较合适。但要注意动作要轻柔,避免过度剧烈的晃动对细胞造成损伤。
无菌操作规范构成了细胞培养的""卫生防疫体系""。从生物安全柜的定期验证到培养器具的高压灭菌,每个环节都堪比婴儿用品的消毒流程。实验人员穿戴的实验服、口罩和手套,就像新生儿护理时的防护装备。曾有研究显示,严格遵循无菌操作规范的实验室,其细胞污染率可控制在0.5%以下,而操作疏忽的实验室污染率可能高达15%。这种对洁净度的追求,使得现代细胞培养间达到了手术室级别的环境标准。
CTX TNA2 Cells大鼠脑I型星形胶质细胞保种中心|STR图谱
MuM-2B Cells人侵袭性脉络膜黑色素瘤细胞保种中心|STR图谱
B16-F10+luc Cells小鼠黑色素瘤细胞保种中心|STR图谱
MDA-MB-436-Luc Cells人乳腺癌细胞保种中心|STR图谱
HCC-9204 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MNNGHOS细胞、Sp2/0-Ag-14细胞、FTC-133细胞
SW1463 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:8传代,每周换液1-2次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:H69细胞、SF539细胞、TE1细胞
U-373MG Cells;背景说明:胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Astrocyte type I clone细胞、FU-97细胞、DoTc2 4510细胞
GM20997 Cells(提供STR鉴定图谱)
HAP1 L1TD1 (-) 1 Cells(提供STR鉴定图谱)
IM-9 Cells人外周血B淋巴细胞保种中心|STR图谱
NCI-H1417 Cells人小细胞肺癌细胞保种中心|STR图谱
MOLM-13 Cells人急性髓白血病细胞保种中心|STR图谱
SKOV3-RFP Cells人卵巢癌细胞保种中心|STR图谱
NCI-H596 Cells人肺腺鳞癌细胞保种中心|STR图谱
Lec1 Cells仓鼠卵巢细胞保种中心|STR图谱
TO175T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:CMT167细胞、U-343 MG细胞、LM TK negative细胞
OV3121 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BCP-1细胞、NCI-BL1339细胞、Hs294细胞
FHs74 Int Cells;背景说明:小肠;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HRC-99细胞、Med 283细胞、LO2细胞
ACHN Cells;背景说明:该细胞1979年建系,源自一名22岁患有肾细胞腺癌的白人男性的胸腔积液。干扰素可抑制该细胞的生长,该细胞多用于干扰素及其诱导剂的抗增殖研究。;传代方法:1:2-1:3传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HCV 29细胞、BrCL18细胞、MDA-MB-468细胞
SCC-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCIH1048细胞、TE7细胞、SUM-149PT细胞
Pa17C Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H-345细胞、2B4-L细胞、HT 1197.T细胞
Gingival carcinoma Neck Metastasis Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NIH/3T3细胞、BV-173细胞、HEK/293细胞
SK-HEP-1 Cells人肝癌细胞保种中心|STR图谱
IM-95M Cells人胃腺癌细胞保种中心|STR图谱
SU.86.86 Cells人胰腺导管癌细胞保种中心|STR图谱
A-875 Cells人黑色素瘤细胞保种中心|STR图谱
U-251MG Cells人胶质瘤细胞保种中心|STR图谱
NCI-H460/cis Cells人大细胞肺癌细胞保种中心|STR图谱
TR-LNN-1 Cells人喉癌淋巴结转移细胞保种中心|STR图谱
Madin-Darby Canine Kidney Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:U-251细胞、CCC-HEL-1细胞、RERF-LC-MS细胞
HPSC-TAg Cells(提供STR鉴定图谱)
K10M5 Cells(提供STR鉴定图谱)
Me Leb Cells(提供STR鉴定图谱)
NGPrMEL4000 Cells(提供STR鉴定图谱)
Sp2/HL Cells(提供STR鉴定图谱)
NIH/3T3-RFP Cells小鼠胚胎成纤维细胞保种中心|STR图谱
YT Cells人NK细胞白血病细胞保种中心|STR图谱
PX2 Cells(提供STR鉴定图谱)
TRNDi012-I Cells(提供STR鉴定图谱)
UKHGi003-A Cells(提供STR鉴定图谱)
HCT116-SLC25A33-KO-c7 Cells(提供STR鉴定图谱)
SGC7901 Cells;背景说明:1979年建系;这株细胞源自一位56岁女性胃腺癌患者的淋巴结转移灶。RPMI-1640培养12天,细胞开始生长,首次传代10天;31个月中传代186代。免疫抑制的ICR小鼠、乳犬皮下移植成功,家兔、乳犬眼前房移植成功;淋巴结转移,从小鼠皮下侵袭至肌层。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Daoy细胞、Tokyo Medical and Dental university 8细胞、MDA-415细胞
R-1059-D Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:DanG细胞、C41细胞、RBL-1细胞
Hs-600-T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:H-1781细胞、TMK1细胞、FM88细胞
H865 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HEK 293FT细胞、B16F0细胞、R-1059-D细胞
HTR-8/SVneo Cells人绒毛膜滋养层细胞保种中心|STR图谱
M5076 Cells小鼠网织细胞肉瘤
Capan-1 Cells人胰腺癌细胞保种中心|STR图谱
GH3 Cells大鼠垂体瘤细胞保种中心|STR图谱
PA-1 Cells人卵巢畸胎瘤细胞保种中心|STR图谱
Daudi-Luc Cells人Burkkit淋巴瘤细胞保种中心|STR图谱
C3H 10T1/2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CA 46细胞、H2126细胞、HCC44细胞
253JB-V Cells;背景说明:膀胱癌;淋巴结转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OV2008细胞、HSC-6细胞、NCI-HUT-520细胞
Ly19 Cells;背景说明:弥漫大B淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CRFK细胞、SuperTube细胞、NB9细胞
Ly19 Cells;背景说明:弥漫大B淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CRFK细胞、SuperTube细胞、NB9细胞
Ly10 Cells;背景说明:弥漫大B细胞淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SK-LU-1细胞、NRK 52E细胞、HT 144细胞
NCIH446 Cells;背景说明:该细胞是1982年由CarneyD和GazdarAF等从一位小细胞肺癌患者的胸腔积液中建立的。细胞的原始形态并不具有小细胞肺癌特征。这个细胞株是小细胞肺癌的生化和形态学上的变种,表达神经元特有的烯醇酶和脑型肌酸激酶同工酶;左旋多巴脱羧酶、蚕素、抗利尿激素、催产素或胃泌激素释放肽未达到可检测水平。与正常细胞相比,该细胞c-mycDNA序列扩增约20倍,RNA增加15倍。最初传代培养基用含有5%FBS的RPMI1640,另外添加10nM化可的松、0.005mg/ml胰岛素、0.01mg/ml转铁;传代方法:1:2传代;生长特性:贴壁/悬浮生长,混合;形态特性:上皮样;相关产品有:Medical University of Graz-Chordoma 1细胞、Ketr3细胞、HRA 19细胞
BHK-21 Cells仓鼠肾成纤维细胞保种中心|STR图谱
sNF96.2 Cells人雪旺细胞保种中心|STR图谱
MT-4 Cells人急性淋巴母细胞白血病细胞保种中心|STR图谱
LLC-PK1 Cells猪肾细胞保种中心|STR图谱
DU145 Cells人前列腺癌细胞保种中心|STR图谱
4T1/OVA Cells小鼠乳腺癌细胞保种中心|STR图谱
DITNC1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维母细胞样;相关产品有:GES-1细胞、LU165细胞、HEK-AD293细胞
Human Kidney-2 Cells;背景说明:该细胞属源于正常肾的近曲小管细胞,通过导入HPV-16 E6/E7基因而获得永生化。将含有HPV-16 E6/E7基因的重组的逆转录病毒载体pLXSN 16 E6/E7转染外生包装细胞Psi-2,Psi-2细胞产生的病毒再去感染兼嗜性包装细胞系PA317,最后将PA317产生的病毒颗粒导入正常的肾皮质近曲小管细胞。尽管pLXSN 16 E6/E7中含有新霉素抗性,但未用G418筛选转导克隆。Southern和FISH分析显示HK-2细胞来源于单克隆。PCR检测证实HK-2细胞基因组中含有E6/E7基因。;传代方法:1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCI-H1975细胞、B16-F10细胞、H-1876细胞
HO1-N-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:143TK-细胞、NCIH847细胞、YD15细胞
SW-620 Cells;背景说明:SW620是从一个51岁男性白人组织中分离得到。由A.Leibovitz等从一个淋巴结建株。细胞系主要由无绒毛的小园球细胞和双极细胞组成。它仅合成少量癌胚抗原(CEA)且在裸鼠中有高度的致瘤性;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:COR-L51细胞、CHO/dhFr-细胞、SW1088细胞
Ly7 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CBRH-7919细胞、H-748细胞、MD Anderson-Metastatic Breast-453细胞
T-47D Cells人乳腺管癌细胞保种中心|STR图谱
LLC/OVA Cells小鼠肺癌细胞保种中心|STR图谱
HLE Cells人肝癌细胞保种中心|STR图谱
BJ [Human fibroblast] Cells人皮肤成纤维细胞保种中心|STR图谱
CCC-ESF-1 Cells人胚皮肤成纤维细胞保种中心|STR图谱
TE-12 Cells人食管癌细胞保种中心|STR图谱
OE19 Cells人食管癌细胞保种中心|STR图谱
L-02 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H4IIE细胞、H-2330细胞、ECC 12细胞
U266B1 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:OV433细胞、EO771细胞、OV1063细胞
MDA-MB157 Cells;背景说明:该细胞源自一位患有乳腺髓样癌的44岁黑人女性,表达WNT7B癌基因,细胞与细胞边界处有细胞桥粒、微绒毛、张力细丝。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:hUC-MSC细胞、CZ-1细胞、SUM-102细胞
RPMI-7666 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:T HEECs细胞、NTERA-2/D1细胞、Hs281T细胞
C26 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LS123细胞、TNC-1B12B4细胞、H9c2细胞
VeroC1008 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Bronchial Epithelium transformed with Ad12-SV40 2B细胞、CEMO-1细胞、EST81细胞
CL MC/9 Cells;背景说明:肥大细胞;C57BL/6 x A/J;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HFL 1细胞、Walker256-TC细胞、NOR-10细胞
AZ-521 Cells;背景说明:详见相关文献介绍;传代方法:1:4传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:上皮样;相关产品有:MADISON LUNG TA-109细胞、MOLT.4细胞、T98-G细胞
" "PubMed=10987304
Girard L., Zochbauer-Muller S., Virmani A.K., Gazdar A.F., Minna J.D.
Genome-wide allelotyping of lung cancer identifies new regions of allelic loss, differences between small cell lung cancer and non-small cell lung cancer, and loci clustering.
Cancer Res. 60:4894-4906(2000)
PubMed=11030152; DOI=10.1038/sj.onc.1203815
Modi S., Kubo A., Oie H.K., Coxon A.B., Rehmatulla A., Kaye F.J.
Protein expression of the RB-related gene family and SV40 large T antigen in mesothelioma and lung cancer.
Oncogene 19:4632-4639(2000)
PubMed=11314036; DOI=10.1038/sj.onc.1204211
Forgacs E., Wren J.D., Kamibayashi C., Kondo M., Xu X.L., Markowitz S.D., Tomlinson G.E., Muller C.Y., Gazdar A.F., Garner H.R., Minna J.D.
Searching for microsatellite mutations in coding regions in lung, breast, ovarian and colorectal cancers.
Oncogene 20:1005-1009(2001)
PubMed=11416159; DOI=10.1073/pnas.121616198; PMCID=PMC35459
Masters J.R.W., Thomson J.A., Daly-Burns B., Reid Y.A., Dirks W.G., Packer P., Toji L.H., Ohno T., Tanabe H., Arlett C.F., Kelland L.R., Harrison M., Virmani A.K., Ward T.H., Ayres K.L., Debenham P.G.
Short tandem repeat profiling provides an international reference standard for human cell lines.
Proc. Natl. Acad. Sci. U.S.A. 98:8012-8017(2001)
PubMed=14762065; DOI=10.1101/gr.2012304; PMCID=PMC327104
Bignell G.R., Huang J., Greshock J., Watt S., Butler A.P., West S., Grigorova M., Jones K.W., Wei W., Stratton M.R., Futreal P.A., Weber B., Shapero M.H., Wooster R.
High-resolution analysis of DNA copy number using oligonucleotide microarrays.
Genome Res. 14:287-295(2004)
PubMed=16157194; DOI=10.1016/j.cancergencyto.2005.03.007
Grigorova M., Lyman R.C., Caldas C., Edwards P.A.W.
Chromosome abnormalities in 10 lung cancer cell lines of the NCI-H series analyzed with spectral karyotyping.
Cancer Genet. Cytogenet. 162:1-9(2005)
PubMed=19153074; DOI=10.1093/hmg/ddp034
Medina P.P., Castillo S.D., Blanco S., Sanz-Garcia M., Largo C., Alvarez S., Yokota J., Gonzalez-Neira A., Benitez J., Clevers H.C., Cigudosa J.C., Lazo P.A., Sanchez-Cespedes M.
The SRY-HMG box gene, SOX4, is a target of gene amplification at chromosome 6p in lung cancer.
Hum. Mol. Genet. 18:1343-1352(2009)
PubMed=19472407; DOI=10.1002/humu.21028; PMCID=PMC2900846
Blanco R., Iwakawa R., Tang M.-Y., Kohno T., Angulo B., Pio R., Montuenga L.M., Minna J.D., Yokota J., Sanchez-Cespedes M.
A gene-alteration profile of human lung cancer cell lines.
Hum. Mutat. 30:1199-1206(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20557307; DOI=10.1111/j.1349-7006.2010.01622.x; PMCID=PMC11158680
Iwakawa R., Kohno T., Enari M., Kiyono T., Yokota J.
Prevalence of human papillomavirus 16/18/33 infection and p53 mutation in lung adenocarcinoma.
Cancer Sci. 101:1891-1896(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22961666; DOI=10.1158/2159-8290.CD-12-0112; PMCID=PMC3567922
Byers L.A., Wang J., Nilsson M.B., Fujimoto J., Saintigny P., Yordy J.S., Giri U., Peyton M., Fan Y.-H., Diao L.-X., Masrorpour F., Shen L., Liu W.-B., Duchemann B., Tumula P., Bhardwaj V., Welsh J., Weber S., Glisson B.S., Kalhor N., Wistuba I.I., Girard L., Lippman S.M., Mills G.B., Coombes K.R., Weinstein J.N., Minna J.D., Heymach J.V.
Proteomic profiling identifies dysregulated pathways in small cell lung cancer and novel therapeutic targets including PARP1.
Cancer Discov. 2:798-811(2012)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26361996; DOI=10.1016/j.jprot.2015.09.003
Grundner-Culemann K., Dybowski J.N., Klammer M., Tebbe A., Schaab C., Daub H.
Comparative proteome analysis across non-small cell lung cancer cell lines.
J. Proteomics 130:1-10(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=29681454; DOI=10.1016/j.cell.2018.03.028; PMCID=PMC5935540
McMillan E.A., Ryu M.-J., Diep C.H., Mendiratta S., Clemenceau J.R., Vaden R.M., Kim J.-H., Motoyaji T., Covington K.R., Peyton M., Huffman K., Wu X.-F., Girard L., Sung Y., Chen P.-H., Mallipeddi P.L., Lee J.Y., Hanson J., Voruganti S., Yu Y., Park S., Sudderth J., DeSevo C., Muzny D.M., Doddapaneni H., Gazdar A.F., Gibbs R.A., Hwang T.H., Heymach J.V., Wistuba I.I., Coombes K.R., Williams N.S., Wheeler D.A., MacMillan J.B., DeBerardinis R.J., Roth M.G., Posner B.A., Minna J.D., Kim H.S., White M.A.
Chemistry-first approach for nomination of personalized treatment in lung cancer.
Cell 173:864-878.e29(2018)
PubMed=30038707; DOI=10.18632/oncotarget.25642; PMCID=PMC6049873
Du L.-Q., Zhao Z.-Z., Suraokar M.B., Shelton S.S., Ma X.-Y., Hsiao T.-H., Minna J.D., Wistuba I.I., Pertsemlidis A.
LMO1 functions as an oncogene by regulating TTK expression and correlates with neuroendocrine differentiation of lung cancer.
Oncotarget 9:29601-29618(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"
RS1 Cells大鼠皮肤成纤维样细胞保种中心|STR图谱
L929-GFP Cells小鼠成纤维细胞保种中心|STR图谱
KG-1 Cells人急性骨髓白血病细胞保种中心|STR图谱
HEK293-FT Cells人胚肾细胞保种中心|STR图谱
HepG2-GS Cells人肝癌细胞保种中心|STR图谱
KHYG-1 Cells人自然杀伤细胞淋巴细胞白血病细胞保种中心|STR图谱
MFC-Luc Cells小鼠前胃癌细胞保种中心|STR图谱
SV40 MES 13 Cells小鼠肾小球系膜细胞保种中心|STR图谱
物种来源:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱来源于人源、鼠源等其它物种来源
生长特性:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱贴壁生长
标准复苏流程包括:快速解冻:将冻存管从液氮中取出后立即置于37℃水浴,持续摇动直至仅剩少量冰晶(通常需1-2分钟),梯度稀释:将细胞悬液缓慢加入预热的完全培养基(含10%血清),逐步降低DMSO浓度离心清洗:1000rpm离心5分钟去除冻存保护剂,重悬接种:按适当密度接种于预处理好的培养器皿。
细胞系在贴壁的过程中仍然会聚集,这个是贴壁培养的细胞,尤其是肿瘤细胞的一个特性,你可以尝试,准备100%的单个细胞悬液,贴壁后观察细胞,仍然是几个几个细胞聚集在一起。一些悬浮培养细胞也是如此,容易聚集,不要过几个小时就拿出来吹打成单细胞悬液。细胞只要能从基质上脱离下来,即使是成片的(比如Calu-3细胞),吹打不超过20次(一般10次即可),成小规模聚集(10个细胞左右)是正常的,不要再去延长消化时间,等待单细胞悬液出现。比较难消化的细胞:润洗方法5min还不能消化,以结肠癌细胞为例,比如:HCT15、LS411和KM12细胞,胰酶消化,一般10 cm培养皿,一次加入300ul-500ul就足够了。即使这样难消化的细胞,一般不超过5min,即可见细胞成片移动,就应该停止消化。
形态特性:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱上皮细胞样
换液周期:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱每周2-3次
产品包装:复苏发货:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
细胞传代培养作为实验室常规操作,看似简单的消化步骤实则暗藏玄机。许多研究者都曾遇到过这样的困惑:细胞在最初几代生长状态良好,但随着传代次数增加,细胞状态逐渐恶化,出现增殖缓慢、形态改变、贴壁能力下降等问题。这往往并非细胞本身的问题,而是消化环节操作不当导致的累积性损伤。深入理解消化过程的生物学机制,掌握正确的操作技巧,对维持细胞长期稳定的生长状态至关重要。
虽然1×10^6 cells/mL是通用推荐值,但特定细胞可能需要调整:原代细胞:如肝细胞或神经元,通常需要更高密度(2-5×10^6 cells/mL)以维持功能完整性;悬浮细胞:如淋巴细胞,对密度耐受性较强,但低于5×10^5 cells/mL仍会导致活性显著降低;干细胞:间充质干细胞(MSCs)在1.5×10^6 cells/mL时表现出最佳分化潜能保留。值得注意的是,密度过高(如>5×10^6 cells/mL)可能引起冻存液渗透压失衡,反而降低复苏效率。因此,优化需结合具体细胞特性。
背景信息:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱详见相关文献介绍
传代比例:1:2-1:4(NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱首次传代建议1:2)
来源说明:NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱主要来源ATCC、ECACC、DSMZ、RCB等细胞库
准确判断胰酶消化是否完成,首先,观察细胞形态变化是判断胰酶消化程度的重要依据。在加入胰酶后,随着消化的进行,原本贴壁生长的细胞会逐渐变圆。当大部分细胞呈现出圆形,且细胞之间的连接变得松散时,说明消化正在进行中。如果细胞完全脱离培养瓶底部,变成单个的圆形细胞悬浮在消化液中,这通常是消化完成的一个重要标志。此时,在显微镜下观察,细胞应该是圆润、饱满且边界清晰的。其次,可以通过轻轻拍打培养瓶或轻轻晃动培养板来辅助判断。如果细胞很容易从瓶底或板底脱落并在液体中悬浮,说明消化程度可能已经比较合适。但要注意动作要轻柔,避免过度剧烈的晃动对细胞造成损伤。
无菌操作规范构成了细胞培养的""卫生防疫体系""。从生物安全柜的定期验证到培养器具的高压灭菌,每个环节都堪比婴儿用品的消毒流程。实验人员穿戴的实验服、口罩和手套,就像新生儿护理时的防护装备。曾有研究显示,严格遵循无菌操作规范的实验室,其细胞污染率可控制在0.5%以下,而操作疏忽的实验室污染率可能高达15%。这种对洁净度的追求,使得现代细胞培养间达到了手术室级别的环境标准。
CTX TNA2 Cells大鼠脑I型星形胶质细胞保种中心|STR图谱
MuM-2B Cells人侵袭性脉络膜黑色素瘤细胞保种中心|STR图谱
B16-F10+luc Cells小鼠黑色素瘤细胞保种中心|STR图谱
MDA-MB-436-Luc Cells人乳腺癌细胞保种中心|STR图谱
HCC-9204 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MNNGHOS细胞、Sp2/0-Ag-14细胞、FTC-133细胞
SW1463 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:8传代,每周换液1-2次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:H69细胞、SF539细胞、TE1细胞
U-373MG Cells;背景说明:胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Astrocyte type I clone细胞、FU-97细胞、DoTc2 4510细胞
GM20997 Cells(提供STR鉴定图谱)
HAP1 L1TD1 (-) 1 Cells(提供STR鉴定图谱)
IM-9 Cells人外周血B淋巴细胞保种中心|STR图谱
NCI-H1417 Cells人小细胞肺癌细胞保种中心|STR图谱
MOLM-13 Cells人急性髓白血病细胞保种中心|STR图谱
SKOV3-RFP Cells人卵巢癌细胞保种中心|STR图谱
NCI-H596 Cells人肺腺鳞癌细胞保种中心|STR图谱
Lec1 Cells仓鼠卵巢细胞保种中心|STR图谱
TO175T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:CMT167细胞、U-343 MG细胞、LM TK negative细胞
OV3121 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BCP-1细胞、NCI-BL1339细胞、Hs294细胞
FHs74 Int Cells;背景说明:小肠;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HRC-99细胞、Med 283细胞、LO2细胞
ACHN Cells;背景说明:该细胞1979年建系,源自一名22岁患有肾细胞腺癌的白人男性的胸腔积液。干扰素可抑制该细胞的生长,该细胞多用于干扰素及其诱导剂的抗增殖研究。;传代方法:1:2-1:3传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HCV 29细胞、BrCL18细胞、MDA-MB-468细胞
SCC-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCIH1048细胞、TE7细胞、SUM-149PT细胞
Pa17C Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H-345细胞、2B4-L细胞、HT 1197.T细胞
Gingival carcinoma Neck Metastasis Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NIH/3T3细胞、BV-173细胞、HEK/293细胞
SK-HEP-1 Cells人肝癌细胞保种中心|STR图谱
IM-95M Cells人胃腺癌细胞保种中心|STR图谱
SU.86.86 Cells人胰腺导管癌细胞保种中心|STR图谱
A-875 Cells人黑色素瘤细胞保种中心|STR图谱
U-251MG Cells人胶质瘤细胞保种中心|STR图谱
NCI-H460/cis Cells人大细胞肺癌细胞保种中心|STR图谱
TR-LNN-1 Cells人喉癌淋巴结转移细胞保种中心|STR图谱
Madin-Darby Canine Kidney Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:U-251细胞、CCC-HEL-1细胞、RERF-LC-MS细胞
HPSC-TAg Cells(提供STR鉴定图谱)
K10M5 Cells(提供STR鉴定图谱)
Me Leb Cells(提供STR鉴定图谱)
NGPrMEL4000 Cells(提供STR鉴定图谱)
Sp2/HL Cells(提供STR鉴定图谱)
NIH/3T3-RFP Cells小鼠胚胎成纤维细胞保种中心|STR图谱
YT Cells人NK细胞白血病细胞保种中心|STR图谱
PX2 Cells(提供STR鉴定图谱)
TRNDi012-I Cells(提供STR鉴定图谱)
UKHGi003-A Cells(提供STR鉴定图谱)
HCT116-SLC25A33-KO-c7 Cells(提供STR鉴定图谱)
SGC7901 Cells;背景说明:1979年建系;这株细胞源自一位56岁女性胃腺癌患者的淋巴结转移灶。RPMI-1640培养12天,细胞开始生长,首次传代10天;31个月中传代186代。免疫抑制的ICR小鼠、乳犬皮下移植成功,家兔、乳犬眼前房移植成功;淋巴结转移,从小鼠皮下侵袭至肌层。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Daoy细胞、Tokyo Medical and Dental university 8细胞、MDA-415细胞
R-1059-D Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:DanG细胞、C41细胞、RBL-1细胞
Hs-600-T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:H-1781细胞、TMK1细胞、FM88细胞
H865 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HEK 293FT细胞、B16F0细胞、R-1059-D细胞
HTR-8/SVneo Cells人绒毛膜滋养层细胞保种中心|STR图谱
M5076 Cells小鼠网织细胞肉瘤
Capan-1 Cells人胰腺癌细胞保种中心|STR图谱
GH3 Cells大鼠垂体瘤细胞保种中心|STR图谱
PA-1 Cells人卵巢畸胎瘤细胞保种中心|STR图谱
Daudi-Luc Cells人Burkkit淋巴瘤细胞保种中心|STR图谱
C3H 10T1/2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CA 46细胞、H2126细胞、HCC44细胞
253JB-V Cells;背景说明:膀胱癌;淋巴结转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OV2008细胞、HSC-6细胞、NCI-HUT-520细胞
Ly19 Cells;背景说明:弥漫大B淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CRFK细胞、SuperTube细胞、NB9细胞
Ly19 Cells;背景说明:弥漫大B淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CRFK细胞、SuperTube细胞、NB9细胞
Ly10 Cells;背景说明:弥漫大B细胞淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SK-LU-1细胞、NRK 52E细胞、HT 144细胞
NCIH446 Cells;背景说明:该细胞是1982年由CarneyD和GazdarAF等从一位小细胞肺癌患者的胸腔积液中建立的。细胞的原始形态并不具有小细胞肺癌特征。这个细胞株是小细胞肺癌的生化和形态学上的变种,表达神经元特有的烯醇酶和脑型肌酸激酶同工酶;左旋多巴脱羧酶、蚕素、抗利尿激素、催产素或胃泌激素释放肽未达到可检测水平。与正常细胞相比,该细胞c-mycDNA序列扩增约20倍,RNA增加15倍。最初传代培养基用含有5%FBS的RPMI1640,另外添加10nM化可的松、0.005mg/ml胰岛素、0.01mg/ml转铁;传代方法:1:2传代;生长特性:贴壁/悬浮生长,混合;形态特性:上皮样;相关产品有:Medical University of Graz-Chordoma 1细胞、Ketr3细胞、HRA 19细胞
BHK-21 Cells仓鼠肾成纤维细胞保种中心|STR图谱
sNF96.2 Cells人雪旺细胞保种中心|STR图谱
MT-4 Cells人急性淋巴母细胞白血病细胞保种中心|STR图谱
LLC-PK1 Cells猪肾细胞保种中心|STR图谱
DU145 Cells人前列腺癌细胞保种中心|STR图谱
4T1/OVA Cells小鼠乳腺癌细胞保种中心|STR图谱
DITNC1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维母细胞样;相关产品有:GES-1细胞、LU165细胞、HEK-AD293细胞
Human Kidney-2 Cells;背景说明:该细胞属源于正常肾的近曲小管细胞,通过导入HPV-16 E6/E7基因而获得永生化。将含有HPV-16 E6/E7基因的重组的逆转录病毒载体pLXSN 16 E6/E7转染外生包装细胞Psi-2,Psi-2细胞产生的病毒再去感染兼嗜性包装细胞系PA317,最后将PA317产生的病毒颗粒导入正常的肾皮质近曲小管细胞。尽管pLXSN 16 E6/E7中含有新霉素抗性,但未用G418筛选转导克隆。Southern和FISH分析显示HK-2细胞来源于单克隆。PCR检测证实HK-2细胞基因组中含有E6/E7基因。;传代方法:1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCI-H1975细胞、B16-F10细胞、H-1876细胞
HO1-N-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:143TK-细胞、NCIH847细胞、YD15细胞
SW-620 Cells;背景说明:SW620是从一个51岁男性白人组织中分离得到。由A.Leibovitz等从一个淋巴结建株。细胞系主要由无绒毛的小园球细胞和双极细胞组成。它仅合成少量癌胚抗原(CEA)且在裸鼠中有高度的致瘤性;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:COR-L51细胞、CHO/dhFr-细胞、SW1088细胞
Ly7 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CBRH-7919细胞、H-748细胞、MD Anderson-Metastatic Breast-453细胞
T-47D Cells人乳腺管癌细胞保种中心|STR图谱
LLC/OVA Cells小鼠肺癌细胞保种中心|STR图谱
HLE Cells人肝癌细胞保种中心|STR图谱
BJ [Human fibroblast] Cells人皮肤成纤维细胞保种中心|STR图谱
CCC-ESF-1 Cells人胚皮肤成纤维细胞保种中心|STR图谱
TE-12 Cells人食管癌细胞保种中心|STR图谱
OE19 Cells人食管癌细胞保种中心|STR图谱
L-02 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H4IIE细胞、H-2330细胞、ECC 12细胞
U266B1 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:OV433细胞、EO771细胞、OV1063细胞
MDA-MB157 Cells;背景说明:该细胞源自一位患有乳腺髓样癌的44岁黑人女性,表达WNT7B癌基因,细胞与细胞边界处有细胞桥粒、微绒毛、张力细丝。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:hUC-MSC细胞、CZ-1细胞、SUM-102细胞
RPMI-7666 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:T HEECs细胞、NTERA-2/D1细胞、Hs281T细胞
C26 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LS123细胞、TNC-1B12B4细胞、H9c2细胞
VeroC1008 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Bronchial Epithelium transformed with Ad12-SV40 2B细胞、CEMO-1细胞、EST81细胞
CL MC/9 Cells;背景说明:肥大细胞;C57BL/6 x A/J;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HFL 1细胞、Walker256-TC细胞、NOR-10细胞
AZ-521 Cells;背景说明:详见相关文献介绍;传代方法:1:4传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:上皮样;相关产品有:MADISON LUNG TA-109细胞、MOLT.4细胞、T98-G细胞
" "PubMed=10987304
Girard L., Zochbauer-Muller S., Virmani A.K., Gazdar A.F., Minna J.D.
Genome-wide allelotyping of lung cancer identifies new regions of allelic loss, differences between small cell lung cancer and non-small cell lung cancer, and loci clustering.
Cancer Res. 60:4894-4906(2000)
PubMed=11030152; DOI=10.1038/sj.onc.1203815
Modi S., Kubo A., Oie H.K., Coxon A.B., Rehmatulla A., Kaye F.J.
Protein expression of the RB-related gene family and SV40 large T antigen in mesothelioma and lung cancer.
Oncogene 19:4632-4639(2000)
PubMed=11314036; DOI=10.1038/sj.onc.1204211
Forgacs E., Wren J.D., Kamibayashi C., Kondo M., Xu X.L., Markowitz S.D., Tomlinson G.E., Muller C.Y., Gazdar A.F., Garner H.R., Minna J.D.
Searching for microsatellite mutations in coding regions in lung, breast, ovarian and colorectal cancers.
Oncogene 20:1005-1009(2001)
PubMed=11416159; DOI=10.1073/pnas.121616198; PMCID=PMC35459
Masters J.R.W., Thomson J.A., Daly-Burns B., Reid Y.A., Dirks W.G., Packer P., Toji L.H., Ohno T., Tanabe H., Arlett C.F., Kelland L.R., Harrison M., Virmani A.K., Ward T.H., Ayres K.L., Debenham P.G.
Short tandem repeat profiling provides an international reference standard for human cell lines.
Proc. Natl. Acad. Sci. U.S.A. 98:8012-8017(2001)
PubMed=14762065; DOI=10.1101/gr.2012304; PMCID=PMC327104
Bignell G.R., Huang J., Greshock J., Watt S., Butler A.P., West S., Grigorova M., Jones K.W., Wei W., Stratton M.R., Futreal P.A., Weber B., Shapero M.H., Wooster R.
High-resolution analysis of DNA copy number using oligonucleotide microarrays.
Genome Res. 14:287-295(2004)
PubMed=16157194; DOI=10.1016/j.cancergencyto.2005.03.007
Grigorova M., Lyman R.C., Caldas C., Edwards P.A.W.
Chromosome abnormalities in 10 lung cancer cell lines of the NCI-H series analyzed with spectral karyotyping.
Cancer Genet. Cytogenet. 162:1-9(2005)
PubMed=19153074; DOI=10.1093/hmg/ddp034
Medina P.P., Castillo S.D., Blanco S., Sanz-Garcia M., Largo C., Alvarez S., Yokota J., Gonzalez-Neira A., Benitez J., Clevers H.C., Cigudosa J.C., Lazo P.A., Sanchez-Cespedes M.
The SRY-HMG box gene, SOX4, is a target of gene amplification at chromosome 6p in lung cancer.
Hum. Mol. Genet. 18:1343-1352(2009)
PubMed=19472407; DOI=10.1002/humu.21028; PMCID=PMC2900846
Blanco R., Iwakawa R., Tang M.-Y., Kohno T., Angulo B., Pio R., Montuenga L.M., Minna J.D., Yokota J., Sanchez-Cespedes M.
A gene-alteration profile of human lung cancer cell lines.
Hum. Mutat. 30:1199-1206(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20557307; DOI=10.1111/j.1349-7006.2010.01622.x; PMCID=PMC11158680
Iwakawa R., Kohno T., Enari M., Kiyono T., Yokota J.
Prevalence of human papillomavirus 16/18/33 infection and p53 mutation in lung adenocarcinoma.
Cancer Sci. 101:1891-1896(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22961666; DOI=10.1158/2159-8290.CD-12-0112; PMCID=PMC3567922
Byers L.A., Wang J., Nilsson M.B., Fujimoto J., Saintigny P., Yordy J.S., Giri U., Peyton M., Fan Y.-H., Diao L.-X., Masrorpour F., Shen L., Liu W.-B., Duchemann B., Tumula P., Bhardwaj V., Welsh J., Weber S., Glisson B.S., Kalhor N., Wistuba I.I., Girard L., Lippman S.M., Mills G.B., Coombes K.R., Weinstein J.N., Minna J.D., Heymach J.V.
Proteomic profiling identifies dysregulated pathways in small cell lung cancer and novel therapeutic targets including PARP1.
Cancer Discov. 2:798-811(2012)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26361996; DOI=10.1016/j.jprot.2015.09.003
Grundner-Culemann K., Dybowski J.N., Klammer M., Tebbe A., Schaab C., Daub H.
Comparative proteome analysis across non-small cell lung cancer cell lines.
J. Proteomics 130:1-10(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=29681454; DOI=10.1016/j.cell.2018.03.028; PMCID=PMC5935540
McMillan E.A., Ryu M.-J., Diep C.H., Mendiratta S., Clemenceau J.R., Vaden R.M., Kim J.-H., Motoyaji T., Covington K.R., Peyton M., Huffman K., Wu X.-F., Girard L., Sung Y., Chen P.-H., Mallipeddi P.L., Lee J.Y., Hanson J., Voruganti S., Yu Y., Park S., Sudderth J., DeSevo C., Muzny D.M., Doddapaneni H., Gazdar A.F., Gibbs R.A., Hwang T.H., Heymach J.V., Wistuba I.I., Coombes K.R., Williams N.S., Wheeler D.A., MacMillan J.B., DeBerardinis R.J., Roth M.G., Posner B.A., Minna J.D., Kim H.S., White M.A.
Chemistry-first approach for nomination of personalized treatment in lung cancer.
Cell 173:864-878.e29(2018)
PubMed=30038707; DOI=10.18632/oncotarget.25642; PMCID=PMC6049873
Du L.-Q., Zhao Z.-Z., Suraokar M.B., Shelton S.S., Ma X.-Y., Hsiao T.-H., Minna J.D., Wistuba I.I., Pertsemlidis A.
LMO1 functions as an oncogene by regulating TTK expression and correlates with neuroendocrine differentiation of lung cancer.
Oncotarget 9:29601-29618(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验该产品被引用文献
"PubMed=10987304
Girard L., Zochbauer-Muller S., Virmani A.K., Gazdar A.F., Minna J.D.
Genome-wide allelotyping of lung cancer identifies new regions of allelic loss, differences between small cell lung cancer and non-small cell lung cancer, and loci clustering.
Cancer Res. 60:4894-4906(2000)
PubMed=11030152; DOI=10.1038/sj.onc.1203815
Modi S., Kubo A., Oie H.K., Coxon A.B., Rehmatulla A., Kaye F.J.
Protein expression of the RB-related gene family and SV40 large T antigen in mesothelioma and lung cancer.
Oncogene 19:4632-4639(2000)
PubMed=11314036; DOI=10.1038/sj.onc.1204211
Forgacs E., Wren J.D., Kamibayashi C., Kondo M., Xu X.L., Markowitz S.D., Tomlinson G.E., Muller C.Y., Gazdar A.F., Garner H.R., Minna J.D.
Searching for microsatellite mutations in coding regions in lung, breast, ovarian and colorectal cancers.
Oncogene 20:1005-1009(2001)
PubMed=11416159; DOI=10.1073/pnas.121616198; PMCID=PMC35459
Masters J.R.W., Thomson J.A., Daly-Burns B., Reid Y.A., Dirks W.G., Packer P., Toji L.H., Ohno T., Tanabe H., Arlett C.F., Kelland L.R., Harrison M., Virmani A.K., Ward T.H., Ayres K.L., Debenham P.G.
Short tandem repeat profiling provides an international reference standard for human cell lines.
Proc. Natl. Acad. Sci. U.S.A. 98:8012-8017(2001)
PubMed=14762065; DOI=10.1101/gr.2012304; PMCID=PMC327104
Bignell G.R., Huang J., Greshock J., Watt S., Butler A.P., West S., Grigorova M., Jones K.W., Wei W., Stratton M.R., Futreal P.A., Weber B., Shapero M.H., Wooster R.
High-resolution analysis of DNA copy number using oligonucleotide microarrays.
Genome Res. 14:287-295(2004)
PubMed=16157194; DOI=10.1016/j.cancergencyto.2005.03.007
Grigorova M., Lyman R.C., Caldas C., Edwards P.A.W.
Chromosome abnormalities in 10 lung cancer cell lines of the NCI-H series analyzed with spectral karyotyping.
Cancer Genet. Cytogenet. 162:1-9(2005)
PubMed=19153074; DOI=10.1093/hmg/ddp034
Medina P.P., Castillo S.D., Blanco S., Sanz-Garcia M., Largo C., Alvarez S., Yokota J., Gonzalez-Neira A., Benitez J., Clevers H.C., Cigudosa J.C., Lazo P.A., Sanchez-Cespedes M.
The SRY-HMG box gene, SOX4, is a target of gene amplification at chromosome 6p in lung cancer.
Hum. Mol. Genet. 18:1343-1352(2009)
PubMed=19472407; DOI=10.1002/humu.21028; PMCID=PMC2900846
Blanco R., Iwakawa R., Tang M.-Y., Kohno T., Angulo B., Pio R., Montuenga L.M., Minna J.D., Yokota J., Sanchez-Cespedes M.
A gene-alteration profile of human lung cancer cell lines.
Hum. Mutat. 30:1199-1206(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20557307; DOI=10.1111/j.1349-7006.2010.01622.x; PMCID=PMC11158680
Iwakawa R., Kohno T., Enari M., Kiyono T., Yokota J.
Prevalence of human papillomavirus 16/18/33 infection and p53 mutation in lung adenocarcinoma.
Cancer Sci. 101:1891-1896(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22961666; DOI=10.1158/2159-8290.CD-12-0112; PMCID=PMC3567922
Byers L.A., Wang J., Nilsson M.B., Fujimoto J., Saintigny P., Yordy J.S., Giri U., Peyton M., Fan Y.-H., Diao L.-X., Masrorpour F., Shen L., Liu W.-B., Duchemann B., Tumula P., Bhardwaj V., Welsh J., Weber S., Glisson B.S., Kalhor N., Wistuba I.I., Girard L., Lippman S.M., Mills G.B., Coombes K.R., Weinstein J.N., Minna J.D., Heymach J.V.
Proteomic profiling identifies dysregulated pathways in small cell lung cancer and novel therapeutic targets including PARP1.
Cancer Discov. 2:798-811(2012)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26361996; DOI=10.1016/j.jprot.2015.09.003
Grundner-Culemann K., Dybowski J.N., Klammer M., Tebbe A., Schaab C., Daub H.
Comparative proteome analysis across non-small cell lung cancer cell lines.
J. Proteomics 130:1-10(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=29681454; DOI=10.1016/j.cell.2018.03.028; PMCID=PMC5935540
McMillan E.A., Ryu M.-J., Diep C.H., Mendiratta S., Clemenceau J.R., Vaden R.M., Kim J.-H., Motoyaji T., Covington K.R., Peyton M., Huffman K., Wu X.-F., Girard L., Sung Y., Chen P.-H., Mallipeddi P.L., Lee J.Y., Hanson J., Voruganti S., Yu Y., Park S., Sudderth J., DeSevo C., Muzny D.M., Doddapaneni H., Gazdar A.F., Gibbs R.A., Hwang T.H., Heymach J.V., Wistuba I.I., Coombes K.R., Williams N.S., Wheeler D.A., MacMillan J.B., DeBerardinis R.J., Roth M.G., Posner B.A., Minna J.D., Kim H.S., White M.A.
Chemistry-first approach for nomination of personalized treatment in lung cancer.
Cell 173:864-878.e29(2018)
PubMed=30038707; DOI=10.18632/oncotarget.25642; PMCID=PMC6049873
Du L.-Q., Zhao Z.-Z., Suraokar M.B., Shelton S.S., Ma X.-Y., Hsiao T.-H., Minna J.D., Wistuba I.I., Pertsemlidis A.
LMO1 functions as an oncogene by regulating TTK expression and correlates with neuroendocrine differentiation of lung cancer.
Oncotarget 9:29601-29618(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"
Girard L., Zochbauer-Muller S., Virmani A.K., Gazdar A.F., Minna J.D.
Genome-wide allelotyping of lung cancer identifies new regions of allelic loss, differences between small cell lung cancer and non-small cell lung cancer, and loci clustering.
Cancer Res. 60:4894-4906(2000)
PubMed=11030152; DOI=10.1038/sj.onc.1203815
Modi S., Kubo A., Oie H.K., Coxon A.B., Rehmatulla A., Kaye F.J.
Protein expression of the RB-related gene family and SV40 large T antigen in mesothelioma and lung cancer.
Oncogene 19:4632-4639(2000)
PubMed=11314036; DOI=10.1038/sj.onc.1204211
Forgacs E., Wren J.D., Kamibayashi C., Kondo M., Xu X.L., Markowitz S.D., Tomlinson G.E., Muller C.Y., Gazdar A.F., Garner H.R., Minna J.D.
Searching for microsatellite mutations in coding regions in lung, breast, ovarian and colorectal cancers.
Oncogene 20:1005-1009(2001)
PubMed=11416159; DOI=10.1073/pnas.121616198; PMCID=PMC35459
Masters J.R.W., Thomson J.A., Daly-Burns B., Reid Y.A., Dirks W.G., Packer P., Toji L.H., Ohno T., Tanabe H., Arlett C.F., Kelland L.R., Harrison M., Virmani A.K., Ward T.H., Ayres K.L., Debenham P.G.
Short tandem repeat profiling provides an international reference standard for human cell lines.
Proc. Natl. Acad. Sci. U.S.A. 98:8012-8017(2001)
PubMed=14762065; DOI=10.1101/gr.2012304; PMCID=PMC327104
Bignell G.R., Huang J., Greshock J., Watt S., Butler A.P., West S., Grigorova M., Jones K.W., Wei W., Stratton M.R., Futreal P.A., Weber B., Shapero M.H., Wooster R.
High-resolution analysis of DNA copy number using oligonucleotide microarrays.
Genome Res. 14:287-295(2004)
PubMed=16157194; DOI=10.1016/j.cancergencyto.2005.03.007
Grigorova M., Lyman R.C., Caldas C., Edwards P.A.W.
Chromosome abnormalities in 10 lung cancer cell lines of the NCI-H series analyzed with spectral karyotyping.
Cancer Genet. Cytogenet. 162:1-9(2005)
PubMed=19153074; DOI=10.1093/hmg/ddp034
Medina P.P., Castillo S.D., Blanco S., Sanz-Garcia M., Largo C., Alvarez S., Yokota J., Gonzalez-Neira A., Benitez J., Clevers H.C., Cigudosa J.C., Lazo P.A., Sanchez-Cespedes M.
The SRY-HMG box gene, SOX4, is a target of gene amplification at chromosome 6p in lung cancer.
Hum. Mol. Genet. 18:1343-1352(2009)
PubMed=19472407; DOI=10.1002/humu.21028; PMCID=PMC2900846
Blanco R., Iwakawa R., Tang M.-Y., Kohno T., Angulo B., Pio R., Montuenga L.M., Minna J.D., Yokota J., Sanchez-Cespedes M.
A gene-alteration profile of human lung cancer cell lines.
Hum. Mutat. 30:1199-1206(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20557307; DOI=10.1111/j.1349-7006.2010.01622.x; PMCID=PMC11158680
Iwakawa R., Kohno T., Enari M., Kiyono T., Yokota J.
Prevalence of human papillomavirus 16/18/33 infection and p53 mutation in lung adenocarcinoma.
Cancer Sci. 101:1891-1896(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22961666; DOI=10.1158/2159-8290.CD-12-0112; PMCID=PMC3567922
Byers L.A., Wang J., Nilsson M.B., Fujimoto J., Saintigny P., Yordy J.S., Giri U., Peyton M., Fan Y.-H., Diao L.-X., Masrorpour F., Shen L., Liu W.-B., Duchemann B., Tumula P., Bhardwaj V., Welsh J., Weber S., Glisson B.S., Kalhor N., Wistuba I.I., Girard L., Lippman S.M., Mills G.B., Coombes K.R., Weinstein J.N., Minna J.D., Heymach J.V.
Proteomic profiling identifies dysregulated pathways in small cell lung cancer and novel therapeutic targets including PARP1.
Cancer Discov. 2:798-811(2012)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26361996; DOI=10.1016/j.jprot.2015.09.003
Grundner-Culemann K., Dybowski J.N., Klammer M., Tebbe A., Schaab C., Daub H.
Comparative proteome analysis across non-small cell lung cancer cell lines.
J. Proteomics 130:1-10(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=29681454; DOI=10.1016/j.cell.2018.03.028; PMCID=PMC5935540
McMillan E.A., Ryu M.-J., Diep C.H., Mendiratta S., Clemenceau J.R., Vaden R.M., Kim J.-H., Motoyaji T., Covington K.R., Peyton M., Huffman K., Wu X.-F., Girard L., Sung Y., Chen P.-H., Mallipeddi P.L., Lee J.Y., Hanson J., Voruganti S., Yu Y., Park S., Sudderth J., DeSevo C., Muzny D.M., Doddapaneni H., Gazdar A.F., Gibbs R.A., Hwang T.H., Heymach J.V., Wistuba I.I., Coombes K.R., Williams N.S., Wheeler D.A., MacMillan J.B., DeBerardinis R.J., Roth M.G., Posner B.A., Minna J.D., Kim H.S., White M.A.
Chemistry-first approach for nomination of personalized treatment in lung cancer.
Cell 173:864-878.e29(2018)
PubMed=30038707; DOI=10.18632/oncotarget.25642; PMCID=PMC6049873
Du L.-Q., Zhao Z.-Z., Suraokar M.B., Shelton S.S., Ma X.-Y., Hsiao T.-H., Minna J.D., Wistuba I.I., Pertsemlidis A.
LMO1 functions as an oncogene by regulating TTK expression and correlates with neuroendocrine differentiation of lung cancer.
Oncotarget 9:29601-29618(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"
 技术资料
技术资料暂无技术资料 索取技术资料
文献支持
NCI-H2126 Cells人肺癌细胞实验室复苏|STR图谱
¥850 - 5500






![NCI-H226[H226]人肺鳞癌传代细胞种子库|送STR图谱](https://img1.dxycdn.com/p/s14/2025/0211/049/8028590892026129881.jpg!wh200)