相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 品系:
详见细胞说明资料
- 细胞类型:
详见细胞说明资料
- 肿瘤类型:
详见细胞说明资料
- 供应商:
上海冠导生物工程有限公司
- 库存:
≥100瓶
- 生长状态:
详见细胞说明资料
- 年限:
详见细胞说明资料
- 运输方式:
常温运输【复苏细胞】或干冰运输【冻存细胞】
- 器官来源:
详见细胞说明资料
- 是否是肿瘤细胞:
详见细胞说明资料
- 细胞形态:
详见细胞说明资料
- 免疫类型:
详见细胞说明资料
- 物种来源:
详见细胞说明资料
- 相关疾病:
详见细胞说明资料
- 组织来源:
详见细胞说明资料
- 英文名:
JVM-2人套细胞淋巴瘤细胞系
- 规格:
1*10(6)Cellls/瓶
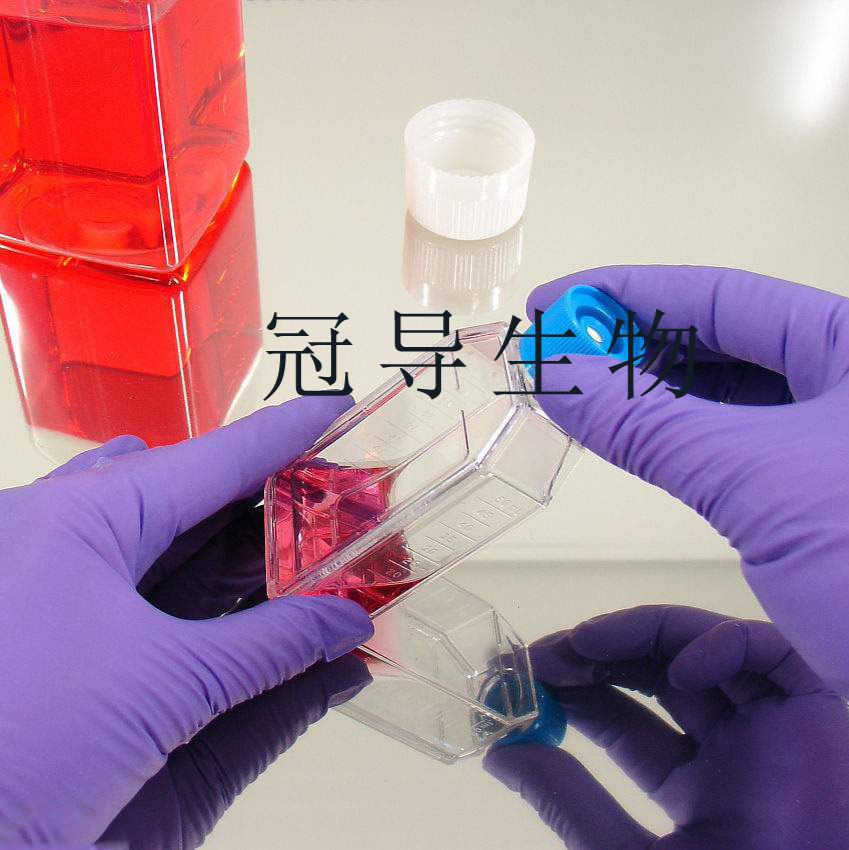
"JVM-2人套细胞淋巴瘤细胞系
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:悬浮生长
换液周期:每周2-3次
公司细胞库冻存并保种有2300多种各类细胞系。针对每种不同的细胞系,公司摸索并积累了大量细胞培养条件和YOU化参数,为您的细胞实验保驾护航。细胞培养是生命科学研究中Zui基础、也是Zui常用的实验手段。从冻存→复苏→传代→实验,在每一次细胞培养的过程中我们都是精心呵护,小心培养,心里都是默默承诺:我要护你一世周全!可是可是……无数次细胞都因污染而离我而去,投入的不只是我们貌美如花的青春,还有那一去不复返的经费!在细胞生物学,生物化学,免疫学,神经生物学,病理学……只要涉及细胞的研究领域,都面临着支原体污染的威胁。在众多污染物中,也就属支原体Zui狡猾了,他Zui善于隐藏自己,因为被支原体污染的细胞不会马上死亡,但会改变细胞的新陈代谢,甚至会改变细胞的基因表达谱,我们就这样被欺骗了无数次,谁让我们就是个看“颜值”的花痴了。可是我们通过被虐了几百次的经验中总结出来,当你的细胞出现了这些症状时:生长速率改变;活性减弱;形态改变;染色体畸变;转染效率显著降低;细胞复苏后存活率降低……。那你就得千万小心了,也许你的细胞已经被支原体欺凌了!
JVM-2人套细胞淋巴瘤细胞系
背景信息:是从一名63岁白人女性的外周血中分离出的淋巴母细胞。
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
LLC-PK(1) Cells;背景说明:肾;自发永生;Hampshire;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:Murine Leydig Tumor Cell line-1 Cells、mRTEC Cells、HLMVEC Cells
SKES1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:5传代;每周换液2-3次;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:上皮样;相关产品有:Madison 109 Cells、Hs819.T Cells、HONE1 Cells
NCI H548 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:HCC 38 Cells、MDAMB175VII Cells、AQ-Mel Cells
CL34 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:HuP-T4 Cells、RCCJF Cells、OCI AML3 Cells
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
JVM-2人套细胞淋巴瘤细胞系
因为细胞生长的时候肯定是要相互联系,大部分的细胞都是要成片生长的,尤其是对于贴壁细胞,单个细胞贴壁之后长着长着就长到一片去了。但是如果是成片的细胞抱团的话,传代后细胞是不能贴壁的,这样抱团的细胞就会死亡。所以,消化传代的时候一定要将细胞消化成单个单个的独立状态,这个啊是非常考究你的功夫的!细胞一旦脱落入悬里你很难再将他吹打成单个,因为他没有受力点了。很难找到受力点让你对他吹打的力分散开细胞。所以必须要在细胞未脱落之前将其吹打开,受力点就是细胞贴壁的地方。这样你就必须要严格控制HAO消化的程度啊,这和大师做饭掌握HAO火候是一样的道理啊。既要消化到容易吹打下来,又不能太过,一吹就整片脱落,要单个单个地往下掉。所以我才要分批分次地加胰酶消化就是为了保证不同生长状态贴壁程度的细胞能够都维持在HAO的受力点分散开。这个是很重要的一点。尤其是对于某些细胞这一点就是他活去死的致命点。像Caco-2细胞就是如此。另外关于传代很重要一点就是一定不能等到细胞长满的时候才去消化传代。要在细胞长到70%左右的时候就要传代了,一旦发现细胞生长中已经有层生长的时候就要立即进行消化传代。不能再拖了。关于换传代时候洗瓶的问题。对于用DMEM培养基培养的细胞,你在洗的时候如果是用无血清1640去洗细胞在随即后的几次中会比用DMEM洗的长的要HAO。同样,用1640培养的也有这个现象。如果不嫌麻烦的话,可以用PBS来洗,洗的效果和用无血清培养基洗的效果基本上是一样的,对于有些细胞会更胜一筹。洗的目的主要是洗去培养瓶里残留的血清,防止它对胰酶的影响。这样能够保证胰酶的消化能力YOU先,是很关键的一点。先补充这么多,还会后续更新。再补充一点:传代时PBS洗是很重要的一步,Zui近发现,用PBS就可以把细胞消化开来。加入PBS放置10-15分钟,有些需要更长的时间,等到细胞一个个分离开来的时候,就可以直接吹打下来,但是和胰酶消化一样,要控制HAO时间,不能时间太过了,要不然损伤细胞,细胞不容易成活,状态容易不HAO。这个方法对于某些细胞是很管用的。有些细胞用胰酶消化不容易消化成单个的时候,或者临时没有胰酶了,可以选用此方法。不过一定要试试,看是否适合你的细胞。
HK-2 [Human kidney] Cells;背景说明:该细胞属源于正常肾的近曲小管细胞,通过导入HPV-16 E6/E7基因而获得永生化。将含有HPV-16 E6/E7基因的重组的逆转录病毒载体pLXSN 16 E6/E7转染外生包装细胞Psi-2,Psi-2细胞产生的病毒再去感染兼嗜性包装细胞系PA317,最后将PA317产生的病毒颗粒导入正常的肾皮质近曲小管细胞。尽管pLXSN 16 E6/E7中含有新霉素抗性,但未用G418筛选转导克隆。Southern和FISH分析显示HK-2细胞来源于单克隆。PCR检测证实HK-2细胞基因组中含有E6/E7基因。;传代方法:1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:University of Arizona Cell Culture-812 Cells、KBM-7/Hap8 Cells、H-522 Cells
HL7702 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:HPAFII Cells、MCAEC Cells、H-1373 Cells
SUDHL-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:MX-1 Cells、NCI-H929 Cells、SKml2 Cells
物种来源:人源、鼠源等其它物种来源
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
形态特性:淋巴母细胞样
细胞培养实验基本操作:①进无菌室前要彻底洗手,按规定穿隔离衣。开始操作前要用75%酒精棉球擦手、擦瓶口和烧灼瓶口。②操作者动作要轻,安装吸管帽、打开或封闭瓶口等操作应在火焰近处并经过烧灼进行。但要注意,金属器械不能在火焰中长时间烧灼,以防退火;烧过的器械要冷却后才能使用;已吸过培养的吸管不能再用火焰烧灼,因残留在吸管内的培养成分如蛋白质等烧焦后会产生有害物质,吸管再用时会将其带到培养中;胶塞、橡皮乳头及塑料的细胞培养用品过火焰是也不能时间太长,以免烧焦产生有毒气体,危害培养细胞,同时塑料细胞培养用品也会产生变形影响使用。③使用培养前不宜过早开瓶,开瓶后的培养应保持斜位,避免直立,以防止下落细菌的污染。不再使用的培养应立即封闭瓶口,培养的细胞在处理之前勿过早暴露在空气中。④操作时尽量不要谈话,咳嗽以防止来自唾沫和呼出的气流所造成的污染。⑤吸取培养、细胞悬时,应专管专用,一旦发现吸管口接触了手和其他污染物品应弃去,以防止污染扩大或造成培养物之间的交叉污染。⑥操作完毕后应整理HAO工作台面,用消毒水浸泡的纱布擦拭台面;防止细胞交叉污染:所有从别处转来的或是自己所建的细胞系都要早期留有的充足的冻存储备,一旦怀疑发生交叉污染,可做细胞遗传学方面的鉴定,如发现原有的细胞遗传物发生改变,可以复苏早期冻存的细胞使用。重要细胞系的传代工作应由两人独立进行;无菌室的彻底消毒①全面彻底擦洗无菌室。使用前应稀释即配即用。和酒精相比,Zui大的YOU点是便宜,而且可以稀释50倍用,而同样量的酒精价格可能是的几十倍。②甲醛熏蒸法:甲醛是一种广谱灭菌剂菌,其水溶和气体对各种细菌、芽孢及真菌等微生物均有杀灭作用。甲醛价廉,熏蒸消毒时不损坏衣服、家具、皮革、橡胶等。市售的甲醛水溶中一般含37-40%的甲醛。
PA317 Cells;背景说明:胚胎;成纤维细胞;NIH Swiss;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:R 2 C Cells、Panc04.03 Cells、H3396 Cells
RPMI.8226 Cells;背景说明:来源于一位61岁的男性浆细胞瘤患者;可产生免疫球蛋白轻链,未检测到重链。;传代方法:按1:2传代,5-6小时可以看到细胞分裂;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:NCIH250 Cells、KYSE 50 Cells、KMB 17 Cells
NCIH2591 Cells;背景说明:上皮样间皮瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:KS-SLK Cells、Mouse Colon 38 Cells、Meat Animal Research Center-145 Cells
Menschliche Und Tierische Zellkulture-1 Cells;背景说明:骨髓增生异常综合征;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:NCIH1838 Cells、H157 Cells、SK 1 Cells
J-774A.1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:CEM-CCRF Cells、OCI-Ly 19 Cells、PC12(高分化) Cells
NCIH1693 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:C4-2 B Cells、Metastatic Variant-522 Cells、COLO-320DM Cells
Cloudman M3 Cells;背景说明:黑色素瘤;雄性;DBA;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:Dami Cells、Hs600T Cells、H1650 Cells
COS1 Cells;背景说明:该细胞源自CV-1细胞株,经转染编码野生型T抗原、起始点缺陷突变的SV40得到;细胞中整合有SV40基因组完整早期区段的单个拷贝。该细胞表达T抗原,适用于需要SV40T抗原表达的载体的转染;保留SV40溶细胞性生长的特性;支持40℃时温度敏感性A209病毒的复制;支持早期区段缺失的SV40纯体的复制。因含有SV40的DNA序列,该细胞需要在2级生物安全柜中操作。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:Pa14C Cells、H-1373 Cells、YT Cells
MDA-1386 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:786-0 Cells、VMM-5A Cells、SK BR 03 Cells
DU-4475 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2—3次;生长特性:悬浮,多细胞聚集;形态特性:上皮细胞样;相关产品有:HCC1359 Cells、CCD 841 CoTr Cells、HSKMC Cells
Ontario Cancer Institute-Acute Myeloid Leukemia-2 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:HFB Cells、PL-5 Cells、MCF 7B Cells
HS-695T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:HCV29 Cells、SCC 25 Cells、L M (TK-) Cells
CCC-HHM-2 Cells;背景说明:心肌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:CL40 Cells、NCIH2171 Cells、CCD19-Lu Cells
P3 NS1 Ag4 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:Hs 706.T Cells、National Medical Center-Glioma 1 Cells、RCC-10 Cells
SKNFI Cells;背景说明:详见相关文献介绍;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Acanthosis Nigricans 3rd attempt-CArcinoma Cells、NCI-H1155 Cells、WBF344 Cells
JVM-2人套细胞淋巴瘤细胞系
SW260 Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:OVCAR-10 Cells、PLMVEC Cells、PG-4(S+L-) Cells
OCI AML5 Cells;背景说明:急性髓系白血病细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:H-1693 Cells、Hs-688A-T Cells、X63Ag8.653 Cells
93 BLCL Cells(拥有STR基因鉴定图谱)
Abcam MCF-7 MALT1 KO Cells(拥有STR基因鉴定图谱)
AtT-20/HNP Cells(拥有STR基因鉴定图谱)
BayGenomics ES cell line RRO203 Cells(拥有STR基因鉴定图谱)
BayGenomics ES cell line YHB279 Cells(拥有STR基因鉴定图谱)
CCRF-CEM C7/HDR Cells(拥有STR基因鉴定图谱)
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
DA01115 Cells(拥有STR基因鉴定图谱)
DD2919 Cells(拥有STR基因鉴定图谱)
GM02227 Cells(拥有STR基因鉴定图谱)
NRK-52E Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Wistar Institute, Susan Hayflick Cells、Mia PACA 2 Cells、PC9 Cells
HCC2157 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2—3次;生长特性:悬浮生长;形态特性:上皮样;相关产品有:IMR-32 Cells、Panc10.05 Cells、OV433 Cells
8305C Cells;背景说明:详见相关文献介绍;传代方法:1:6传代;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:MKN28 Cells、NCI-H719 Cells、SK-MEL2 Cells
HEK293A Cells;背景说明:胚肾;腺病毒包装;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:NK62a Cells、Panc 4.03 Cells、L-540 Cells
L132 Cells;背景说明:胚肺;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:SW 579 Cells、UCLA SO M14 Cells、SK-LMS-1 Cells
CTV1 Cells;背景说明:急性T淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:Intestinal Epithelioid Cell line No. 6 Cells、NEC Cells、PANC.1 Cells
Hs274T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代,每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:NCI-H1437 Cells、CAOV3 Cells、PC-9/S1 Cells
CT26WT Cells;背景说明:CT26细胞是被N-亚硝基-N-甲基脲烷(NNMU)诱导得到的未分化的小鼠结肠癌细胞,该细胞的一个克隆形成的细胞系被命名为CT26.WT。CT26.WT被逆转录病毒载体LXSN稳定转化形成了一个致死性的亚克隆CT26.CL25,这一病毒载体含有lacZ基因、编码肿瘤相关抗原(TAA)和beta半乳糖苷酶。CT26.WT和CT26.CL25细胞在小鼠中生长速度和致死率都很相似,不同的是CT26.CL25细胞可以表达肿瘤相关抗原和beta半乳糖苷酶,因此这两株细胞可以联合用于免疫治疗和宿主免疫反应的研究。 ;传代方法:1:4-1:10传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:RAW-264.7 Cells、hRMECs Cells、P3JHR-1 Cells
Ontario Cancer Institute-Acute Myeloid Leukemia-4 Cells;背景说明:急性髓系白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:Hs 294T Cells、U-118MG Cells、HNE-1 Cells
TI-73 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:Hep 3B2.1-7 Cells、KS-SLK Cells、P3J.HR1K Cells
Wien133 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:CHL-CL-11 Cells、BALL1 Cells、LTEP-a2 Cells
Huh-7.5.1 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:L-cells Cells、NPA87 Cells、SMA-560 Cells
MHHCALL2 Cells;背景说明:急性B淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:SK-MEL-24 Cells、Mouse Forestomach Carcinoma Cells、WM35 Cells
47N9-11 Cells(拥有STR基因鉴定图谱)
NCIH196 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明;相关产品有:YES 2 Cells、AZ521 Cells、ZR75-1 Cells
SL-29 Cells;背景说明:胚成纤维细胞; White Leghorn SPAFAS;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:MB39 Cells、LED-WiDr Cells、E0771 Cells
HEK-293FT Cells;背景说明:该细胞稳定表达SV40大T抗原,并且促进最适病毒产物的产生。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:圆形;相关产品有:B16F10 Cells、Jiyoye(P-2003) Cells、High 5 Cells
Colo320 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:淋巴母细胞;相关产品有:PLC/PRF/5 Cells、RBL-I Cells、TERT-RPE1 Cells
H-295R Cells;背景说明:肾上腺皮质癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:SNU-182 Cells、HCMEC Cells、UCH1 Cells
Panc 08.13 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:AZ 521 Cells、HKF Cells、HSAS1 Cells
207 Cells;背景说明:儿童急性髓系白血病;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:CHOS Cells、Panc-03.27 Cells、HEC1-A Cells
SK-N-MC Cells;背景说明:这株细胞与HTB-11都是神经源的。1971年9月分离得到SK-N-MC后,发现它有中性多巴胺-β-羟化酶活性,也有细胞内儿茶胺,用甲醛可以诱导出荧光。;传代方法:1:6-1:12传代,每周2-3次换液。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Panc-02 Cells、TE354T Cells、MDA-MB231 Cells
GM11041 Cells(拥有STR基因鉴定图谱)
HAP1 CLK2 (-) 1 Cells(拥有STR基因鉴定图谱)
COLO_320DM Cells;背景说明:该细胞可产生5-羟色胺、去甲、、ACTH和甲状旁腺素。角蛋白、波形蛋白弱阳性。培养条件: RPMI 1640 10%FBS;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮+贴壁;形态特性:淋巴细胞;相关产品有:HANK-1 Cells、SUDHL-8 Cells、H22 Cells
HG04193 Cells(拥有STR基因鉴定图谱)
iMOSEC-6/BCL-2 Cells(拥有STR基因鉴定图谱)
LUTC-12 Cells(拥有STR基因鉴定图谱)
ND03973 Cells(拥有STR基因鉴定图谱)
PDL-L2 Cells(拥有STR基因鉴定图谱)
UCLAi089-A Cells(拥有STR基因鉴定图谱)
ZP115 Cells(拥有STR基因鉴定图谱)
HCC954 Cells(拥有STR基因鉴定图谱)
MAC1 Cells;背景说明:皮肤T淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:MuM-2B Cells、HCC-1428 Cells、SACC-LM Cells
KNS-81 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H-1876 Cells、RS1 Cells、BRL 3A Cells
HEL 92.1.7 Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:成淋巴细胞;相关产品有:MA104 Cells、MSTO-211 H Cells、T-24 Cells
NCIH1435 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,每周换液2-3次;生长特性:贴壁生长,松散;形态特性:详见产品说明;相关产品有:CHG-5 Cells、293-GP Cells、JIII Cells
NTERA2 Cells;背景说明:畸胎瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:NBL-4 Cells、NK-92 MI Cells、SW 1353 Cells
MESSA-DX5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:8传代;每周2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞样 ;相关产品有:GLC-15 Cells、PLA801D Cells、OCI-AML2 Cells
KU1919 Cells;背景说明:膀胱癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:MC9 Cells、NMC-G1 Cells、Panc 04.03 Cells
OCI/AML-5 Cells;背景说明:急性髓系白血病细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:EC-GI-10 Cells、253J-Bladder-V Cells、MHCC97-H Cells
NHLF Cells;背景说明:肺;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:149PT Cells、A-1847 Cells、Anglne Cells
TOV21G Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Ontario Cancer Institute-Acute Myeloid Leukemia-4 Cells、PANC-1 Cells、SNU398 Cells
Metastatic Variant 522 Cells;背景说明:肺腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:ZR-75-30 Cells、MV3 Cells、MKN-74 Cells
Mevo Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代,2-3天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:Sci-1 Cells、BxPC-3 Cells、MDA2B Cells
OUMS-23 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:210RCY3-Ag1.2.3 Cells、SF 763 Cells、COLO206F Cells
NCI-H1573 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,每周2-3次。;生长特性:贴壁生长;形态特性:详见产品说明;相关产品有:CCD19-Lu Cells、CNE1-LUC Cells、Scott Cells
RAW2647 Cells;背景说明:单核巨噬细胞白血病;雄性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:P30/OHK Cells、0V-1063 Cells、SNU520 Cells
SCVIi077-A Cells(拥有STR基因鉴定图谱)
MDA-MB-435-S Cells;背景说明:MDA-MB-435S是一种纺锤形的细胞,1976年由其亲本(435)中筛选得到。435是从31岁的转移性乳腺导管腺癌女性患者胸水中分离得到。当用荧光染料对微管蛋白进行染色时亲本细胞显现散布特征(II型)。最近通过cDNA阵列研究表明,亲本(MDA-MB-435)可归入黑素瘤起源。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:纺锤形;相关产品有:Kit225-K6 Cells、NTERA2D1 Cells、Hs 737.T Cells
NBLS Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:CEF Cells、RT-BM 1 Cells、RK 13 Cells
SKO007 Cells;背景说明:骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:L1210 Cells、BL-6 Cells、HNE-3 Cells
Panc-1-P Cells;背景说明:这株人胰腺癌细胞株源自于胰腺癌导管细胞,其倍增时间为52小时。染色体研究表明,该细胞染色体众数为63,包括3个独特标记的染色体和1个小环状染色体。该细胞的生长可被1unit/ml的左旋天冬酰胺酶抑制;能在软琼脂上生长;能在裸鼠上成瘤。;传代方法:1:2-1:4传代;每周2-3次。;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:NCI-H460 Cells、GM03570E Cells、PLA801-95D Cells
BT-20 Cells;背景说明:该细胞1958年由E.Y. Lasfargues 和 L. Ozzello 建系,源自一位74岁白人女性的乳腺癌组织。该细胞表达WNT3和WNT78。TNF alpha抑制该细胞生长。该细胞雌激素受体阴性,但表达5'外显子缺失的雌激素mRNA。;传代方法:1:2—1:4传代,2—3天换液一次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Ramos (RA 1) Cells、H-1694 Cells、MC-4 Cells
RT4P Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,每周2-3次。;生长特性:贴壁生长;形态特性:多角 ;相关产品有:Hep 3B2 Cells、DMS153 Cells、FRhK-4 Cells
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
C17 Cells;背景说明:神经干细胞;C57BL/6 x CD-1;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:MDA-453 Cells、SK-MEL-31 Cells、Tn5B1-4 Cells
MCF/Adr Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:BE(2)M-17 Cells、BEL-7404 Cells、CCC-ESF-1 Cells
COLO-320-HSR Cells;背景说明:该细胞1984年建系,源自一位33岁患有大肠腺癌男性经5-fu治疗后的腹水。;传代方法:1:2传代。3天内可长满。;生长特性:半贴壁生长;形态特性:详见产品说明;相关产品有:OVCA 433 Cells、TERT-RPE1 Cells、RPTEC/TERT1 Cells
Hs852 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:FDCP1 Cells、OV-90 Cells、NALM 6 Cells
WM-115 Cells;背景说明:黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:NCIH1944 Cells、V-79 Cells、Pt K2 Cells
L-cells Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:UPCI-SCC-090 Cells、IMR32 Cells、NK-10A Cells
Jijoye Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:MDA-157 Cells、NCI-H220 Cells、BT 549 Cells
JVM-2人套细胞淋巴瘤细胞系
MKN 45 Cells;背景说明:该细胞系由S Akiyama建立,源于一位35岁患有印戒细胞癌的女性的胃淋巴结。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁+悬浮;形态特性:淋巴母细胞;相关产品有:BOWES Cells、SF 295 Cells、ESC-410 Cells
BayGenomics ES cell line RRJ207 Cells(拥有STR基因鉴定图谱)
BayGenomics ES cell line XK166 Cells(拥有STR基因鉴定图谱)
FM3A AC1 Cells(拥有STR基因鉴定图谱)
NKI/beteb Cells(拥有STR基因鉴定图谱)
WEGLI-9 Cells(拥有STR基因鉴定图谱)
MCF10A-EGFR1m-3 Cells(拥有STR基因鉴定图谱)
" "DOI=10.1007/0-306-46861-1_11
Ali-Osman F.
Brain tumors.
(In book chapter) Human cell culture. Vol. 2. Cancer cell lines part 2; Masters J.R.W., Palsson B.O. (eds.); pp.167-184; Kluwer Academic Publishers; New York; USA (1999)
PubMed=10416987; DOI=10.1111/j.1750-3639.1999.tb00536.x; PMCID=PMC8098486
Ishii N., Maier D., Merlo A., Tada M., Sawamura Y., Diserens A.-C., Van Meir E.G.
Frequent co-alterations of TP53, p16/CDKN2A, p14ARF, PTEN tumor suppressor genes in human glioma cell lines.
Brain Pathol. 9:469-479(1999)
PubMed=11414198; DOI=10.1007/s004320000207
Lahm H., Andre S., Hoeflich A., Fischer J.R., Sordat B., Kaltner H., Wolf E., Gabius H.-J.
Comprehensive galectin fingerprinting in a panel of 61 human tumor cell lines by RT-PCR and its implications for diagnostic and therapeutic procedures.
J. Cancer Res. Clin. Oncol. 127:375-386(2001)
PubMed=21406405; DOI=10.1158/0008-5472.CAN-10-3112
Grzmil M., Morin P. Jr., Lino M.M., Merlo A., Frank S., Wang Y.-H., Moncayo G., Hemmings B.A.
MAP kinase-interacting kinase 1 regulates SMAD2-dependent TGF-beta signaling pathway in human glioblastoma.
Cancer Res. 71:2392-2402(2011)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22570425; DOI=10.1093/neuonc/nos072; PMCID=PMC3367844
Bady P., Diserens A.-C., Castella V., Kalt S., Heinimann K., Hamou M.-F., Delorenzi M., Hegi M.E.
DNA fingerprinting of glioma cell lines and considerations on similarity measurements.
Neuro-oncol. 14:701-711(2012)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:悬浮生长
换液周期:每周2-3次
公司细胞库冻存并保种有2300多种各类细胞系。针对每种不同的细胞系,公司摸索并积累了大量细胞培养条件和YOU化参数,为您的细胞实验保驾护航。细胞培养是生命科学研究中Zui基础、也是Zui常用的实验手段。从冻存→复苏→传代→实验,在每一次细胞培养的过程中我们都是精心呵护,小心培养,心里都是默默承诺:我要护你一世周全!可是可是……无数次细胞都因污染而离我而去,投入的不只是我们貌美如花的青春,还有那一去不复返的经费!在细胞生物学,生物化学,免疫学,神经生物学,病理学……只要涉及细胞的研究领域,都面临着支原体污染的威胁。在众多污染物中,也就属支原体Zui狡猾了,他Zui善于隐藏自己,因为被支原体污染的细胞不会马上死亡,但会改变细胞的新陈代谢,甚至会改变细胞的基因表达谱,我们就这样被欺骗了无数次,谁让我们就是个看“颜值”的花痴了。可是我们通过被虐了几百次的经验中总结出来,当你的细胞出现了这些症状时:生长速率改变;活性减弱;形态改变;染色体畸变;转染效率显著降低;细胞复苏后存活率降低……。那你就得千万小心了,也许你的细胞已经被支原体欺凌了!
JVM-2人套细胞淋巴瘤细胞系
背景信息:是从一名63岁白人女性的外周血中分离出的淋巴母细胞。
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
LLC-PK(1) Cells;背景说明:肾;自发永生;Hampshire;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:Murine Leydig Tumor Cell line-1 Cells、mRTEC Cells、HLMVEC Cells
SKES1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:5传代;每周换液2-3次;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:上皮样;相关产品有:Madison 109 Cells、Hs819.T Cells、HONE1 Cells
NCI H548 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:HCC 38 Cells、MDAMB175VII Cells、AQ-Mel Cells
CL34 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:HuP-T4 Cells、RCCJF Cells、OCI AML3 Cells
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
JVM-2人套细胞淋巴瘤细胞系
因为细胞生长的时候肯定是要相互联系,大部分的细胞都是要成片生长的,尤其是对于贴壁细胞,单个细胞贴壁之后长着长着就长到一片去了。但是如果是成片的细胞抱团的话,传代后细胞是不能贴壁的,这样抱团的细胞就会死亡。所以,消化传代的时候一定要将细胞消化成单个单个的独立状态,这个啊是非常考究你的功夫的!细胞一旦脱落入悬里你很难再将他吹打成单个,因为他没有受力点了。很难找到受力点让你对他吹打的力分散开细胞。所以必须要在细胞未脱落之前将其吹打开,受力点就是细胞贴壁的地方。这样你就必须要严格控制HAO消化的程度啊,这和大师做饭掌握HAO火候是一样的道理啊。既要消化到容易吹打下来,又不能太过,一吹就整片脱落,要单个单个地往下掉。所以我才要分批分次地加胰酶消化就是为了保证不同生长状态贴壁程度的细胞能够都维持在HAO的受力点分散开。这个是很重要的一点。尤其是对于某些细胞这一点就是他活去死的致命点。像Caco-2细胞就是如此。另外关于传代很重要一点就是一定不能等到细胞长满的时候才去消化传代。要在细胞长到70%左右的时候就要传代了,一旦发现细胞生长中已经有层生长的时候就要立即进行消化传代。不能再拖了。关于换传代时候洗瓶的问题。对于用DMEM培养基培养的细胞,你在洗的时候如果是用无血清1640去洗细胞在随即后的几次中会比用DMEM洗的长的要HAO。同样,用1640培养的也有这个现象。如果不嫌麻烦的话,可以用PBS来洗,洗的效果和用无血清培养基洗的效果基本上是一样的,对于有些细胞会更胜一筹。洗的目的主要是洗去培养瓶里残留的血清,防止它对胰酶的影响。这样能够保证胰酶的消化能力YOU先,是很关键的一点。先补充这么多,还会后续更新。再补充一点:传代时PBS洗是很重要的一步,Zui近发现,用PBS就可以把细胞消化开来。加入PBS放置10-15分钟,有些需要更长的时间,等到细胞一个个分离开来的时候,就可以直接吹打下来,但是和胰酶消化一样,要控制HAO时间,不能时间太过了,要不然损伤细胞,细胞不容易成活,状态容易不HAO。这个方法对于某些细胞是很管用的。有些细胞用胰酶消化不容易消化成单个的时候,或者临时没有胰酶了,可以选用此方法。不过一定要试试,看是否适合你的细胞。
HK-2 [Human kidney] Cells;背景说明:该细胞属源于正常肾的近曲小管细胞,通过导入HPV-16 E6/E7基因而获得永生化。将含有HPV-16 E6/E7基因的重组的逆转录病毒载体pLXSN 16 E6/E7转染外生包装细胞Psi-2,Psi-2细胞产生的病毒再去感染兼嗜性包装细胞系PA317,最后将PA317产生的病毒颗粒导入正常的肾皮质近曲小管细胞。尽管pLXSN 16 E6/E7中含有新霉素抗性,但未用G418筛选转导克隆。Southern和FISH分析显示HK-2细胞来源于单克隆。PCR检测证实HK-2细胞基因组中含有E6/E7基因。;传代方法:1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:University of Arizona Cell Culture-812 Cells、KBM-7/Hap8 Cells、H-522 Cells
HL7702 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:HPAFII Cells、MCAEC Cells、H-1373 Cells
SUDHL-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:MX-1 Cells、NCI-H929 Cells、SKml2 Cells
物种来源:人源、鼠源等其它物种来源
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
形态特性:淋巴母细胞样
细胞培养实验基本操作:①进无菌室前要彻底洗手,按规定穿隔离衣。开始操作前要用75%酒精棉球擦手、擦瓶口和烧灼瓶口。②操作者动作要轻,安装吸管帽、打开或封闭瓶口等操作应在火焰近处并经过烧灼进行。但要注意,金属器械不能在火焰中长时间烧灼,以防退火;烧过的器械要冷却后才能使用;已吸过培养的吸管不能再用火焰烧灼,因残留在吸管内的培养成分如蛋白质等烧焦后会产生有害物质,吸管再用时会将其带到培养中;胶塞、橡皮乳头及塑料的细胞培养用品过火焰是也不能时间太长,以免烧焦产生有毒气体,危害培养细胞,同时塑料细胞培养用品也会产生变形影响使用。③使用培养前不宜过早开瓶,开瓶后的培养应保持斜位,避免直立,以防止下落细菌的污染。不再使用的培养应立即封闭瓶口,培养的细胞在处理之前勿过早暴露在空气中。④操作时尽量不要谈话,咳嗽以防止来自唾沫和呼出的气流所造成的污染。⑤吸取培养、细胞悬时,应专管专用,一旦发现吸管口接触了手和其他污染物品应弃去,以防止污染扩大或造成培养物之间的交叉污染。⑥操作完毕后应整理HAO工作台面,用消毒水浸泡的纱布擦拭台面;防止细胞交叉污染:所有从别处转来的或是自己所建的细胞系都要早期留有的充足的冻存储备,一旦怀疑发生交叉污染,可做细胞遗传学方面的鉴定,如发现原有的细胞遗传物发生改变,可以复苏早期冻存的细胞使用。重要细胞系的传代工作应由两人独立进行;无菌室的彻底消毒①全面彻底擦洗无菌室。使用前应稀释即配即用。和酒精相比,Zui大的YOU点是便宜,而且可以稀释50倍用,而同样量的酒精价格可能是的几十倍。②甲醛熏蒸法:甲醛是一种广谱灭菌剂菌,其水溶和气体对各种细菌、芽孢及真菌等微生物均有杀灭作用。甲醛价廉,熏蒸消毒时不损坏衣服、家具、皮革、橡胶等。市售的甲醛水溶中一般含37-40%的甲醛。
PA317 Cells;背景说明:胚胎;成纤维细胞;NIH Swiss;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:R 2 C Cells、Panc04.03 Cells、H3396 Cells
RPMI.8226 Cells;背景说明:来源于一位61岁的男性浆细胞瘤患者;可产生免疫球蛋白轻链,未检测到重链。;传代方法:按1:2传代,5-6小时可以看到细胞分裂;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:NCIH250 Cells、KYSE 50 Cells、KMB 17 Cells
NCIH2591 Cells;背景说明:上皮样间皮瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:KS-SLK Cells、Mouse Colon 38 Cells、Meat Animal Research Center-145 Cells
Menschliche Und Tierische Zellkulture-1 Cells;背景说明:骨髓增生异常综合征;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:NCIH1838 Cells、H157 Cells、SK 1 Cells
J-774A.1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:CEM-CCRF Cells、OCI-Ly 19 Cells、PC12(高分化) Cells
NCIH1693 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:C4-2 B Cells、Metastatic Variant-522 Cells、COLO-320DM Cells
Cloudman M3 Cells;背景说明:黑色素瘤;雄性;DBA;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:Dami Cells、Hs600T Cells、H1650 Cells
COS1 Cells;背景说明:该细胞源自CV-1细胞株,经转染编码野生型T抗原、起始点缺陷突变的SV40得到;细胞中整合有SV40基因组完整早期区段的单个拷贝。该细胞表达T抗原,适用于需要SV40T抗原表达的载体的转染;保留SV40溶细胞性生长的特性;支持40℃时温度敏感性A209病毒的复制;支持早期区段缺失的SV40纯体的复制。因含有SV40的DNA序列,该细胞需要在2级生物安全柜中操作。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:Pa14C Cells、H-1373 Cells、YT Cells
MDA-1386 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:786-0 Cells、VMM-5A Cells、SK BR 03 Cells
DU-4475 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2—3次;生长特性:悬浮,多细胞聚集;形态特性:上皮细胞样;相关产品有:HCC1359 Cells、CCD 841 CoTr Cells、HSKMC Cells
Ontario Cancer Institute-Acute Myeloid Leukemia-2 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:HFB Cells、PL-5 Cells、MCF 7B Cells
HS-695T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:HCV29 Cells、SCC 25 Cells、L M (TK-) Cells
CCC-HHM-2 Cells;背景说明:心肌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:CL40 Cells、NCIH2171 Cells、CCD19-Lu Cells
P3 NS1 Ag4 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:Hs 706.T Cells、National Medical Center-Glioma 1 Cells、RCC-10 Cells
SKNFI Cells;背景说明:详见相关文献介绍;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Acanthosis Nigricans 3rd attempt-CArcinoma Cells、NCI-H1155 Cells、WBF344 Cells
JVM-2人套细胞淋巴瘤细胞系
SW260 Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:OVCAR-10 Cells、PLMVEC Cells、PG-4(S+L-) Cells
OCI AML5 Cells;背景说明:急性髓系白血病细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:H-1693 Cells、Hs-688A-T Cells、X63Ag8.653 Cells
93 BLCL Cells(拥有STR基因鉴定图谱)
Abcam MCF-7 MALT1 KO Cells(拥有STR基因鉴定图谱)
AtT-20/HNP Cells(拥有STR基因鉴定图谱)
BayGenomics ES cell line RRO203 Cells(拥有STR基因鉴定图谱)
BayGenomics ES cell line YHB279 Cells(拥有STR基因鉴定图谱)
CCRF-CEM C7/HDR Cells(拥有STR基因鉴定图谱)
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
DA01115 Cells(拥有STR基因鉴定图谱)
DD2919 Cells(拥有STR基因鉴定图谱)
GM02227 Cells(拥有STR基因鉴定图谱)
NRK-52E Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Wistar Institute, Susan Hayflick Cells、Mia PACA 2 Cells、PC9 Cells
HCC2157 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2—3次;生长特性:悬浮生长;形态特性:上皮样;相关产品有:IMR-32 Cells、Panc10.05 Cells、OV433 Cells
8305C Cells;背景说明:详见相关文献介绍;传代方法:1:6传代;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:MKN28 Cells、NCI-H719 Cells、SK-MEL2 Cells
HEK293A Cells;背景说明:胚肾;腺病毒包装;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:NK62a Cells、Panc 4.03 Cells、L-540 Cells
L132 Cells;背景说明:胚肺;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:SW 579 Cells、UCLA SO M14 Cells、SK-LMS-1 Cells
CTV1 Cells;背景说明:急性T淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:Intestinal Epithelioid Cell line No. 6 Cells、NEC Cells、PANC.1 Cells
Hs274T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代,每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:NCI-H1437 Cells、CAOV3 Cells、PC-9/S1 Cells
CT26WT Cells;背景说明:CT26细胞是被N-亚硝基-N-甲基脲烷(NNMU)诱导得到的未分化的小鼠结肠癌细胞,该细胞的一个克隆形成的细胞系被命名为CT26.WT。CT26.WT被逆转录病毒载体LXSN稳定转化形成了一个致死性的亚克隆CT26.CL25,这一病毒载体含有lacZ基因、编码肿瘤相关抗原(TAA)和beta半乳糖苷酶。CT26.WT和CT26.CL25细胞在小鼠中生长速度和致死率都很相似,不同的是CT26.CL25细胞可以表达肿瘤相关抗原和beta半乳糖苷酶,因此这两株细胞可以联合用于免疫治疗和宿主免疫反应的研究。 ;传代方法:1:4-1:10传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:RAW-264.7 Cells、hRMECs Cells、P3JHR-1 Cells
Ontario Cancer Institute-Acute Myeloid Leukemia-4 Cells;背景说明:急性髓系白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:Hs 294T Cells、U-118MG Cells、HNE-1 Cells
TI-73 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:Hep 3B2.1-7 Cells、KS-SLK Cells、P3J.HR1K Cells
Wien133 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:CHL-CL-11 Cells、BALL1 Cells、LTEP-a2 Cells
Huh-7.5.1 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:L-cells Cells、NPA87 Cells、SMA-560 Cells
MHHCALL2 Cells;背景说明:急性B淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:SK-MEL-24 Cells、Mouse Forestomach Carcinoma Cells、WM35 Cells
47N9-11 Cells(拥有STR基因鉴定图谱)
NCIH196 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明;相关产品有:YES 2 Cells、AZ521 Cells、ZR75-1 Cells
SL-29 Cells;背景说明:胚成纤维细胞; White Leghorn SPAFAS;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:MB39 Cells、LED-WiDr Cells、E0771 Cells
HEK-293FT Cells;背景说明:该细胞稳定表达SV40大T抗原,并且促进最适病毒产物的产生。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:圆形;相关产品有:B16F10 Cells、Jiyoye(P-2003) Cells、High 5 Cells
Colo320 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:淋巴母细胞;相关产品有:PLC/PRF/5 Cells、RBL-I Cells、TERT-RPE1 Cells
H-295R Cells;背景说明:肾上腺皮质癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:SNU-182 Cells、HCMEC Cells、UCH1 Cells
Panc 08.13 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:AZ 521 Cells、HKF Cells、HSAS1 Cells
207 Cells;背景说明:儿童急性髓系白血病;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:CHOS Cells、Panc-03.27 Cells、HEC1-A Cells
SK-N-MC Cells;背景说明:这株细胞与HTB-11都是神经源的。1971年9月分离得到SK-N-MC后,发现它有中性多巴胺-β-羟化酶活性,也有细胞内儿茶胺,用甲醛可以诱导出荧光。;传代方法:1:6-1:12传代,每周2-3次换液。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Panc-02 Cells、TE354T Cells、MDA-MB231 Cells
GM11041 Cells(拥有STR基因鉴定图谱)
HAP1 CLK2 (-) 1 Cells(拥有STR基因鉴定图谱)
COLO_320DM Cells;背景说明:该细胞可产生5-羟色胺、去甲、、ACTH和甲状旁腺素。角蛋白、波形蛋白弱阳性。培养条件: RPMI 1640 10%FBS;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮+贴壁;形态特性:淋巴细胞;相关产品有:HANK-1 Cells、SUDHL-8 Cells、H22 Cells
HG04193 Cells(拥有STR基因鉴定图谱)
iMOSEC-6/BCL-2 Cells(拥有STR基因鉴定图谱)
LUTC-12 Cells(拥有STR基因鉴定图谱)
ND03973 Cells(拥有STR基因鉴定图谱)
PDL-L2 Cells(拥有STR基因鉴定图谱)
UCLAi089-A Cells(拥有STR基因鉴定图谱)
ZP115 Cells(拥有STR基因鉴定图谱)
HCC954 Cells(拥有STR基因鉴定图谱)
MAC1 Cells;背景说明:皮肤T淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:MuM-2B Cells、HCC-1428 Cells、SACC-LM Cells
KNS-81 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H-1876 Cells、RS1 Cells、BRL 3A Cells
HEL 92.1.7 Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:成淋巴细胞;相关产品有:MA104 Cells、MSTO-211 H Cells、T-24 Cells
NCIH1435 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,每周换液2-3次;生长特性:贴壁生长,松散;形态特性:详见产品说明;相关产品有:CHG-5 Cells、293-GP Cells、JIII Cells
NTERA2 Cells;背景说明:畸胎瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:NBL-4 Cells、NK-92 MI Cells、SW 1353 Cells
MESSA-DX5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:8传代;每周2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞样 ;相关产品有:GLC-15 Cells、PLA801D Cells、OCI-AML2 Cells
KU1919 Cells;背景说明:膀胱癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:MC9 Cells、NMC-G1 Cells、Panc 04.03 Cells
OCI/AML-5 Cells;背景说明:急性髓系白血病细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:EC-GI-10 Cells、253J-Bladder-V Cells、MHCC97-H Cells
NHLF Cells;背景说明:肺;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:149PT Cells、A-1847 Cells、Anglne Cells
TOV21G Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Ontario Cancer Institute-Acute Myeloid Leukemia-4 Cells、PANC-1 Cells、SNU398 Cells
Metastatic Variant 522 Cells;背景说明:肺腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:ZR-75-30 Cells、MV3 Cells、MKN-74 Cells
Mevo Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代,2-3天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:Sci-1 Cells、BxPC-3 Cells、MDA2B Cells
OUMS-23 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:210RCY3-Ag1.2.3 Cells、SF 763 Cells、COLO206F Cells
NCI-H1573 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,每周2-3次。;生长特性:贴壁生长;形态特性:详见产品说明;相关产品有:CCD19-Lu Cells、CNE1-LUC Cells、Scott Cells
RAW2647 Cells;背景说明:单核巨噬细胞白血病;雄性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:P30/OHK Cells、0V-1063 Cells、SNU520 Cells
SCVIi077-A Cells(拥有STR基因鉴定图谱)
MDA-MB-435-S Cells;背景说明:MDA-MB-435S是一种纺锤形的细胞,1976年由其亲本(435)中筛选得到。435是从31岁的转移性乳腺导管腺癌女性患者胸水中分离得到。当用荧光染料对微管蛋白进行染色时亲本细胞显现散布特征(II型)。最近通过cDNA阵列研究表明,亲本(MDA-MB-435)可归入黑素瘤起源。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:纺锤形;相关产品有:Kit225-K6 Cells、NTERA2D1 Cells、Hs 737.T Cells
NBLS Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:CEF Cells、RT-BM 1 Cells、RK 13 Cells
SKO007 Cells;背景说明:骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明;相关产品有:L1210 Cells、BL-6 Cells、HNE-3 Cells
Panc-1-P Cells;背景说明:这株人胰腺癌细胞株源自于胰腺癌导管细胞,其倍增时间为52小时。染色体研究表明,该细胞染色体众数为63,包括3个独特标记的染色体和1个小环状染色体。该细胞的生长可被1unit/ml的左旋天冬酰胺酶抑制;能在软琼脂上生长;能在裸鼠上成瘤。;传代方法:1:2-1:4传代;每周2-3次。;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:NCI-H460 Cells、GM03570E Cells、PLA801-95D Cells
BT-20 Cells;背景说明:该细胞1958年由E.Y. Lasfargues 和 L. Ozzello 建系,源自一位74岁白人女性的乳腺癌组织。该细胞表达WNT3和WNT78。TNF alpha抑制该细胞生长。该细胞雌激素受体阴性,但表达5'外显子缺失的雌激素mRNA。;传代方法:1:2—1:4传代,2—3天换液一次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Ramos (RA 1) Cells、H-1694 Cells、MC-4 Cells
RT4P Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,每周2-3次。;生长特性:贴壁生长;形态特性:多角 ;相关产品有:Hep 3B2 Cells、DMS153 Cells、FRhK-4 Cells
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
C17 Cells;背景说明:神经干细胞;C57BL/6 x CD-1;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:MDA-453 Cells、SK-MEL-31 Cells、Tn5B1-4 Cells
MCF/Adr Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:BE(2)M-17 Cells、BEL-7404 Cells、CCC-ESF-1 Cells
COLO-320-HSR Cells;背景说明:该细胞1984年建系,源自一位33岁患有大肠腺癌男性经5-fu治疗后的腹水。;传代方法:1:2传代。3天内可长满。;生长特性:半贴壁生长;形态特性:详见产品说明;相关产品有:OVCA 433 Cells、TERT-RPE1 Cells、RPTEC/TERT1 Cells
Hs852 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:FDCP1 Cells、OV-90 Cells、NALM 6 Cells
WM-115 Cells;背景说明:黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明;相关产品有:NCIH1944 Cells、V-79 Cells、Pt K2 Cells
L-cells Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明部分;形态特性:详见产品说明;相关产品有:UPCI-SCC-090 Cells、IMR32 Cells、NK-10A Cells
Jijoye Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:MDA-157 Cells、NCI-H220 Cells、BT 549 Cells
JVM-2人套细胞淋巴瘤细胞系
MKN 45 Cells;背景说明:该细胞系由S Akiyama建立,源于一位35岁患有印戒细胞癌的女性的胃淋巴结。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁+悬浮;形态特性:淋巴母细胞;相关产品有:BOWES Cells、SF 295 Cells、ESC-410 Cells
BayGenomics ES cell line RRJ207 Cells(拥有STR基因鉴定图谱)
BayGenomics ES cell line XK166 Cells(拥有STR基因鉴定图谱)
FM3A AC1 Cells(拥有STR基因鉴定图谱)
NKI/beteb Cells(拥有STR基因鉴定图谱)
WEGLI-9 Cells(拥有STR基因鉴定图谱)
MCF10A-EGFR1m-3 Cells(拥有STR基因鉴定图谱)
" "DOI=10.1007/0-306-46861-1_11
Ali-Osman F.
Brain tumors.
(In book chapter) Human cell culture. Vol. 2. Cancer cell lines part 2; Masters J.R.W., Palsson B.O. (eds.); pp.167-184; Kluwer Academic Publishers; New York; USA (1999)
PubMed=10416987; DOI=10.1111/j.1750-3639.1999.tb00536.x; PMCID=PMC8098486
Ishii N., Maier D., Merlo A., Tada M., Sawamura Y., Diserens A.-C., Van Meir E.G.
Frequent co-alterations of TP53, p16/CDKN2A, p14ARF, PTEN tumor suppressor genes in human glioma cell lines.
Brain Pathol. 9:469-479(1999)
PubMed=11414198; DOI=10.1007/s004320000207
Lahm H., Andre S., Hoeflich A., Fischer J.R., Sordat B., Kaltner H., Wolf E., Gabius H.-J.
Comprehensive galectin fingerprinting in a panel of 61 human tumor cell lines by RT-PCR and its implications for diagnostic and therapeutic procedures.
J. Cancer Res. Clin. Oncol. 127:375-386(2001)
PubMed=21406405; DOI=10.1158/0008-5472.CAN-10-3112
Grzmil M., Morin P. Jr., Lino M.M., Merlo A., Frank S., Wang Y.-H., Moncayo G., Hemmings B.A.
MAP kinase-interacting kinase 1 regulates SMAD2-dependent TGF-beta signaling pathway in human glioblastoma.
Cancer Res. 71:2392-2402(2011)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22570425; DOI=10.1093/neuonc/nos072; PMCID=PMC3367844
Bady P., Diserens A.-C., Castella V., Kalt S., Heinimann K., Hamou M.-F., Delorenzi M., Hegi M.E.
DNA fingerprinting of glioma cell lines and considerations on similarity measurements.
Neuro-oncol. 14:701-711(2012)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验该产品被引用文献
"DOI=10.1007/0-306-46861-1_11
Ali-Osman F.
Brain tumors.
(In book chapter) Human cell culture. Vol. 2. Cancer cell lines part 2; Masters J.R.W., Palsson B.O. (eds.); pp.167-184; Kluwer Academic Publishers; New York; USA (1999)
PubMed=10416987; DOI=10.1111/j.1750-3639.1999.tb00536.x; PMCID=PMC8098486
Ishii N., Maier D., Merlo A., Tada M., Sawamura Y., Diserens A.-C., Van Meir E.G.
Frequent co-alterations of TP53, p16/CDKN2A, p14ARF, PTEN tumor suppressor genes in human glioma cell lines.
Brain Pathol. 9:469-479(1999)
PubMed=11414198; DOI=10.1007/s004320000207
Lahm H., Andre S., Hoeflich A., Fischer J.R., Sordat B., Kaltner H., Wolf E., Gabius H.-J.
Comprehensive galectin fingerprinting in a panel of 61 human tumor cell lines by RT-PCR and its implications for diagnostic and therapeutic procedures.
J. Cancer Res. Clin. Oncol. 127:375-386(2001)
PubMed=21406405; DOI=10.1158/0008-5472.CAN-10-3112
Grzmil M., Morin P. Jr., Lino M.M., Merlo A., Frank S., Wang Y.-H., Moncayo G., Hemmings B.A.
MAP kinase-interacting kinase 1 regulates SMAD2-dependent TGF-beta signaling pathway in human glioblastoma.
Cancer Res. 71:2392-2402(2011)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22570425; DOI=10.1093/neuonc/nos072; PMCID=PMC3367844
Bady P., Diserens A.-C., Castella V., Kalt S., Heinimann K., Hamou M.-F., Delorenzi M., Hegi M.E.
DNA fingerprinting of glioma cell lines and considerations on similarity measurements.
Neuro-oncol. 14:701-711(2012)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"
Ali-Osman F.
Brain tumors.
(In book chapter) Human cell culture. Vol. 2. Cancer cell lines part 2; Masters J.R.W., Palsson B.O. (eds.); pp.167-184; Kluwer Academic Publishers; New York; USA (1999)
PubMed=10416987; DOI=10.1111/j.1750-3639.1999.tb00536.x; PMCID=PMC8098486
Ishii N., Maier D., Merlo A., Tada M., Sawamura Y., Diserens A.-C., Van Meir E.G.
Frequent co-alterations of TP53, p16/CDKN2A, p14ARF, PTEN tumor suppressor genes in human glioma cell lines.
Brain Pathol. 9:469-479(1999)
PubMed=11414198; DOI=10.1007/s004320000207
Lahm H., Andre S., Hoeflich A., Fischer J.R., Sordat B., Kaltner H., Wolf E., Gabius H.-J.
Comprehensive galectin fingerprinting in a panel of 61 human tumor cell lines by RT-PCR and its implications for diagnostic and therapeutic procedures.
J. Cancer Res. Clin. Oncol. 127:375-386(2001)
PubMed=21406405; DOI=10.1158/0008-5472.CAN-10-3112
Grzmil M., Morin P. Jr., Lino M.M., Merlo A., Frank S., Wang Y.-H., Moncayo G., Hemmings B.A.
MAP kinase-interacting kinase 1 regulates SMAD2-dependent TGF-beta signaling pathway in human glioblastoma.
Cancer Res. 71:2392-2402(2011)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22570425; DOI=10.1093/neuonc/nos072; PMCID=PMC3367844
Bady P., Diserens A.-C., Castella V., Kalt S., Heinimann K., Hamou M.-F., Delorenzi M., Hegi M.E.
DNA fingerprinting of glioma cell lines and considerations on similarity measurements.
Neuro-oncol. 14:701-711(2012)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"
文献支持
JVM-2人套细胞淋巴瘤细胞系
¥850 - 5500