相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 品系:
详见细胞说明资料
- 细胞类型:
详见细胞说明资料
- 肿瘤类型:
详见细胞说明资料
- 供应商:
上海冠导生物工程有限公司
- 库存:
≥100瓶
- 生长状态:
详见细胞说明资料
- 年限:
详见细胞说明资料
- 运输方式:
常温运输【复苏细胞】或干冰运输【冻存细胞】
- 器官来源:
详见细胞说明资料
- 是否是肿瘤细胞:
详见细胞说明资料
- 细胞形态:
详见细胞说明资料
- 免疫类型:
详见细胞说明资料
- 物种来源:
详见细胞说明资料
- 相关疾病:
详见细胞说明资料
- 组织来源:
详见细胞说明资料
- 英文名:
NCI-H1975人肺腺癌细胞种子库|送STR图谱
- 规格:
1*10(6)Cellls/瓶
"NCI-H1975人肺腺癌细胞种子库|送STR图谱
传代方法:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
换液频率:每周2-3次
UT-SCC-30 Cells(拥有STR基因鉴定图谱)
HG02838 Cells(拥有STR基因鉴定图谱)
SW 626 Cells;背景说明:卵巢癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LC-1/sq Cells、HK-2 [Human kidney] Cells、159 PT Cells
NCI-H1975人肺腺癌细胞种子库|送STR图谱
背景资料:这株细胞于1988年七月建株。组织提供者是一位非吸烟人士。
DSMZ菌株保藏中心成立于1969年,是德国的国家菌种保藏中心。该中心一直致力于细菌、真菌、质粒、抗菌素、人体和动物细胞、植物病毒等的分类、鉴定和保藏工作。DSMZ菌种保藏中心是欧洲规模最大的生物资源中心,保藏有动物细胞500多株。Riken BRC成立于1920年,是英国的国家菌种保藏中心。该中心一直致力于细菌、真菌、植物病毒等的分类、鉴定和保藏工作。日本Riken BRC(Riken生物资源保藏中心)是全球三大典型培养物收集中心之一。Riken保藏中心提供了很多细胞系。在世界范围内,这些细胞系,都在医学、科学和兽医中具有重要意义。Riken生物资源中心支持了各种学术、健康、食品和兽医机构的研究工作,并在世界各地不同组织的微生物实验室和研究机构中使用。
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
产品包装形式:复苏细胞:T25培养瓶(一瓶)或冻存细胞:1ml冻存管(两支)
NCI-H1975人肺腺癌细胞种子库|送STR图谱
在细胞传代过程中,离心操作后的上清液处理是一个关键步骤,而关于是用移液枪吸走还是直接倒掉上清液,需要综合多方面因素来考量。使用移液枪吸走上清液具有一定的优势。移液枪能够较为精准地控制吸取的量和速度,可以地减少对细胞沉淀的扰动。可以根据实际情况尽可能地吸净上清液,减少残留血清或培养基成分对后续细胞培养的潜在影响,比如残留的血清可能会改变新培养基的营养成分比例或带来一些未知的生长因子干扰。然而,直接倒掉上清液也并非不可行。在处理一些细胞数量较多、细胞耐受性较好且对实验精度要求并非极高的细胞传代时,直接倒掉上清液能够提高操作效率,节省时间。但这种方式存在一定风险,直接倾倒时较难控制力度和角度,如果操作不慎,容易使细胞沉淀随着上清液一起流出,导致细胞损失,而且可能会因液体快速流下冲击细胞沉淀造成细胞的物理性损伤。在实际的细胞传代操作中,应根据细胞的特性、实验的要求以及个人的操作熟练程度来选择合适的上清液处理方式。对于新手或者处理珍贵细胞系时,建议优先采用移液枪吸走的方式,以保障细胞的活性和传代的成功率。而在积累了丰富经验且对实验条件有充分把握的情况下,可以根据具体情况灵活运用直接倒掉上清液的方法,在保证实验质量的同时提高工作效率。
来源说明:细胞主要来源ATCC、DSMZ等细胞库
物种来源:Human\Mouse\Rat\Others
MN60 Cells;背景说明:B细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:COLO-679 Cells、VMRCRCZ Cells、WISH Cells
RMS 13 Cells;背景说明:肺泡横纹肌肉瘤;骨髓转移;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1793 Cells、EBC1 Cells、LC-1Sq Cells
BNL.1ME A.7R.1 Cells;背景说明:肝;上皮细胞;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H740 Cells、NCI-SNU-C1 Cells、3T3-Swiss albino Cells
Hep 3B2_1-7 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SMMC 7721 Cells、QGP 1 Cells、HEH2 Cells
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
NCI-H1975人肺腺癌细胞种子库|送STR图谱
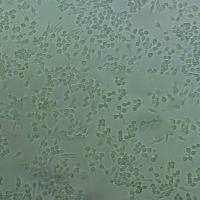
形态特性:上皮细胞样
培养细胞真的不难,Zui关键几点:1)所有的东西使用,如果你用的血清、培养皿、培养基、胰酶什么的都是进口的,真的很难相信你会把细胞养坏;2)动作要快,胰酶消化,换什么,细胞暴露在空气中的时间越少越HAO。另外,要掌握HAO胰酶消化时间,所谓的细胞状态差,很多时候是传代的原因;3)有时间的话,需要细胞计数,传相应数目的细胞到培养皿中,可以大致清楚细胞的生长曲线,不会临时要处理,手足无措;4)要做什么心里要非常清楚,合理安排时间,传代细胞的实验穿插进行,可以节省很多时间,也不会耽误别人的实验;5)个人的实验器具自己收一套,不要和别人混用,Zui近换了什么新的东西稍微记一下,试剂一旦怀疑污染,马上丢;6)传代细胞不能进去太多人,避免相互干扰。每天对待细胞比孝敬爹妈还用心,那真是捧在手里怕碎了,含在嘴里怕化了,看在眼里怕丢了,培养细胞时有哪些经验教训可以分享呢?1)严格无菌操作,多喷喷酒精,要是对灭菌的东西不放心,自己包自己送自己烘干;2)不要和别人共用试剂耗材,自己准备一套;3)有可能的话在拿到新细胞的时候做HAO支原体衣原体检测;4)水浴锅很脏,生化培养箱要是得手动加湿的话盘里的水也会很脏,勤换(灭菌水加进去);5)晚上离开的时候ZuiHAO给传代细胞照紫外,超净台是用完就开;6)是同一时间就你一个人在用传代细胞在养细胞。
UWB1289 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:A 2780 CP Cells、Panc_05_04 Cells、NCIH2081 Cells
Panc-327 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HCEC-12 Cells、SN12C Cells、SNU-C2B Cells
SKHEP1 Cells;背景说明:SK-HEP-1细胞系已被鉴定为内皮来源。该细胞系为异倍体女性人(XX),染色体在亚三倍体范围内。在裸鼠中,它能形成与肝癌相一致的大细胞癌;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Rat 1 Cells、Human Liver-7702 Cells、KHYG1 Cells
HEL-92.1.7 Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:成淋巴细胞;相关产品有:H-35 Reuber Cells、SW620 Cells、H-1836 Cells
SW-1417 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液1-2次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Jurkat FHCRC Cells、NCI-H889 Cells、JURKAT E-6.1 Cells
B16 melanoma Cells;背景说明:该细胞源于C57BL/6J小鼠黑色素瘤,可以产生黑色素,同基因小鼠体内移植可成瘤;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:梭形;相关产品有:As-PC1 Cells、End1/E6E7 Cells、H889 Cells
SO-RB50 Cells;背景说明:视网膜母细胞瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:U118-MG Cells、NP69SV40T Cells、H-660 Cells
Granta519 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TE15 Cells、Balb/c 3T3 Cells、Ishikawa Cells
MC 116 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:GM06141 Cells、KFB Cells、MH-S Cells
EST50 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代,2-3天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:CCD-1095Sk Cells、GLRK 13 Cells、SKES1 Cells
EB-3 Cells;背景说明:该细胞源自一名3岁患有Burkitt's淋巴瘤的黑人男孩的B淋巴细胞,EBNA阳性。;传代方法:1:2-1:4传代,每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:SW 1783 Cells、SK-NM-C Cells、Y3-Ag 1.2.3 Cells
ESC-410 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HOS-143B Cells、FOXNY Cells、HANK-1 Cells
H3255_DA Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HCS2/8 Cells、KPL-4 Cells、OCI Ly3 Cells
GM2131 Cells;背景说明:B淋巴细胞;EBV转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:RT-112 Cells、P3-X63-Ag8-6-5-3 Cells、PIEC Cells
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
Ca Ski Cells;背景说明:这株细胞是从小肠肠系膜转移灶的细胞中建立的。 据报道,它含有完整的HPV-16(每个细胞大约600个拷贝)和HPV-18相关序列。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Tb 1 Lu (NBL-12) Cells、University of Michigan-Urothelial Carcinoma-14 Cells、D-407 Cells
MLE-12 Cells;背景说明:肺;上皮细胞;SV40转化;FVB/N;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MDA361 Cells、Sp 2817 Cells、TMK1 Cells
L-cell Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MC116 Cells、TW-039 Cells、NCI-H1755 Cells
Abcam HEK293T FAM174C KO Cells(拥有STR基因鉴定图谱)
AG10097 Cells(拥有STR基因鉴定图谱)
BayGenomics ES cell line NPX481 Cells(拥有STR基因鉴定图谱)
BayGenomics ES cell line XC671 Cells(拥有STR基因鉴定图谱)
BT073 Cells(拥有STR基因鉴定图谱)
CP5 Cells(拥有STR基因鉴定图谱)
DA04083 Cells(拥有STR基因鉴定图谱)
FM3A AC1 Cells(拥有STR基因鉴定图谱)
GM09488 Cells(拥有STR基因鉴定图谱)
NCI-H1975人肺腺癌细胞种子库|送STR图谱
NCIH82 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代,每周换液2-3次;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:HT55 Cells、NTera 2 Cells、Stanford University-Diffuse Histiocytic Lymphoma-1 Cells
A427 Cells;背景说明:肺腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CCD 841 CoN Cells、SW 954 Cells、RH-35 Cells
TE-13 Cells;背景说明:食管鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Jurkat clone A3 Cells、HOS (TE85) Cells、NRK Cells
NCI-H1668 Cells;背景说明:小细胞肺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NPA Cells、Michigan Cancer Foundation-7 Cells、HOS TE85 Cells
OCILY10 Cells;背景说明:弥漫大B细胞淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HLF-a Cells、RSMC Cells、C3H10T1/2 clone8 Cells
KHYG-1 Cells;背景说明:NK细胞淋巴瘤/白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Y3 Cells、GM00346 Cells、BALB 3T3 clone A31 Cells
GLC82 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCTC 929 Cells、Panc5.04 Cells、TE-8 Cells
H-87 Cells;背景说明:NCI-N87细胞表达表面糖蛋白癌胚抗原(CEA)和TAG72,并且没有左旋多巴胺脱羧酶(DDC)活性。它们的血管活性的肠肽(VIP)受体活性极低并缺乏胃泌激素受体。它们表达蕈毒碱胆碱受体。没有证据表明存在N-myc,L-myc,myb和EGF受体基因的重组。这个细胞株表达的c-myc和c-erb-B2RNA水平与其它细胞株相当。以下基因不表达:N-myc,L-myc,c-cis,IGF-2,或胃泌激素释放肽。报道NCI-N87细胞的植板率为4.3%。;传代方法:消化15-20分钟。1:2。4-5天长满。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Panc813 Cells、Karpas299 Cells、ZR75-30 Cells
NT-2 Cells;背景说明:畸胎瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CATH.a Cells、CT 26 Cells、3T3-L1 Cells
UCD-MLA-144 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SW-900 Cells、L132 Cells、SK MEL 5 Cells
H-441 Cells;背景说明:NCI-H441建系于1982年(A.F.Gazdar,etal.)。该细胞分离自一名肺腺癌患者的心包液。该细胞能在半固体琼脂糖中形成克隆,并能表达肺泡表面活性蛋白A。该细胞在有血清培养基中倍增时间为58小时,在无血清培养基中倍增时间为99-138小时。;传代方法:1:3传代,2-3天传一代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:769-P Cells、OVCAR 5 Cells、SUM-190PT Cells
COR-L105 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HSC1 Cells、NCIH1435 Cells、Tca-8113 Cells
WiDr/S Cells;背景说明:结肠癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:WERI-Rb1 Cells、MDCK (NBL-2) Cells、Hs-343-T Cells
BayGenomics ES cell line CSA008 Cells(拥有STR基因鉴定图谱)
Tu-177 Cells;背景说明:喉鳞癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MUG-Chor1 Cells、NIH:OVCAR-10 Cells、WM2664 Cells
FUOV1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COLO320 DM Cells、MonoMac 6 Cells、MNNGHOS Cells
MD Anderson-Metastatic Breast-435 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LNCaP-FGC Cells、HOC-1 Cells、CMT-64 Cells
Mono-Mac-1 Cells;背景说明:急性单核细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:ssMCF7 Cells、ATC241 Cells、Normal Rat Kidney Cells
RBL-I Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PFSK1 Cells、B16 Cells、Institute for Medical Research-32 Cells
NCI H69 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2次;生长特性:悬浮生长,聚团;形态特性:聚团悬浮;相关产品有:SU.86.86 Cells、CAL51 Cells、SNU-387 Cells
T8 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Lu-65 Cells、SK-SH-SY5Y Cells、RCC23 Cells
WPMY-1 Cells;背景说明:肌成纤维基质细胞株,WPMY-1,与RWPE-1cells(ATCCCRL-11609)一样,来源于同一张成人前列腺组织切片的周围区域的基质细胞。通过一个pRSTV质料结构,用SV40大T抗原对基质细胞进行永生化。WPMY-1细胞,与RWPE-1细胞及其它上皮细胞衍生株一样,属于来源于同一个前列腺的一系列细胞株。由于它们来源于同一个前列腺的周围区域,WPMY-1细胞株对于研究分泌和基质与上皮细胞相互作用尤其有用。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:成肌细胞;相关产品有:8226 Cells、K7M2-WT Cells、VM Cub 1 Cells
GM28476 Cells(拥有STR基因鉴定图谱)
HAP1 PDK1 (-) 1 Cells(拥有STR基因鉴定图谱)
MOLM13 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CAL 85-1 Cells、HCC-1954BL Cells、Hep-3B Cells
OCIAML3 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:BetaTC6 Cells、GM2132 Cells、HS-5 Cells
32D clone3 Cells;背景说明:骨髓淋巴瘤;C3H/HeJ;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:A9 Cells、4T1-A Cells、PANC-02-03 Cells
Hs 611.T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次。;生长特性:混合型;形态特性:淋巴母细胞样;相关产品有:PE/CA-PJ34 (clone C12) Cells、P3/X63-Ag8 Cells、HPAF/CD18 Cells
Dysplastic Oral Keratinocyte Cells;背景说明:口腔异常增生;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ACC2 Cells、SK-RC-42 Cells、SKMEL1 Cells
NPC-TW039 Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Madison lung Cells、HCC-1954 Cells、Ramos (RA) Cells
GC-2 Cells;背景说明:精母细胞;SV40转化;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OVCAR 4 Cells、C57/B6-L Cells、TSCC1 Cells
DHL-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KS-1 [Human glioblastoma] Cells、Human Microvascular Endothelial Cell line-1 Cells、HCC0070 Cells
HS841 Cells(拥有STR基因鉴定图谱)
KOLF2.1J PRKN R42P SNV/WT Cells(拥有STR基因鉴定图谱)
MOCL8 Cells(拥有STR基因鉴定图谱)
NWR 5765 iPSC-c507 Cells(拥有STR基因鉴定图谱)
RG-286 Cells(拥有STR基因鉴定图谱)
Ubigene A-549 NFKB1 KO Cells(拥有STR基因鉴定图谱)
Hep 3B2_1-7 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SMMC 7721 Cells、QGP 1 Cells、HEH2 Cells
FHCRC subclone 11 Cells;背景说明:该细胞源自一位14岁患有T淋巴细胞白血病男性的外周血;传代方法:保持细胞密度在3—9×105cells/ml之间,1:5—1:10传代,每周换液2—3次;生长特性:悬浮生长;形态特性:圆形,单个或呈片;相关产品有:3T3 Cells、OCM-1A Cells、ARO81 Cells
FCCH1024 Cells;背景说明:该细胞源自一位14岁患有T淋巴细胞白血病男性的外周血;传代方法:保持细胞密度在3—9×105cells/ml之间,1:5—1:10传代,每周换液2—3次;生长特性:悬浮生长;形态特性:圆形,单个或呈片;相关产品有:751-NA Cells、KBM-7 Cells、Emory University-3 Cells
P3-X63Ag8.653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PL-11 Cells、RMS1598 Cells、MUTZ-1 Cells
Hep-G2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Emory University-2 Cells、FK81 Cells、CCC-HSF-1 Cells
HeLa-229 Cells;背景说明:宫颈癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEK 293T/17 Cells、HSC-5 [Human skin squamous cell carcinoma] Cells、SP2/0 Ag-14 Cells
Hep 3B2_1-7 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SMMC 7721 Cells、QGP 1 Cells、HEH2 Cells
MCF-7/ADR-RES Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PT67 Cells、B16-F0 Cells、MDAMB-231 Cells
NCI-H87 Cells;背景说明:NCI-N87细胞表达表面糖蛋白癌胚抗原(CEA)和TAG72,并且没有左旋多巴胺脱羧酶(DDC)活性。它们的血管活性的肠肽(VIP)受体活性极低并缺乏胃泌激素受体。它们表达蕈毒碱胆碱受体。没有证据表明存在N-myc,L-myc,myb和EGF受体基因的重组。这个细胞株表达的c-myc和c-erb-B2RNA水平与其它细胞株相当。以下基因不表达:N-myc,L-myc,c-cis,IGF-2,或胃泌激素释放肽。报道NCI-N87细胞的植板率为4.3%。;传代方法:消化15-20分钟。1:2。4-5天长满。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:7404 Cells、RH-35 Cells、COLO824 Cells
HCC1954 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2-3次。;生长特性:偶尔上皮细胞空泡;形态特性:上皮细胞样;相关产品有:MFE280 Cells、A549-Taxol Cells、SKO3 Cells
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
STPLF-fF Cells(拥有STR基因鉴定图谱)
LC-2 ad Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C17.2 Cells、IMR-32 Cells、SMA 560 Cells
NCIH1581 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:混合型;形态特性:上皮样;相关产品有:H-125 Cells、EB-2 Cells、Soleus clone 8 Cells
GM00215A Cells;背景说明:肺;自发永生;雄性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HOSEpiC Cells、H2122 Cells、GM02219D Cells
NCI-H2085 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代 ;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:L-6 Cells、H-7721 Cells、HT55 Cells
OCI/AML3 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:hRMECs Cells、Fetal Bovine Heart Endothelial Cells、PC.3 Cells
OP9 Cells;背景说明:骨髓基质;C57BL/6 x C3H;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU354 Cells、P3X63 Ag8 Cells、HCC-1187 Cells
D6P2T Cells;背景说明:详见相关文献介绍;传代方法:1:5—1:10传代;生长特性:贴壁生长;形态特性:神经元 ;相关产品有:HFL1 Cells、SAS Cells、Hs 675.T Cells
COLO 738 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:T 98 G Cells、Hs 688(A).T Cells、P560 Cells
HOP-92 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:L-WRN Cells、SCC25 Cells、LA-N-6 Cells
Henrietta Lacks cells Cells;背景说明:HeLa是第一个来自人体组织经连续培养获得的非整倍体上皮样细胞系,它由GeyGO等在1951年从31岁女性黑人的宫颈癌组织建立。经原始组织切片重新观察,Jones等将其诊断为腺癌。已知该细胞系含有人乳头状瘤病毒HPV18序列,需在2级生物安全防护台操作。该细胞角蛋白阳性,p53表达量较低,但表达正常水平的pRB(视网膜母细胞瘤抑制因子)。;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:IM 9 Cells、U 937 Cells、ECV Cells
Mouse Bladder Tumor line-2 Cells;背景说明:膀胱移行细胞癌;C3H/He;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NPA87-1 Cells、Douglas Foster-1 Cells、Ca 9-22 Cells
IHC-ST1 Cells;背景说明:肝内胆管癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Be-Wo Cells、CF-1 MEF Cells、M-1 myeloid leukemia Cells
SK-MEL-24 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:Capan 2 Cells、639 V Cells、NCI-HUT-292 Cells
MLA144 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:A875 Cells、HT-144 Cells、PANC0203 Cells
BayGenomics ES cell line RRJ104 Cells(拥有STR基因鉴定图谱)
NCI-H1975人肺腺癌细胞种子库|送STR图谱
BayGenomics ES cell line XK089 Cells(拥有STR基因鉴定图谱)
FE1 Cells(拥有STR基因鉴定图谱)
NIH 3T3/DC-SIGN Cells(拥有STR基因鉴定图谱)
W2 [Mouse ESC] Cells(拥有STR基因鉴定图谱)
MCF-7R Cells(拥有STR基因鉴定图谱)
"
传代方法:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
换液频率:每周2-3次
UT-SCC-30 Cells(拥有STR基因鉴定图谱)
HG02838 Cells(拥有STR基因鉴定图谱)
SW 626 Cells;背景说明:卵巢癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LC-1/sq Cells、HK-2 [Human kidney] Cells、159 PT Cells
NCI-H1975人肺腺癌细胞种子库|送STR图谱
背景资料:这株细胞于1988年七月建株。组织提供者是一位非吸烟人士。
DSMZ菌株保藏中心成立于1969年,是德国的国家菌种保藏中心。该中心一直致力于细菌、真菌、质粒、抗菌素、人体和动物细胞、植物病毒等的分类、鉴定和保藏工作。DSMZ菌种保藏中心是欧洲规模最大的生物资源中心,保藏有动物细胞500多株。Riken BRC成立于1920年,是英国的国家菌种保藏中心。该中心一直致力于细菌、真菌、植物病毒等的分类、鉴定和保藏工作。日本Riken BRC(Riken生物资源保藏中心)是全球三大典型培养物收集中心之一。Riken保藏中心提供了很多细胞系。在世界范围内,这些细胞系,都在医学、科学和兽医中具有重要意义。Riken生物资源中心支持了各种学术、健康、食品和兽医机构的研究工作,并在世界各地不同组织的微生物实验室和研究机构中使用。
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
产品包装形式:复苏细胞:T25培养瓶(一瓶)或冻存细胞:1ml冻存管(两支)
NCI-H1975人肺腺癌细胞种子库|送STR图谱
在细胞传代过程中,离心操作后的上清液处理是一个关键步骤,而关于是用移液枪吸走还是直接倒掉上清液,需要综合多方面因素来考量。使用移液枪吸走上清液具有一定的优势。移液枪能够较为精准地控制吸取的量和速度,可以地减少对细胞沉淀的扰动。可以根据实际情况尽可能地吸净上清液,减少残留血清或培养基成分对后续细胞培养的潜在影响,比如残留的血清可能会改变新培养基的营养成分比例或带来一些未知的生长因子干扰。然而,直接倒掉上清液也并非不可行。在处理一些细胞数量较多、细胞耐受性较好且对实验精度要求并非极高的细胞传代时,直接倒掉上清液能够提高操作效率,节省时间。但这种方式存在一定风险,直接倾倒时较难控制力度和角度,如果操作不慎,容易使细胞沉淀随着上清液一起流出,导致细胞损失,而且可能会因液体快速流下冲击细胞沉淀造成细胞的物理性损伤。在实际的细胞传代操作中,应根据细胞的特性、实验的要求以及个人的操作熟练程度来选择合适的上清液处理方式。对于新手或者处理珍贵细胞系时,建议优先采用移液枪吸走的方式,以保障细胞的活性和传代的成功率。而在积累了丰富经验且对实验条件有充分把握的情况下,可以根据具体情况灵活运用直接倒掉上清液的方法,在保证实验质量的同时提高工作效率。
来源说明:细胞主要来源ATCC、DSMZ等细胞库
物种来源:Human\Mouse\Rat\Others
MN60 Cells;背景说明:B细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:COLO-679 Cells、VMRCRCZ Cells、WISH Cells
RMS 13 Cells;背景说明:肺泡横纹肌肉瘤;骨髓转移;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1793 Cells、EBC1 Cells、LC-1Sq Cells
BNL.1ME A.7R.1 Cells;背景说明:肝;上皮细胞;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H740 Cells、NCI-SNU-C1 Cells、3T3-Swiss albino Cells
Hep 3B2_1-7 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SMMC 7721 Cells、QGP 1 Cells、HEH2 Cells
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
NCI-H1975人肺腺癌细胞种子库|送STR图谱
形态特性:上皮细胞样
培养细胞真的不难,Zui关键几点:1)所有的东西使用,如果你用的血清、培养皿、培养基、胰酶什么的都是进口的,真的很难相信你会把细胞养坏;2)动作要快,胰酶消化,换什么,细胞暴露在空气中的时间越少越HAO。另外,要掌握HAO胰酶消化时间,所谓的细胞状态差,很多时候是传代的原因;3)有时间的话,需要细胞计数,传相应数目的细胞到培养皿中,可以大致清楚细胞的生长曲线,不会临时要处理,手足无措;4)要做什么心里要非常清楚,合理安排时间,传代细胞的实验穿插进行,可以节省很多时间,也不会耽误别人的实验;5)个人的实验器具自己收一套,不要和别人混用,Zui近换了什么新的东西稍微记一下,试剂一旦怀疑污染,马上丢;6)传代细胞不能进去太多人,避免相互干扰。每天对待细胞比孝敬爹妈还用心,那真是捧在手里怕碎了,含在嘴里怕化了,看在眼里怕丢了,培养细胞时有哪些经验教训可以分享呢?1)严格无菌操作,多喷喷酒精,要是对灭菌的东西不放心,自己包自己送自己烘干;2)不要和别人共用试剂耗材,自己准备一套;3)有可能的话在拿到新细胞的时候做HAO支原体衣原体检测;4)水浴锅很脏,生化培养箱要是得手动加湿的话盘里的水也会很脏,勤换(灭菌水加进去);5)晚上离开的时候ZuiHAO给传代细胞照紫外,超净台是用完就开;6)是同一时间就你一个人在用传代细胞在养细胞。
UWB1289 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:A 2780 CP Cells、Panc_05_04 Cells、NCIH2081 Cells
Panc-327 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HCEC-12 Cells、SN12C Cells、SNU-C2B Cells
SKHEP1 Cells;背景说明:SK-HEP-1细胞系已被鉴定为内皮来源。该细胞系为异倍体女性人(XX),染色体在亚三倍体范围内。在裸鼠中,它能形成与肝癌相一致的大细胞癌;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Rat 1 Cells、Human Liver-7702 Cells、KHYG1 Cells
HEL-92.1.7 Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:成淋巴细胞;相关产品有:H-35 Reuber Cells、SW620 Cells、H-1836 Cells
SW-1417 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液1-2次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Jurkat FHCRC Cells、NCI-H889 Cells、JURKAT E-6.1 Cells
B16 melanoma Cells;背景说明:该细胞源于C57BL/6J小鼠黑色素瘤,可以产生黑色素,同基因小鼠体内移植可成瘤;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:梭形;相关产品有:As-PC1 Cells、End1/E6E7 Cells、H889 Cells
SO-RB50 Cells;背景说明:视网膜母细胞瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:U118-MG Cells、NP69SV40T Cells、H-660 Cells
Granta519 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TE15 Cells、Balb/c 3T3 Cells、Ishikawa Cells
MC 116 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:GM06141 Cells、KFB Cells、MH-S Cells
EST50 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代,2-3天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:CCD-1095Sk Cells、GLRK 13 Cells、SKES1 Cells
EB-3 Cells;背景说明:该细胞源自一名3岁患有Burkitt's淋巴瘤的黑人男孩的B淋巴细胞,EBNA阳性。;传代方法:1:2-1:4传代,每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:SW 1783 Cells、SK-NM-C Cells、Y3-Ag 1.2.3 Cells
ESC-410 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HOS-143B Cells、FOXNY Cells、HANK-1 Cells
H3255_DA Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HCS2/8 Cells、KPL-4 Cells、OCI Ly3 Cells
GM2131 Cells;背景说明:B淋巴细胞;EBV转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:RT-112 Cells、P3-X63-Ag8-6-5-3 Cells、PIEC Cells
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
Ca Ski Cells;背景说明:这株细胞是从小肠肠系膜转移灶的细胞中建立的。 据报道,它含有完整的HPV-16(每个细胞大约600个拷贝)和HPV-18相关序列。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Tb 1 Lu (NBL-12) Cells、University of Michigan-Urothelial Carcinoma-14 Cells、D-407 Cells
MLE-12 Cells;背景说明:肺;上皮细胞;SV40转化;FVB/N;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MDA361 Cells、Sp 2817 Cells、TMK1 Cells
L-cell Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MC116 Cells、TW-039 Cells、NCI-H1755 Cells
Abcam HEK293T FAM174C KO Cells(拥有STR基因鉴定图谱)
AG10097 Cells(拥有STR基因鉴定图谱)
BayGenomics ES cell line NPX481 Cells(拥有STR基因鉴定图谱)
BayGenomics ES cell line XC671 Cells(拥有STR基因鉴定图谱)
BT073 Cells(拥有STR基因鉴定图谱)
CP5 Cells(拥有STR基因鉴定图谱)
DA04083 Cells(拥有STR基因鉴定图谱)
FM3A AC1 Cells(拥有STR基因鉴定图谱)
GM09488 Cells(拥有STR基因鉴定图谱)
NCI-H1975人肺腺癌细胞种子库|送STR图谱
NCIH82 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代,每周换液2-3次;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:HT55 Cells、NTera 2 Cells、Stanford University-Diffuse Histiocytic Lymphoma-1 Cells
A427 Cells;背景说明:肺腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CCD 841 CoN Cells、SW 954 Cells、RH-35 Cells
TE-13 Cells;背景说明:食管鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Jurkat clone A3 Cells、HOS (TE85) Cells、NRK Cells
NCI-H1668 Cells;背景说明:小细胞肺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NPA Cells、Michigan Cancer Foundation-7 Cells、HOS TE85 Cells
OCILY10 Cells;背景说明:弥漫大B细胞淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HLF-a Cells、RSMC Cells、C3H10T1/2 clone8 Cells
KHYG-1 Cells;背景说明:NK细胞淋巴瘤/白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Y3 Cells、GM00346 Cells、BALB 3T3 clone A31 Cells
GLC82 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCTC 929 Cells、Panc5.04 Cells、TE-8 Cells
H-87 Cells;背景说明:NCI-N87细胞表达表面糖蛋白癌胚抗原(CEA)和TAG72,并且没有左旋多巴胺脱羧酶(DDC)活性。它们的血管活性的肠肽(VIP)受体活性极低并缺乏胃泌激素受体。它们表达蕈毒碱胆碱受体。没有证据表明存在N-myc,L-myc,myb和EGF受体基因的重组。这个细胞株表达的c-myc和c-erb-B2RNA水平与其它细胞株相当。以下基因不表达:N-myc,L-myc,c-cis,IGF-2,或胃泌激素释放肽。报道NCI-N87细胞的植板率为4.3%。;传代方法:消化15-20分钟。1:2。4-5天长满。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Panc813 Cells、Karpas299 Cells、ZR75-30 Cells
NT-2 Cells;背景说明:畸胎瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CATH.a Cells、CT 26 Cells、3T3-L1 Cells
UCD-MLA-144 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SW-900 Cells、L132 Cells、SK MEL 5 Cells
H-441 Cells;背景说明:NCI-H441建系于1982年(A.F.Gazdar,etal.)。该细胞分离自一名肺腺癌患者的心包液。该细胞能在半固体琼脂糖中形成克隆,并能表达肺泡表面活性蛋白A。该细胞在有血清培养基中倍增时间为58小时,在无血清培养基中倍增时间为99-138小时。;传代方法:1:3传代,2-3天传一代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:769-P Cells、OVCAR 5 Cells、SUM-190PT Cells
COR-L105 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HSC1 Cells、NCIH1435 Cells、Tca-8113 Cells
WiDr/S Cells;背景说明:结肠癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:WERI-Rb1 Cells、MDCK (NBL-2) Cells、Hs-343-T Cells
BayGenomics ES cell line CSA008 Cells(拥有STR基因鉴定图谱)
Tu-177 Cells;背景说明:喉鳞癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MUG-Chor1 Cells、NIH:OVCAR-10 Cells、WM2664 Cells
FUOV1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COLO320 DM Cells、MonoMac 6 Cells、MNNGHOS Cells
MD Anderson-Metastatic Breast-435 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LNCaP-FGC Cells、HOC-1 Cells、CMT-64 Cells
Mono-Mac-1 Cells;背景说明:急性单核细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:ssMCF7 Cells、ATC241 Cells、Normal Rat Kidney Cells
RBL-I Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PFSK1 Cells、B16 Cells、Institute for Medical Research-32 Cells
NCI H69 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2次;生长特性:悬浮生长,聚团;形态特性:聚团悬浮;相关产品有:SU.86.86 Cells、CAL51 Cells、SNU-387 Cells
T8 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Lu-65 Cells、SK-SH-SY5Y Cells、RCC23 Cells
WPMY-1 Cells;背景说明:肌成纤维基质细胞株,WPMY-1,与RWPE-1cells(ATCCCRL-11609)一样,来源于同一张成人前列腺组织切片的周围区域的基质细胞。通过一个pRSTV质料结构,用SV40大T抗原对基质细胞进行永生化。WPMY-1细胞,与RWPE-1细胞及其它上皮细胞衍生株一样,属于来源于同一个前列腺的一系列细胞株。由于它们来源于同一个前列腺的周围区域,WPMY-1细胞株对于研究分泌和基质与上皮细胞相互作用尤其有用。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:成肌细胞;相关产品有:8226 Cells、K7M2-WT Cells、VM Cub 1 Cells
GM28476 Cells(拥有STR基因鉴定图谱)
HAP1 PDK1 (-) 1 Cells(拥有STR基因鉴定图谱)
MOLM13 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CAL 85-1 Cells、HCC-1954BL Cells、Hep-3B Cells
OCIAML3 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:BetaTC6 Cells、GM2132 Cells、HS-5 Cells
32D clone3 Cells;背景说明:骨髓淋巴瘤;C3H/HeJ;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:A9 Cells、4T1-A Cells、PANC-02-03 Cells
Hs 611.T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次。;生长特性:混合型;形态特性:淋巴母细胞样;相关产品有:PE/CA-PJ34 (clone C12) Cells、P3/X63-Ag8 Cells、HPAF/CD18 Cells
Dysplastic Oral Keratinocyte Cells;背景说明:口腔异常增生;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ACC2 Cells、SK-RC-42 Cells、SKMEL1 Cells
NPC-TW039 Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Madison lung Cells、HCC-1954 Cells、Ramos (RA) Cells
GC-2 Cells;背景说明:精母细胞;SV40转化;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OVCAR 4 Cells、C57/B6-L Cells、TSCC1 Cells
DHL-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KS-1 [Human glioblastoma] Cells、Human Microvascular Endothelial Cell line-1 Cells、HCC0070 Cells
HS841 Cells(拥有STR基因鉴定图谱)
KOLF2.1J PRKN R42P SNV/WT Cells(拥有STR基因鉴定图谱)
MOCL8 Cells(拥有STR基因鉴定图谱)
NWR 5765 iPSC-c507 Cells(拥有STR基因鉴定图谱)
RG-286 Cells(拥有STR基因鉴定图谱)
Ubigene A-549 NFKB1 KO Cells(拥有STR基因鉴定图谱)
Hep 3B2_1-7 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SMMC 7721 Cells、QGP 1 Cells、HEH2 Cells
FHCRC subclone 11 Cells;背景说明:该细胞源自一位14岁患有T淋巴细胞白血病男性的外周血;传代方法:保持细胞密度在3—9×105cells/ml之间,1:5—1:10传代,每周换液2—3次;生长特性:悬浮生长;形态特性:圆形,单个或呈片;相关产品有:3T3 Cells、OCM-1A Cells、ARO81 Cells
FCCH1024 Cells;背景说明:该细胞源自一位14岁患有T淋巴细胞白血病男性的外周血;传代方法:保持细胞密度在3—9×105cells/ml之间,1:5—1:10传代,每周换液2—3次;生长特性:悬浮生长;形态特性:圆形,单个或呈片;相关产品有:751-NA Cells、KBM-7 Cells、Emory University-3 Cells
P3-X63Ag8.653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PL-11 Cells、RMS1598 Cells、MUTZ-1 Cells
Hep-G2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Emory University-2 Cells、FK81 Cells、CCC-HSF-1 Cells
HeLa-229 Cells;背景说明:宫颈癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEK 293T/17 Cells、HSC-5 [Human skin squamous cell carcinoma] Cells、SP2/0 Ag-14 Cells
Hep 3B2_1-7 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SMMC 7721 Cells、QGP 1 Cells、HEH2 Cells
MCF-7/ADR-RES Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PT67 Cells、B16-F0 Cells、MDAMB-231 Cells
NCI-H87 Cells;背景说明:NCI-N87细胞表达表面糖蛋白癌胚抗原(CEA)和TAG72,并且没有左旋多巴胺脱羧酶(DDC)活性。它们的血管活性的肠肽(VIP)受体活性极低并缺乏胃泌激素受体。它们表达蕈毒碱胆碱受体。没有证据表明存在N-myc,L-myc,myb和EGF受体基因的重组。这个细胞株表达的c-myc和c-erb-B2RNA水平与其它细胞株相当。以下基因不表达:N-myc,L-myc,c-cis,IGF-2,或胃泌激素释放肽。报道NCI-N87细胞的植板率为4.3%。;传代方法:消化15-20分钟。1:2。4-5天长满。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:7404 Cells、RH-35 Cells、COLO824 Cells
HCC1954 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2-3次。;生长特性:偶尔上皮细胞空泡;形态特性:上皮细胞样;相关产品有:MFE280 Cells、A549-Taxol Cells、SKO3 Cells
┈订┈购┈热┈线:1┈5┈8┈0┈0┈5┈7┈6┈8┈6┈7【微信同号】┈Q┈Q:3┈3┈0┈7┈2┈0┈4┈2┈7┈1;
STPLF-fF Cells(拥有STR基因鉴定图谱)
LC-2 ad Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C17.2 Cells、IMR-32 Cells、SMA 560 Cells
NCIH1581 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:混合型;形态特性:上皮样;相关产品有:H-125 Cells、EB-2 Cells、Soleus clone 8 Cells
GM00215A Cells;背景说明:肺;自发永生;雄性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HOSEpiC Cells、H2122 Cells、GM02219D Cells
NCI-H2085 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代 ;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:L-6 Cells、H-7721 Cells、HT55 Cells
OCI/AML3 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:hRMECs Cells、Fetal Bovine Heart Endothelial Cells、PC.3 Cells
OP9 Cells;背景说明:骨髓基质;C57BL/6 x C3H;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU354 Cells、P3X63 Ag8 Cells、HCC-1187 Cells
D6P2T Cells;背景说明:详见相关文献介绍;传代方法:1:5—1:10传代;生长特性:贴壁生长;形态特性:神经元 ;相关产品有:HFL1 Cells、SAS Cells、Hs 675.T Cells
COLO 738 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:T 98 G Cells、Hs 688(A).T Cells、P560 Cells
HOP-92 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:L-WRN Cells、SCC25 Cells、LA-N-6 Cells
Henrietta Lacks cells Cells;背景说明:HeLa是第一个来自人体组织经连续培养获得的非整倍体上皮样细胞系,它由GeyGO等在1951年从31岁女性黑人的宫颈癌组织建立。经原始组织切片重新观察,Jones等将其诊断为腺癌。已知该细胞系含有人乳头状瘤病毒HPV18序列,需在2级生物安全防护台操作。该细胞角蛋白阳性,p53表达量较低,但表达正常水平的pRB(视网膜母细胞瘤抑制因子)。;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:IM 9 Cells、U 937 Cells、ECV Cells
Mouse Bladder Tumor line-2 Cells;背景说明:膀胱移行细胞癌;C3H/He;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NPA87-1 Cells、Douglas Foster-1 Cells、Ca 9-22 Cells
IHC-ST1 Cells;背景说明:肝内胆管癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Be-Wo Cells、CF-1 MEF Cells、M-1 myeloid leukemia Cells
SK-MEL-24 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:Capan 2 Cells、639 V Cells、NCI-HUT-292 Cells
MLA144 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:A875 Cells、HT-144 Cells、PANC0203 Cells
BayGenomics ES cell line RRJ104 Cells(拥有STR基因鉴定图谱)
NCI-H1975人肺腺癌细胞种子库|送STR图谱
BayGenomics ES cell line XK089 Cells(拥有STR基因鉴定图谱)
FE1 Cells(拥有STR基因鉴定图谱)
NIH 3T3/DC-SIGN Cells(拥有STR基因鉴定图谱)
W2 [Mouse ESC] Cells(拥有STR基因鉴定图谱)
MCF-7R Cells(拥有STR基因鉴定图谱)
"
风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验该产品被引用文献
"PubMed=19727395; DOI=10.1371/journal.pone.0006888; PMCID=PMC2731225
Wadlow R.C., Wittner B.S., Finley S.A., Bergquist H., Upadhyay R., Finn S.P., Loda M., Mahmood U., Ramaswamy S.
Systems-level modeling of cancer-fibroblast interaction.
PLoS ONE 4:E6888-E6888(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20557307; DOI=10.1111/j.1349-7006.2010.01622.x; PMCID=PMC11158680
Iwakawa R., Kohno T., Enari M., Kiyono T., Yokota J.
Prevalence of human papillomavirus 16/18/33 infection and p53 mutation in lung adenocarcinoma.
Cancer Sci. 101:1891-1896(2010)
PubMed=21498706
Onitsuka T., Uramoto H., Tanaka F.
Lack of direct association between EGFR mutations and ER beta expression in lung cancer.
Anticancer Res. 31:855-860(2011)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22961666; DOI=10.1158/2159-8290.CD-12-0112; PMCID=PMC3567922
Byers L.A., Wang J., Nilsson M.B., Fujimoto J., Saintigny P., Yordy J.S., Giri U., Peyton M., Fan Y.-H., Diao L.-X., Masrorpour F., Shen L., Liu W.-B., Duchemann B., Tumula P., Bhardwaj V., Welsh J., Weber S., Glisson B.S., Kalhor N., Wistuba I.I., Girard L., Lippman S.M., Mills G.B., Coombes K.R., Weinstein J.N., Minna J.D., Heymach J.V.
Proteomic profiling identifies dysregulated pathways in small cell lung cancer and novel therapeutic targets including PARP1.
Cancer Discov. 2:798-811(2012)
PubMed=25008024; DOI=10.1007/s10735-014-9583-2
Wang J., Wei H., Zhao B.-X., Li M., Lv W.-P., Lv L., Song B., Lv S.
The reverse effect of X-ray irradiation on acquired gefitinib resistance in non-small cell lung cancer cell line NCI-H1975 in vitro.
J. Mol. Histol. 45:641-652(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25483995; DOI=10.3892/mmr.2014.3058
Zhao B.-X., Wang J., Song B., Wei H., Lv W.-P., Tian L.-M., Li M., Lv S.
Establishment and biological characteristics of acquired gefitinib resistance in cell line NCI-H1975/gefinitib-resistant with epidermal growth factor receptor T790M mutation.
Mol. Med. Rep. 11:2767-2774(2015)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26202522; DOI=10.1021/acs.jproteome.5b00477; PMCID=PMC4761227
Kitata R.B., Dimayacyac-Esleta B.R.T., Choong W.-K., Tsai C.-F., Lin T.-D., Tsou C.-C., Weng S.-H., Chen Y.-J., Yang P.-C., Arco S.D., Nesvizhskii A.I., Sung T.-Y., Chen Y.-J.
Mining missing membrane proteins by high-pH reverse-phase stagetip fractionation and multiple reaction monitoring mass spectrometry.
J. Proteome Res. 14:3658-3669(2015)
PubMed=26554430; DOI=10.1021/acs.analchem.5b03639
Dimayacyac-Esleta B.R.T., Tsai C.-F., Kitata R.B., Lin P.-Y., Choong W.-K., Lin T.-D., Wang Y.-T., Weng S.-H., Yang P.-C., Arco S.D., Sung T.-Y., Chen Y.-J.
Rapid high-pH reverse phase stagetip for sensitive small-scale membrane proteomic profiling.
Anal. Chem. 87:12016-12023(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27835594; DOI=10.18632/oncotarget.13150; PMCID=PMC5348415
Tang Z.-H., Jiang X.-M., Guo X., Fong C.M.V., Chen X.-P., Lu J.-J.
Characterization of osimertinib (AZD9291)-resistant non-small cell lung cancer NCI-H1975/OSIR cell line.
Oncotarget 7:81598-81610(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=29681454; DOI=10.1016/j.cell.2018.03.028; PMCID=PMC5935540
McMillan E.A., Ryu M.-J., Diep C.H., Mendiratta S., Clemenceau J.R., Vaden R.M., Kim J.-H., Motoyaji T., Covington K.R., Peyton M., Huffman K., Wu X.-F., Girard L., Sung Y., Chen P.-H., Mallipeddi P.L., Lee J.Y., Hanson J., Voruganti S., Yu Y., Park S., Sudderth J., DeSevo C., Muzny D.M., Doddapaneni H., Gazdar A.F., Gibbs R.A., Hwang T.H., Heymach J.V., Wistuba I.I., Coombes K.R., Williams N.S., Wheeler D.A., MacMillan J.B., DeBerardinis R.J., Roth M.G., Posner B.A., Minna J.D., Kim H.S., White M.A.
Chemistry-first approach for nomination of personalized treatment in lung cancer.
Cell 173:864-878.e29(2018)
PubMed=30710758; DOI=10.1016/j.jprot.2019.01.018
Bottger F., Schaaij-Visser T.B.M., de Reus I., Piersma S.R., Pham T.V., Nagel R., Brakenhoff R.H., Thunnissen E., Smit E.F., Jimenez C.R.
Proteome analysis of non-small cell lung cancer cell line secretomes and patient sputum reveals biofluid biomarker candidates for cisplatin response prediction.
J. Proteomics 196:106-119(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31803961; DOI=10.1002/jcb.29564; PMCID=PMC7496084
Mulshine J.L., Ujhazy P., Antman M., Burgess C.M., Kuzmin I.A., Bunn P.A. Jr., Johnson B.E., Roth J.A., Pass H.I., Ross S.M., Aldige C.R., Wistuba I.I., Minna J.D.
From clinical specimens to human cancer preclinical models -- a journey the NCI-cell line database-25 years later.
J. Cell. Biochem. 121:3986-3999(2020)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023; PMCID=PMC7339254
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. 3rd, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)
PubMed=32351780; DOI=10.7717/peerj.8779; PMCID=PMC7183755
Wu J.-Y., Hao Z.-F., Ma C., Li P.-F., Dang L.-Y., Sun S.-S.
Comparative proteogenomics profiling of non-small and small lung carcinoma cell lines using mass spectrometry.
PeerJ 8:e8779.1-e8779.19(2020)"
Wadlow R.C., Wittner B.S., Finley S.A., Bergquist H., Upadhyay R., Finn S.P., Loda M., Mahmood U., Ramaswamy S.
Systems-level modeling of cancer-fibroblast interaction.
PLoS ONE 4:E6888-E6888(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20557307; DOI=10.1111/j.1349-7006.2010.01622.x; PMCID=PMC11158680
Iwakawa R., Kohno T., Enari M., Kiyono T., Yokota J.
Prevalence of human papillomavirus 16/18/33 infection and p53 mutation in lung adenocarcinoma.
Cancer Sci. 101:1891-1896(2010)
PubMed=21498706
Onitsuka T., Uramoto H., Tanaka F.
Lack of direct association between EGFR mutations and ER beta expression in lung cancer.
Anticancer Res. 31:855-860(2011)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22961666; DOI=10.1158/2159-8290.CD-12-0112; PMCID=PMC3567922
Byers L.A., Wang J., Nilsson M.B., Fujimoto J., Saintigny P., Yordy J.S., Giri U., Peyton M., Fan Y.-H., Diao L.-X., Masrorpour F., Shen L., Liu W.-B., Duchemann B., Tumula P., Bhardwaj V., Welsh J., Weber S., Glisson B.S., Kalhor N., Wistuba I.I., Girard L., Lippman S.M., Mills G.B., Coombes K.R., Weinstein J.N., Minna J.D., Heymach J.V.
Proteomic profiling identifies dysregulated pathways in small cell lung cancer and novel therapeutic targets including PARP1.
Cancer Discov. 2:798-811(2012)
PubMed=25008024; DOI=10.1007/s10735-014-9583-2
Wang J., Wei H., Zhao B.-X., Li M., Lv W.-P., Lv L., Song B., Lv S.
The reverse effect of X-ray irradiation on acquired gefitinib resistance in non-small cell lung cancer cell line NCI-H1975 in vitro.
J. Mol. Histol. 45:641-652(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25483995; DOI=10.3892/mmr.2014.3058
Zhao B.-X., Wang J., Song B., Wei H., Lv W.-P., Tian L.-M., Li M., Lv S.
Establishment and biological characteristics of acquired gefitinib resistance in cell line NCI-H1975/gefinitib-resistant with epidermal growth factor receptor T790M mutation.
Mol. Med. Rep. 11:2767-2774(2015)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26202522; DOI=10.1021/acs.jproteome.5b00477; PMCID=PMC4761227
Kitata R.B., Dimayacyac-Esleta B.R.T., Choong W.-K., Tsai C.-F., Lin T.-D., Tsou C.-C., Weng S.-H., Chen Y.-J., Yang P.-C., Arco S.D., Nesvizhskii A.I., Sung T.-Y., Chen Y.-J.
Mining missing membrane proteins by high-pH reverse-phase stagetip fractionation and multiple reaction monitoring mass spectrometry.
J. Proteome Res. 14:3658-3669(2015)
PubMed=26554430; DOI=10.1021/acs.analchem.5b03639
Dimayacyac-Esleta B.R.T., Tsai C.-F., Kitata R.B., Lin P.-Y., Choong W.-K., Lin T.-D., Wang Y.-T., Weng S.-H., Yang P.-C., Arco S.D., Sung T.-Y., Chen Y.-J.
Rapid high-pH reverse phase stagetip for sensitive small-scale membrane proteomic profiling.
Anal. Chem. 87:12016-12023(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27835594; DOI=10.18632/oncotarget.13150; PMCID=PMC5348415
Tang Z.-H., Jiang X.-M., Guo X., Fong C.M.V., Chen X.-P., Lu J.-J.
Characterization of osimertinib (AZD9291)-resistant non-small cell lung cancer NCI-H1975/OSIR cell line.
Oncotarget 7:81598-81610(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=29681454; DOI=10.1016/j.cell.2018.03.028; PMCID=PMC5935540
McMillan E.A., Ryu M.-J., Diep C.H., Mendiratta S., Clemenceau J.R., Vaden R.M., Kim J.-H., Motoyaji T., Covington K.R., Peyton M., Huffman K., Wu X.-F., Girard L., Sung Y., Chen P.-H., Mallipeddi P.L., Lee J.Y., Hanson J., Voruganti S., Yu Y., Park S., Sudderth J., DeSevo C., Muzny D.M., Doddapaneni H., Gazdar A.F., Gibbs R.A., Hwang T.H., Heymach J.V., Wistuba I.I., Coombes K.R., Williams N.S., Wheeler D.A., MacMillan J.B., DeBerardinis R.J., Roth M.G., Posner B.A., Minna J.D., Kim H.S., White M.A.
Chemistry-first approach for nomination of personalized treatment in lung cancer.
Cell 173:864-878.e29(2018)
PubMed=30710758; DOI=10.1016/j.jprot.2019.01.018
Bottger F., Schaaij-Visser T.B.M., de Reus I., Piersma S.R., Pham T.V., Nagel R., Brakenhoff R.H., Thunnissen E., Smit E.F., Jimenez C.R.
Proteome analysis of non-small cell lung cancer cell line secretomes and patient sputum reveals biofluid biomarker candidates for cisplatin response prediction.
J. Proteomics 196:106-119(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31803961; DOI=10.1002/jcb.29564; PMCID=PMC7496084
Mulshine J.L., Ujhazy P., Antman M., Burgess C.M., Kuzmin I.A., Bunn P.A. Jr., Johnson B.E., Roth J.A., Pass H.I., Ross S.M., Aldige C.R., Wistuba I.I., Minna J.D.
From clinical specimens to human cancer preclinical models -- a journey the NCI-cell line database-25 years later.
J. Cell. Biochem. 121:3986-3999(2020)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023; PMCID=PMC7339254
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. 3rd, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)
PubMed=32351780; DOI=10.7717/peerj.8779; PMCID=PMC7183755
Wu J.-Y., Hao Z.-F., Ma C., Li P.-F., Dang L.-Y., Sun S.-S.
Comparative proteogenomics profiling of non-small and small lung carcinoma cell lines using mass spectrometry.
PeerJ 8:e8779.1-e8779.19(2020)"
文献支持
NCI-H1975人肺腺癌细胞种子库|送STR图谱
¥850 - 2150