相关产品推荐更多 >
万千商家帮你免费找货
0 人在求购买到急需产品
- 详细信息
- 文献和实验
- 技术资料
- 英文名:
HCC-1833
- 库存:
100
- 供应商:
中乔新舟
- 品系:
细胞系
- 运输方式:
常温
- 年限:
液氮长期
- 生长状态:
贴壁生长
- 规格:
T25
|
产品名称 |
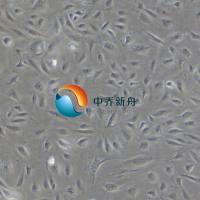
HCC-1833 人肺癌腺癌细胞 |
|
货号 |
ZQ1037 |
|
产品介绍 |
HCC-1833细胞系源自一位患有肺腺癌的患者的肿瘤组织。肺腺癌是非小细胞肺癌(NSCLC)的一种类型,是肺癌中最常见的亚型之一。HCC-1833细胞系保留了原始肿瘤的许多生物学特性,它们被广泛用于癌症研究,包括癌症生物学、药理学、毒理学和新药开发等领域。HCC-1833细胞系也可用于肿瘤生物学研究、药物筛选、分子机制研究等领域。是研究肺腺癌的发病机制、探索治疗靶点,尤其是与吸烟、环境因素及遗传易感性相关的研究提供了重要基础。 |
|
种属 |
人 |
|
性别/年龄 |
女/69岁 |
|
组织 |
肺 |
|
疾病 |
肺癌;腺癌癌 |
|
细胞类型 |
肿瘤细胞 |
|
形态学 |
上皮 |
|
生长方式 |
贴壁 |
|
倍增时间 |
46.8 hours (PubMed=29681454) |
|
培养基和添加剂 |
RPMI-1640(品牌:中乔新舟 货号:ZQ-200)+10%胎牛血清(中乔新舟 货号:ZQ500-A)+1%P/S(中乔新舟 货号:CSP006) |
|
推荐完全培养基货号 |
ZM1037 |
|
生物安全等级 |
BSL-1 |
|
培养条件 |
95%空气,5%二氧化碳;37℃ |
|
STR位点信息 |
Amelogenin: X CSF1PO :12 D2S1338 :19,23 D3S1358: 16 D5S818 :9 D7S820 :11,12 D8S1179: 13,15 D13S317 :8,11 D16S539 :9,11 (PubMed=25877200) :11 (PubMed=20679594) D18S51 :17 D19S433 :13,14 D21S11: 28 FGA :20 Penta D: 7 Penta E: 12 TH01: 8,9.3 (KCLB=71833; PubMed=20679594) :9 .3 (PubMed=25877200) TPOX: 8 vWA: 19 |
|
抗原表达/受体表达 |
*** |
|
基因表达 |
*** |
|
保藏机构 |
KCLB; 71833 |
|
供应限制 |
仅供科研使用 |
上海中乔新舟生物科技有限公司成立于2011年,历经十多年发展,主要专注于细胞生物学产品的研究和开发,专注于为药企、各类科研机构及CRO企业提供符合标准规范的细胞培养服务、细胞培养基、细胞检测试剂盒、细胞培养试剂,胎牛血清和细胞生物学技术服务等。
公司一直致力于为高等院校、研究机构、医院、CRO及CDMO企业提供细胞培养完整解决方案,这些产品旨在满足细胞培养的多样需求,确保实验和研究的有效进行。引用中乔新舟(ZQXZBIO)产品和服务的文献超数千篇。

产品服务
细胞资源:原代细胞、细胞株、干细胞、示踪细胞、耐药株细胞、永生化细胞等基因工程细胞。
试剂产品:胎牛血清、完全培养基(适用于原代细胞及细胞株)、无血清培养基、基础培养基、细胞转染试剂、重组因子、胰酶和双抗等等细胞培养所有实验相关产品。
技术服务:稳转株构建、原代细胞分离、特殊培养基定制服务、细胞检测等。

目前产品已经畅销国内30多个省市,与客户建立长期的合作伙伴关系,共同实现成功。全体员工将不懈努力,继续为科研人员提供优良的产品和服务,致力成为全球细胞培养领域的参与者。
企业愿景
致力于成为国内细胞培养基产业的佼佼者,生物医药领域上游原材料的优良提供商。
企业使命
成长为专业细胞系及原代细胞培养供应商、专业细胞培养基及培养试剂生产商。
企业荣誉


风险提示:丁香通仅作为第三方平台,为商家信息发布提供平台空间。用户咨询产品时请注意保护个人信息及财产安全,合理判断,谨慎选购商品,商家和用户对交易行为负责。对于医疗器械类产品,请先查证核实企业经营资质和医疗器械产品注册证情况。
 文献和实验
文献和实验
PubMed=10353731
Wistuba I.I., Bryant D., Behrens C., Milchgrub S., Virmani A.K., Ashfaq R., Minna J.D., Gazdar A.F.
Comparison of features of human lung cancer cell lines and their corresponding tumors.
Clin. Cancer Res. 5:991-1000(1999)
PubMed=16187286; DOI=10.1002/ijc.21491
Garnis C., Lockwood W.W., Vucic E., Ge Y., Girard L., Minna J.D., Gazdar A.F., Lam S., MacAulay C., Lam W.L.
High resolution analysis of non-small cell lung cancer cell lines by whole genome tiling path array CGH.
Int. J. Cancer 118:1556-1564(2006)
PubMed=20679594; DOI=10.1093/jnci/djq279
Gazdar A.F., Girard L., Lockwood W.W., Lam W.L., Minna J.D.
Lung cancer cell lines as tools for biomedical discovery and research.
J. Natl. Cancer Inst. 102:1310-1321(2010)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26361996; DOI=10.1016/j.jprot.2015.09.003
Grundner-Culemann K., Dybowski J.N., Klammer M., Tebbe A., Schaab C., Daub H.
Comparative proteome analysis across non-small cell lung cancer cell lines.
J. Proteomics 130:1-10(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29681454; DOI=10.1016/j.cell.2018.03.028
McMillan E.A., Ryu M.-J., Diep C.H., Mendiratta S., Clemenceau J.R., Vaden R.M., Kim J.-H., Motoyaji T., Covington K.R., Peyton M., Huffman K., Wu X.-F., Girard L., Sung Y., Chen P.-H., Mallipeddi P.L., Lee J.Y., Hanson J., Voruganti S., Yu Y., Park S., Sudderth J., DeSevo C., Muzny D.M., Doddapaneni H., Gazdar A.F., Gibbs R.A., Hwang T.H., Heymach J.V., Wistuba I.I., Coombes K.R., Williams N.S., Wheeler D.A., MacMillan J.B., DeBerardinis R.J., Roth M.G., Posner B.A., Minna J.D., Kim H.S., White M.A.
Chemistry-first approach for nomination of personalized treatment in lung cancer.
Cell 173:864-878.e29(2018)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. III, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31803961; DOI=10.1002/jcb.29564
Mulshine J.L., Ujhazy P., Antman M., Burgess C.M., Kuzmin I.A., Bunn P.A. Jr., Johnson B.E., Roth J.A., Pass H.I., Ross S.M., Aldige C.R., Wistuba I.I., Minna J.D.
From clinical specimens to human cancer preclinical models -- a journey the NCI-cell line database-25 years later.
J. Cell. Biochem. 121:3986-3999(2020)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. III, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)
littleyan0418 我现在的任务是要构建人肺癌细胞的cDNA文库,但我不是这个方面科班出身,好多地方还不是很明白,想请教师兄师姐的经验,应该注意些什么。现在对总RNA的提取我有点找不出头绪。麻烦师兄师姐们可以给我分享些这方面的资料和网址吗?非常感谢!! neuronboy http://www.takara.com.cn/ 价格、试剂盒和说明书都有。呵呵 littleyan0418
Direct DNA Injection (p53) into HCC Tumors
Liver tumors, specially HCC, are among the most common malignancies in the world, and their annual world incidence is about 250,000 cases, with a male to female ratio 4:1 (1 ). HCC is one of the most important neoplasms in tropical
涂片中腺癌(adenocarcinoma)分为分化较好的和分化差两种亚型。 1.分化较好的腺癌细胞:癌细胞体积较大,呈圆形、卵圆形、成排脱落者可呈不规则的柱状。胞质丰富略嗜碱染色,。胞质内可见粘液空泡,呈透明的空泡状,有的空泡很大,核被挤压在一边呈半月状,称为印戒细胞。医学教育|网搜集整理有时在胞质内见到圆形细胞围腺样结构。癌细胞核为圆形或卵圆形、略畸形,染色质丰富,略深染,呈粗块或粗网状,核边不规则增进取,常见1-2个增大核仁直径是可达3-5μm.胞质核常偏于癌细胞的一侧
 技术资料
技术资料暂无技术资料 索取技术资料