北京基尔比生物科技有限公司
3 年
手机商铺
- NaN
- 0
- 1
- 0
- 3
推荐产品
技术资料/正文
期刊编辑如何看这篇无脑畸形(Lissencephaly)的类器官研究及其药物干预?
95 人阅读发布时间:2025-11-03 15:25
北京基尔比生物研制生产——3D细胞类器官孔板培养重力摇床
 中国医学科学院药物研究所-北京基尔比生物Kilby Bio 3D细胞类器官摆动式精密灌注仪(重力摇床)安装现场
中国医学科学院药物研究所-北京基尔比生物Kilby Bio 3D细胞类器官摆动式精密灌注仪(重力摇床)安装现场
北京基尔比生物研发的Kilby STPI型摆动式灌注仪(重力摇床),是一款专为 3D 细胞培养及类器官高通量生物科研实验设计的精密设备。其核心优势在于通过实现持续灌流和低剪切力的可控流体环境,解决传统培养中营养交换不均、类器官核心坏死等关键痛点,完美适配在分子医学研究、精准医疗转化及高通量筛选中的核心需求,为生物样本功能研究、药物靶点验证等场景提供稳定可靠的培养条件支撑。
关键技术参数:
可支持堆叠两层不少于4块96孔板同时灌注培养,也可定制适配器官芯片等
兼容实验室二氧化碳培养箱内长期(连续)使用
北 京 基 尔 比 生物科技公司主营产品:
Kilby 多通道旋转细胞培养系统(RCCS),
Kilby Gravity 微超重力三维细胞培养系统,
动植物/微生物等地面重力环境模拟装置【可以定制】,
Kilby Bio 3D细胞类器官摆动式精密灌注仪(重力摇床),
Kirkstall Quasi Vivo 类器官3D串联芯片灌注培养系统
LIS1无脑畸形:一种神经发育障碍,特征为大脑皮层折叠减少和严重的神经功能障碍。所有病例都由LIS1基因的杂合突变引起,但患者的症状严重程度存在广泛差异。
LIS1基因:编码一种同源二聚体蛋白,是dynein-dynactin复合体组装和微管马达功能的关键调节因子。LIS1通过增强dynein马达的进程性,支持对多种神经发育过程至关重要的逆向运输,包括神经元迁移、轴突生长等

Nature Communications | (2025) 16:9091核心内容是关于LIS1基因突变导致的无脑畸形(Lissencephaly)的研究,特别是探讨了不同严重程度的LIS1无脑畸形在患者来源的前脑类器官中蛋白质稳态失调的机制。研究通过整合形态学、转录组学和蛋白质组学分析,揭示了LIS1无脑畸形的疾病严重程度与神经祖细胞稳态和神经发生的渐进性变化相关,并提出了可能的治疗靶点。
(一)从期刊编辑和审稿人的角度来看,这篇论文被录用的原因主要集中在以下几个方面:
1. 研究的创新性和科学价值
- 创新性:这篇论文通过患者来源的前脑类器官模型,系统地研究了LIS1无脑畸形的疾病严重程度与蛋白质稳态失调之间的关系。这种多组学整合分析方法(包括形态学、转录组学和蛋白质组学)在LIS1无脑畸形研究中是较为新颖的,为理解该疾病提供了新的视角。
- 科学价值:研究揭示了LIS1蛋白水平与临床严重程度之间的相关性,并发现了微管不稳定、WNT信号通路受损以及蛋白质稳态失调等新的分子机制。这些发现不仅增进了我们对LIS1无脑畸形病理机制的理解,还为未来的治疗策略提供了潜在的靶点。
2. 研究方法的严谨性
- 样本选择:研究者精心选择了涵盖轻度到重度的LIS1无脑畸形患者样本,确保了研究结果的广泛适用性。这种基于疾病严重程度的分层设计,能够更好地捕捉不同突变类型对疾病表型的影响。
- 实验设计:实验设计合理,包括细胞重编程、前脑类器官的生成、多组学分析以及药物干预实验等。每一步骤都遵循了严格的科学标准,并且通过多种方法对结果进行了验证,确保了研究的可靠性和重复性。
- 数据分析:论文中详细描述了数据分析的方法,包括单细胞RNA测序和蛋白质组学分析。使用了先进的分析工具和统计方法,如SCTransform、DoubletFinder、WGCNA等,确保了数据分析的准确性和深度。
3. 研究结果的影响力
- 临床相关性:研究结果直接关联到患者的临床表现,揭示了LIS1无脑畸形的分子基础,这对于临床诊断和治疗具有重要意义。特别是发现的潜在治疗靶点(如mTORC1抑制剂),为未来的临床试验提供了科学依据。
- 学术贡献:论文不仅提供了新的病理机制,还展示了患者来源的类器官模型在研究神经发育障碍中的强大潜力。这种模型可以用于研究其他神经发育疾病,具有广泛的学术价值。
4. 论文的呈现质量
- 写作质量:论文的写作清晰、逻辑严谨,实验方法和结果的描述详细,使得其他研究者能够理解和重复实验。图表和数据的呈现也符合学术标准,有助于读者快速理解研究的核心内容。
- 引用和讨论:论文在讨论部分详细讨论了研究结果的意义,并与现有文献进行了比较和对比。这表明作者对相关领域的研究有深入的了解,并能够将他们的发现放在更广泛的学术背景下进行讨论。
5. 研究的伦理和合规性
- 伦理审查:研究涉及人类样本的使用,所有参与者均提供了书面知情同意,且研究得到了相关伦理委员会的批准。这表明研究符合伦理标准,保护了参与者的权益。
- 数据共享:论文提供了详细的实验方法和数据分析代码,并将RNA测序数据和蛋白质组学数据存储在公共数据库中。这种开放科学的做法有助于其他研究者验证和扩展研究结果,符合学术界的数据共享原则。
6. 研究的潜在应用前景
- 治疗靶点:研究中发现的mTORC1抑制剂等潜在治疗靶点,为未来的药物开发和临床试验提供了新的方向。这不仅对LIS1无脑畸形患者有潜在的治疗意义,也可能对其他神经发育障碍的治疗提供借鉴。
- 模型应用:患者来源的前脑类器官模型的成功应用,为研究其他神经发育疾病提供了一个强大的工具。这种模型可以用于药物筛选、疾病机制研究等多个领域,具有广泛的应用前景。
结论
综合来看,这篇论文因其创新性、科学价值、严谨的研究方法、重要的临床相关性、高质量的呈现以及潜在的应用前景,被期刊编辑和审稿人认为是值得发表的。这些因素共同确保了论文能够为学术界和临床实践做出重要贡献。
(二)原文中患者来源的前脑类器官研究过程的详细描述
1. 患者样本选择与细胞重编程
样本来源:研究者从63例LIS1无脑畸形患者中筛选出7例,涵盖从轻度(Dobyns分级5)到重度(Dobyns分级1)的全部皮质发育异常范围。每例患者均携带经分子鉴定的LIS1致病变异。
细胞重编程:将患者来源的体细胞(如皮肤成纤维细胞或淋巴细胞)通过非整合型方法重编程为诱导多能干细胞(iPSC)。具体使用CTSTM CytoTuneTM-iPS 2.1 Sendai重编程试剂盒,严格遵循制造商的说明进行操作,将OCT4、SOX2、KLF4和c-MYC这4个转录因子导入体细胞,使其重编程为iPSC。
2. 前脑类器官的生成
iPSC的准备与培养:将重编程后的iPSC在Geltrex包被的细胞培养板上,使用Pluripromedium(PP)或Essential 8(E8)培养基进行培养,每天更换培养基。传代时,使用TrypLE Express或EDTA处理细胞,并在传代后的培养基中补充5μM Y-27632以促进细胞存活。
类器官诱导与培养:将iPSC细胞团聚后,置于含有特定诱导因子的培养基中,诱导其向神经外胚层分化,进而形成前脑类器官。在诱导过程中,培养基会根据不同的发育阶段进行调整,以模拟人类胚胎大脑发育的环境。
质量控制:在培养过程中,定期对类器官进行形态学观察和免疫组化分析,以确保其正常发育并表现出预期的神经外胚层特征。例如,通过检测神经外胚层标志物(如SOX2)的表达来评估类器官的分化程度。
3. 类器官的形态学分析
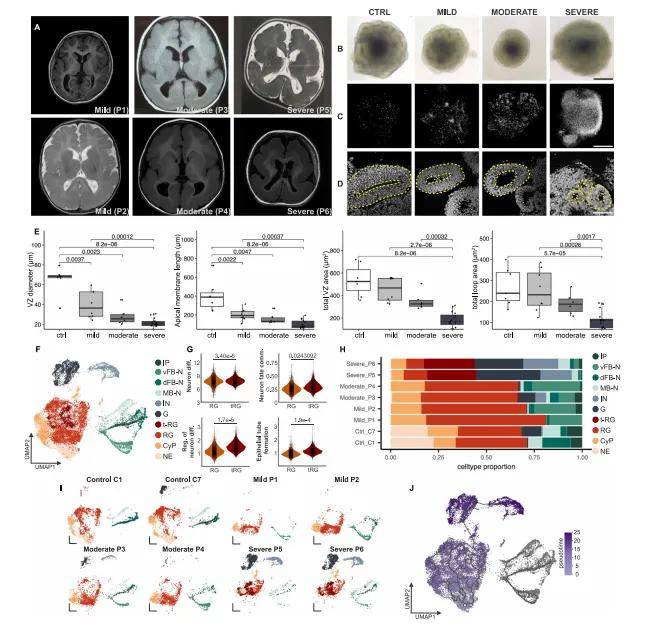
早期形态学观察:在培养的第20天左右,对类器官进行亮场显微镜观察,记录其整体形态特征。轻度LIS1患者来源的类器官形成规则的神经上皮环状结构,中度类器官通常较小,重度类器官则表现出不规则边缘,有细胞向嵌入基质中突出。
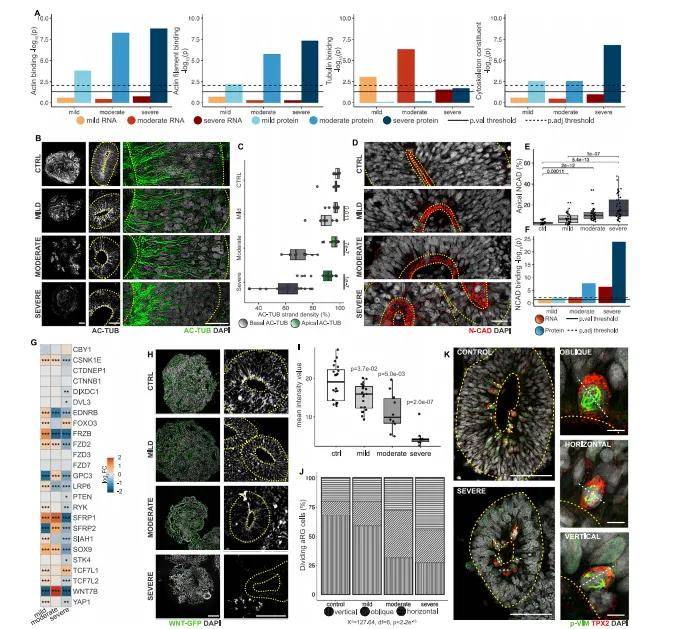
免疫组化染色:对类器官进行免疫组化染色,使用特异性抗体标记不同的细胞类型和结构。例如,使用β-III微管蛋白(TUBB3)抗体标记神经元,通过共聚焦显微镜观察神经元的分布和组织情况。结果显示,重度类器官中存在明显的神经元带,而轻度和中度类器官中该带不明显,对照组中几乎不存在。
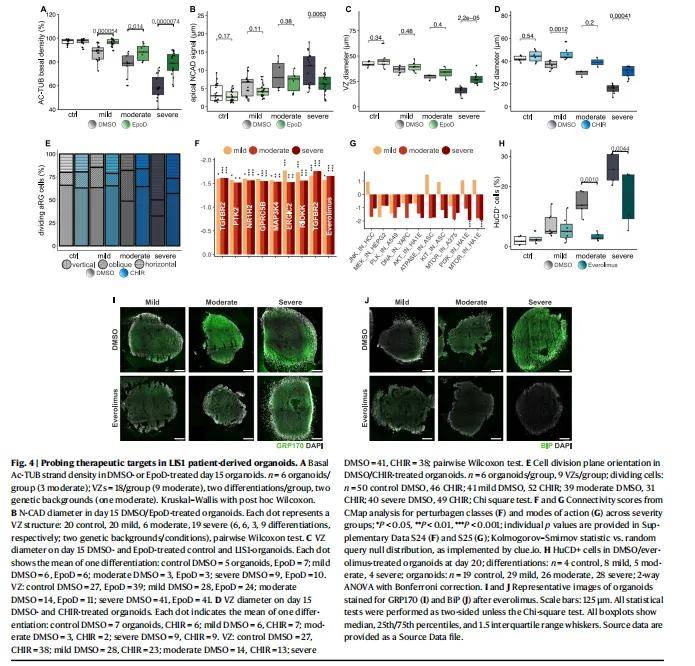
结构分析与量化:对类器官的脑室带(VZ)结构进行苏木精-伊红(HE)染色,并使用图像分析软件对VZ的大小、组织结构等进行量化分析。测量的参数包括顶膜和基底膜的长度、VZ的直径和大小、总环面积和大小等。结果表明,随着疾病严重程度的增加,VZ结构的大小和组织有序性逐渐下降,重度类器官与对照组在所有六个参数上均存在显著差异,中度类器官在四个参数上存在差异,轻度类器官在两个参数上存在差异。
4. 单细胞RNA测序(scRNA-seq)
样本收集与处理:在培养的第23天左右,收集对照组和不同严重程度的LIS1患者来源的类器官,进行单细胞RNA测序。将类器官切片后,使用已发表的单细胞制备协议进行细胞分离,或使用10x Genomics Chromium核分离试剂盒进行核分离。
文库制备与测序:使用10x Genomics Chromium平台进行单细胞或核文库制备,按照10x Genomics Chromium Single Cell 3' Library & Gel Bead Kit v3.1化学试剂盒的用户指南进行操作。制备好的cDNA文库由德国癌症研究中心(DKFZ)的高通量测序单位进行处理,并在Illumina NovaSeq 6k平台上进行测序。
数据分析:使用Cell Ranger软件从FASTQ文件生成单核RNA-seq数据集的计数矩阵,并使用Seurat v4.0.5进行数据分析。首先对特征进行过滤,去除在任何细胞中均未表达的特征,并根据检测到的特征数量、总UMI计数和线粒体基因比例(<10%)对细胞进行单独过滤。然后使用SCTransform进行归一化,并使用DoubletFinder v2.0.3排除细胞多重性。通过主成分分析(PCA)进行降维,并基于前50个主成分构建邻域图,使用Leiden算法进行聚类,最终得到10个细胞簇。通过已知标记基因对簇进行注释,并手动将中间祖细胞(IP)簇根据EOMES表达(>0.25)进一步细分为两个亚簇。
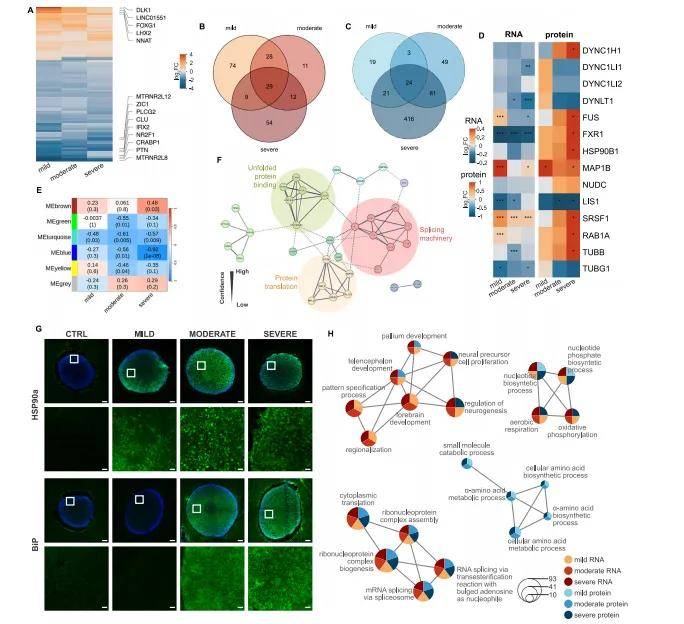
5. 蛋白质组学分析
样本收集与蛋白提取:从对照组、轻度、中度和重度LIS1患者来源的iPSC中生成均质化的皮质祖细胞培养物,使用TriFast试剂盒按照制造商的协议进行蛋白提取。将干燥的、快速冷冻的蛋白沉淀物溶解在含有50 mM Tris-HCl缓冲液(pH 7.8)、5% SDS和cOmplete ULTRA蛋白酶抑制剂(Roche)的溶液中,然后使用Bioruptor®进行处理,以确保蛋白充分溶解。
蛋白消化与标记:使用S-trap协议对蛋白进行消化,将蛋白与胰蛋白酶的比例保持在20:1,并在45°C下消化2小时。通过添加甲酸使消化液酸化,以终止消化过程。然后使用PepSwift单体柱在Ultimate 3000 HPLC上进行脱盐处理,并通过直接注射1μg样品进行HPLC分离,以确保蛋白完全消化。
质谱分析:将1μg的肽样品在Ultimate 3000快速分离液相色谱(RSLC)nano系统上进行分离,并与Q Exactive HF Orbitrap质谱仪(Thermo Scientific)联用。使用Acclaim C18 PepMap100捕获柱和反相色谱柱进行分离,并设置相应的梯度洗脱条件。质谱仪在数据依赖采集模式(DDA)下运行,进行全MS扫描和离子选择碎裂,以获取肽段的质谱图。
数据分析:使用Proteome Discoverer软件对质谱原始数据进行处理,并在人类Uniprot数据库中进行目标/诱饵模式搜索。设置相应的搜索参数,包括酶、修饰、离子容忍度等。使用Percolator进行假发现率(FDR)控制,并对蛋白进行定量分析。通过Wilcoxon秩和检验比较样本间的蛋白表达差异,并使用加权相关网络分析(WGCNA)构建蛋白共调控网络,以识别与疾病严重程度相关的蛋白模块。
6. 药物干预实验
药物处理:在类器官培养的第10天至第15天,对类器官进行药物处理。使用1μM的CHIR99021(GSK3β抑制剂,激活WNT信号通路)或1nM的Epothilone D(微管稳定剂)进行处理,并在处理后对类器官进行固定和冰冻切片,以便后续的免疫组化分析。
效果评估:通过免疫组化染色观察药物处理后类器官的形态学变化,如VZ结构的恢复、神经元带的减少等。结果表明,EpoD处理部分恢复了重度类器官中的乙酰化微管蛋白(Ac-TUB)纤维密度、顶膜N-cadherin定位和VZ形态,并减少了神经元带的大小,表明减轻了过早神经发生。CHIR99021处理也改善了VZ结构并减少了神经元带,且在重度类器官中效果最为显著。这些结果进一步支持了LIS1相关缺陷的特异性,并强调了治疗时机和剂量的重要性。
7. 晚期类器官的转录组分析
样本收集与处理:在培养的第60天左右,收集对照组和重度LIS1患者来源的类器官,进行单细胞RNA测序。同样使用10x Genomics Chromium平台进行文库制备和测序。
数据分析:使用Cell Ranger multi软件生成计数矩阵,并使用Seurat v5.0.2进行数据分析。过滤掉检测到基因数量较少或线粒体含量较高的细胞后,应用SCTransform归一化。